차세대 시퀀싱 기술의 합성 생물학 적용 연구동향

연세대학교 방두희 교수
1. 차세대 시퀀싱 기술의 개발 및 발전
지구상에 존재하는 모든 생명체는 본인의 유전정보를 염기서열에 암호화하여 저장하고 있다. 따라서 생명체의 게놈 염기서열을 해독하는 것으로 생명체의 구성요소 및 여러 생화학적 기작을 이해할 수 있고 이를 바탕으로 의학적이나 산업적으로 중요한 고부가가치 물질을 생산하는 인공생명체 합성을 게놈염기서열 조작을 통해서 유도해낼 수 있다. 이와 같은 파급효과를 가지고 있는 게놈 염기서열 해독 기술인 시퀀싱(sequencing)기술은 1970년 Frederick Sanger에 의해 처음 개발되었다1. Sanger에 의해 개발된 시퀀싱 기술은 해독하고자 하는 게놈 염기서열을 template으로 하여 2’,3’-dideoxynucleotides형태의 A, T, G, C를 순차적으로 반응함으로써 chain-terminating inhibitors기법으로 생명체의 염기서열을 해독하였고 이후 Dye-terminator를 연결하여 자동화 플랫폼을 통한 시퀀싱 기술 산업화가 진행되었다2
하지만 sanger sequencing에 기반한 시퀀싱 기술은 한 개의 tube에서 한 종류의 DNA 가닥 염기서열을 해독하는 기법으로 한번에 해독할 수 있는 DNA 종류 수의 throughput에 제한이 있었고 이에 따라 생명체의 게놈 염기서열을 해독하는 것은 사실상 불가능해 보였다.
따라서 2000년도 휴먼게놈을 해독하고자 하는 프로젝트가 (Human Genome project) 진행되었을 때 기존의 sanger sequencing 기술보다 더 빠르고, 더 많은 DNA 염기서열 정보를 해독할 수 있는 차세대 시퀀싱 기술 개발에 대한 노력이 시작되게 되었다. 이와 같은 노력의 결과로 차세대 시퀀싱 기술 (Next generation sequencing)이 개발되었고 수백만 개의 DNA 염기 서열 정보를 병렬적으로 처리할 수 있게 되었다. 차세대 시퀀싱 기술의 개발로 시퀀싱 기술은 혁명을 이루었고 보다 저렴한 비용으로 대량의 염기서열을 해독할 수 있게 되었으며 더 이상 생명체의 게놈염기서열을 해독하는 것에 그치지 않고 이를 통한 개인 맞춤형 의약품 개발, 미생물을 통한 고부가가치 무질 생산 인공생명체 합성 등에 응용되기 시작하였다.
현재의 차세대 시퀀싱 기술은 여러 개의 플랫폼으로 상업화되었으며 대표적으로 시퀀싱 기술을 제공하는 회사는 Illumina, Life technology, Roche, Pacific Biosciences들이 있다(Figure 1. 참고). 각각의 회사에서 제공하는 시퀀싱 기술은 플랫폼에 따라 시퀀서(sequencer) 사양이 달라 연구자는 해독하고자 하는 목적에 맞는 시퀀서를 선택하여 사용하고 있다.본 보고서에서는 대표적인 예들을 중심으로 차세대 시퀀서 기술의 합성 생물학 적용과 앞으로의 차세대 시퀀싱 기술 개발 연구동향에 대해 고찰해 보고자 한다.
2. 차세대 시퀀싱 기술의 합성 생물학 적용 사례
2.1 단백질 엔지니어링 연구의 차세대 시퀀싱 기술 적용
단백질 엔지니어링은 단백질 서열에 변화를 주어 genotype 변화를 통해 단백질의 phenotype을 변화시키고 연구자가 원하는 성질을 갖는 단백질을 선별하는 과정으로 이루어 진다. 이를 좀 더 실험적 측면에서 자세히 설명하면, 단백질 염기서열 조합을 변형시키는 방식으로 디자인 하고 디자인된 다양한 단백질 염기서열 조합의 DNA 라이브러리를 합성하여 최종적으로 단백질을 발현시키는 것으로 genotype 변이를 통한 phenotype변화를 관찰하게 된다. 이때 단백질 phenotype을 관찰 하는 기법으로 protein display 기술을 이용하게 되면 106~1012종류의 다른 genotype을 가지는 단백질 variant들을 한번에 병렬적으로 관찰 할 수 있게 된다. 하지만 최종적으로 단백질의 다양한 genotype과 phenotype의 상관 관계를 이해하려면 합성된 다양한 염기서열 조합의 DNA 라이브러리 염기서열 정보를 확인하여야 하는데 이 과정이 노동집약적이고 비용이 많이 소요되기 때문에 분석 할 수 있는 DNA 염기서열 조합 수에 제한이 있었다. 기본적으로 다양한 조합의 DNA 라이브러리 염기서열 정보를 sanger sequencing을 통한 분석은 DNA 라이브러리를 개별적으로 분리하기 위해 clonal selection 방법을 이용하고 이는 cloning-individualization-preparation-sequencing 순서로 진행된다.
따라서 이러한 한계를 해결하고자 2010년 미국 워싱턴 주립대학교 David baker교수 연구진은 차세대 시퀀싱 기술을 이용한 단백질 엔지니어링 연구를 진행하였다3.(Fowler et al. 2010) 이 연구진은 사람의 몸 속에서 function을 가지는 가장 작은 단백질, human Yes-associated protein65, 에 대하여 아미노산 영역에 변이를 준 60만가지 이상의 variant 를 합성한 뒤 차세대 시퀀싱을 이용하여 genotype 분석을 진행하였다. 이 과정을 통해 단백질 domain전체의 영역에서 아미노산 변형을 통한 phenotype 변화의 상관관계를 나타내는 sequence-function map을 얻을 수 있었다. (그림 1참고)
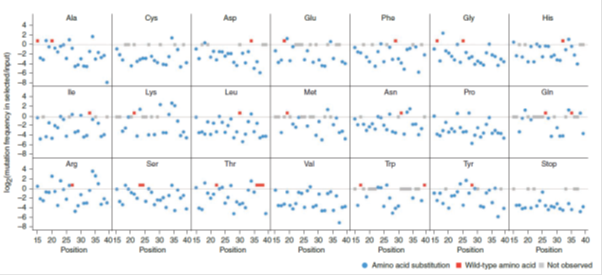
그림 1. 단백질의 genotype에 따른 phenotype 의 상관관계를 나타내는
Sequence-Function map의 예시
2.2 유전자 합성 연구의 차세대 시퀀싱 기술 적용 사례
유전자 합성은 GenBank와 같은 생명체 게놈 염기서열 데이터베이스에 저장되어있는 유전정보를 이용하여 화학적인 방법으로 염기서열을 합성하는 기술이다. 하지만 화학적으로 올리고뉴클레오티드(oligonucleotides)를 합성 할 때 뉴클레오티드 하나하나를 연결하는 base coupling 효율이 100%가 아니기 때문에 원하지 않은 형태의 합성된 올리고뉴클레오티드에는 분자들이 존재하게 된다. 합성하고자 한 유전자의 염기서열과 한 개의 염기서열이라도 다르게 되면 이는 곧 아미노산 서열을 변형시키는 요인으로 작용하게 되어 합성하고자 하는 유전자를 합성하지 못하게 된다. 따라서 한번에 합성 할 수 있는 올리고뉴클레오티드 합성 길이에는 사이즈 제한이 걸리게 되고 이 때문에 합성하고자 하는 유전자를 여러 짧은 길이의 올리고뉴클레오티드로 나누어 합성 한 뒤 이를 유전자 크기로 연결하여 합성하게 된다.
하지만 이와 같은 과정에도 올리고뉴클레오티드 자체에 포함되어 있는 오류율 이나 또는 유전자 합성 과정 중에 생성되는 오류를 확인하기 위해선 합성된 유전자 염기서열을 확인하여야 하는데 sanger sequencing을 통한 염기서열 분석은 앞서 2.1 단백질 엔지니어링 연구의 차세대 시퀀싱 기술 적용에서도 언급 하였듯이 오류를 포함하고 있지 않은 유전자를 선별하는 상당히 노동 집약적이다. 또한 이와 같은 과정은 합성하고자 하는 유전자의 크기가 크면 클수록 또한 합성하고자 하는 유전자 가지 수가 많아질수록 천문학적인 비용과 시간이 소요되어 합성 할 수 있는 유전자 수와 유전자 및 유전체의 크기에 많은 제약이 있었다.
이와 같은 Sanger sequencing 기술에 따른 유전자 합성 과정의 제약을 해결하고자 2012년 본 연구진은 DNA 마이크로어레이(Microarray) 올리고뉴클레오티드를 이용한 유전자 합성기술에 차세대 시퀀싱 기술을 적용하여 11.4kb에 해당하는 유전자 클러스터를 합성한 연구를 발표하였다4.(Kim et al. 2012) 본 연구진은 DNA 마이크로어레이 기술을 이용하여 합성된 올리고뉴클레오티드를 차세대 시퀀싱으로 분석할 수 있는 DNA 조각 사이즈까지 어셈블(assemble) 한 뒤, 이를 차세대 시퀀싱을 이용하여 후차적인 유전자 합성에 사용할 수 있는 오류를 포함하고 있지 않은 합성된 DNA조각만을 선별하였다. 그리고 최종적으로 선별된 오류 없는 DNA조각들을 이용하여 11.4kb에 해당하는 유전 자클러스터를 고효율적으로 합성하는 것에 성공하였다 (그림 2 참고).
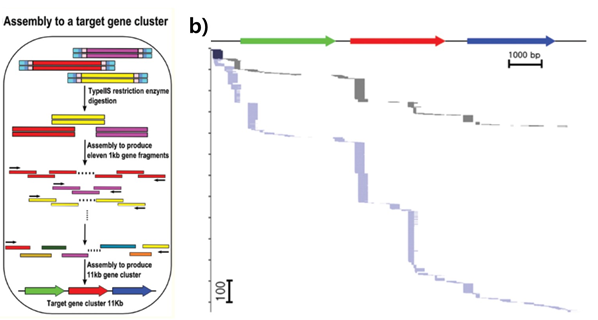
그림2. 올리고뉴클레오티드 연결을 통한 유전자 클러스터 합성 기술의 모식도와 합성된 유전자
클러스터의 차세대 시퀀싱을 통한 각 영역별 분석된 데이터 양의 그래프
3. 앞으로의 차세대 시퀀싱 기술 개발 연구 동향의 고찰
이렇듯 방대한 DNA 양의 염기서열 정보를 병렬적으로 처리할 수 있는 차세대 시퀀싱 기술을 이용한 합성 생물학 연구는 새로운 생명체의 유전정보를 확인하기 위한 유전체 염기서열 분석과 암이나 기타 질병진단을 위한 용도로 발전되어 사용되는 등 점점 그 활용도가 넓어지고 있다. 하지만, 현재 상용화 되어있는 대부분의 차세대 시퀀서들은 시퀀싱 기술의 플랫폼에 따라 분석할 수 있는 DNA 종류 수, DNA 조각의 크기, 분석 효율 등의 시퀀서 사양이 달라 연구 목적에 맞는 시퀀서를 선택하여 사용하여 한다. 이는 상업화 되어 있는 대표적 시퀀서들의 사양 및 사양에 따른 다양한 합성 생물학 연구 분야 적용을 리뷰한 논문을 통해 확인해 볼 수 있다5.(Liu et al. 2012) (그림 4,5 참조) SOLiD/Ion Torrent PGM from Life Sciences, Genome Analyzer/HiSeq 2000/MiSeq from Illumina, and GS FLX Titanium/GS Junior from Roche
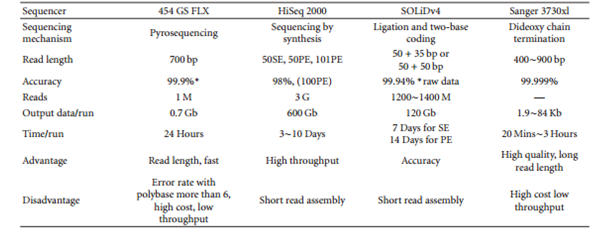
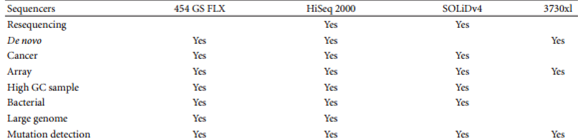
위의 표에서 확인할 수 있듯이 플랫폼마다 차세대 시퀀서들의 사양이 다르고 이를 통해 다른 합성 생물학 연구 분야 적용되어 사용되게 된다. 차세대 시퀀싱 기술의 발달로 분석할 수 있는 DNA의 양의 제한과 비용이 혁신적으로 상당 부분 해소되었지만 여전히 대부분의 차세대 시퀀서 사양 중에서 분석의 정확율은 기존의 sanger sequencing의 정확도에 못 미치고 있다.
따라서 sanger sequencing 분석 효율에 버금가면서 대량의 염기서열 데이터를 구축할 수 있는시퀀싱 기술을 개발 하는 것으로 더 많은 합성 생물학 연구 분야에 활발히 적용될 수 있을 것이며 더 나아가 차세대 시퀀싱 기술을 적용한 합성 생물학 연구가 의학분야 산업분야에 미칠 수 있는 파급효과는 매우 클 것으로 예상한다.
참고문헌
1. Sanger F at el., A rapid method for determining sequences in DNA by primed synthesis with
DNA polymerase. J Mol Biol. 1975 May 25;94(3):441-8.
2. Smith, Lloyd M., et al., Fluorescence Detection in Automated DNA Sequence Analysis. Nature
1986 Jun 12-18;321(6071):674-9.
3. Flowler DM at el., High-resolution mapping of protein sequence-function relationships. Nat
Methods 2010 Sep;7(9):741-6
4. Kim at el., 'Shotgun DNA synthesis' for the high-throughput construction of large DNA
molecules. Nucleic Acids Res. 2012 Oct;40(18);e140
5. Liu L at el., Comparison of next-generation sequencing systems. J Biomed Biotechnol.
2012;2012:251364





