효모 합성생물학의 최신 연구 동향
중앙대학교 생명과학과 김현아, 유수진, 강현아
1. 개요
수천 년 동안 인류의 일상생활에 밀접하게 이용되어온 효모는 가장 오랜 기간 동안 인류에게 길들어진 생물체로서 전통적인 발효산물 생산을 위한 숙주에서 생명공학 기술의 도입으로 현재에는 다양한 식품 및 의약품 소재 생산을 위한 숙주로도 많이 활용되고 있다. 특히, 인체에 대한 안전성이 보장된 GRAS (Generally Recognized As Safe) 미생물인 효모를 이용한 다양한 생명공학 산업제품들은 소비자들에게 거부감 없이 일상생활에 유용하게 적용될 수 있는 장점이 있어, 효모 대상의 대사공학적 연구 및 환경 친화적 청정 생물공정 개발 연구는 지속적인 관심을 받아 왔다. 효모 대상의 산업용 균주 개발 연구는 진핵생물로는 최초로 1995년 유전체 염기서열이 해독된 전통효모 Saccharomyces cerevisiae를 중심으로 다양한 'omics' 기술을 활용하여 세포의 신호전달체계와 대사회로에 대한 총체적인 정보를 확보하고, 이러한 정보를 조합하여 재래 분자유전학 방법으로는 해결할 수 없는 부분을 보완하여 새로운 기능이 부여된 고기능 효모 세포공장(cell factory)을 제작하려는 합성생물학적 접근으로 전환되고 있다(그림 1).
합성 생물학은 기존 유전 공학의 의미를 개별적인 유전자 혹은 경로에 대한 접근에서 유전자와 유전자 산물의 전체 시스템에 초점을 맞추는 것으로 확장시켜 생명체를 전체적 수준에서 분석하는 시스템 생물학 연구와 매우 밀접하게 연계되어 있다. 합성 생물학자들은 모듈화, 표준화와 같은 공학적 접근을 통해 생물 시스템의 주요 개념을 분석하고 설계하기 위해 시스템 생물학자들에 의해 발견된 많은 통찰을 기반으로 하며 그들의 전체론적 관점을 공유한다. 최근에는 메탄올자화효모 Pichia pastoris, Hansenula polymorpha, 알칸자화 효모 Yarrowia lipolytic 등을 비롯한 산업적으로 유용한 다른 비전통 효모들에 대한 유전체 염기서열 분석 및 기능유전체학 연구에 대한 관심이 고조되고 있으며, 전통효모와의 비교유전체학적 분석연구를 통해 이들 비전통 효모들에서의 세포 네트워크 조절 기작을 규명하고 이를 산업적으로 활용하고자 하는 연구에 대한 비중이 점차로 증가되고 있다(Kellis et al., 2003, Harbison et al., 2004).
본 논고에서는 효모 합성생물학을 위한 분자적 도구 개발, 효모를 이용한 재조합 단백질 의약품과 유용 대사산물 생산 시스템 개발을 위한 합성생물학적 연구 사례에 대한 최신 동향을 중점적으로 살펴보고자 한다.
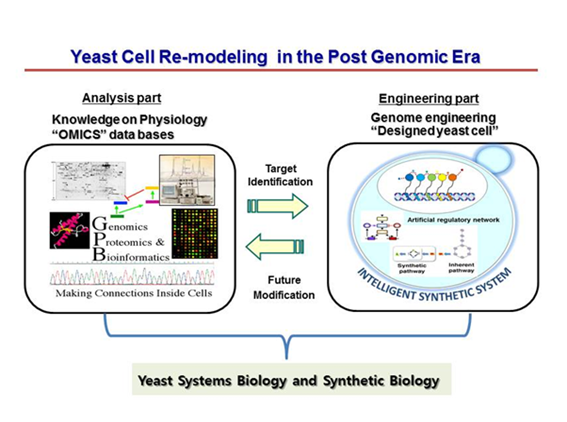
그림 1. 효모 세포 재설계를 위한 오믹스 분석 기반 시스템 및 합성 생물학 연구
2. 효모 합성생물학을 위한 분자적 도구상자(Tool boxes)
합성생물학의 특징은 나사나 볼트, 베어링 등 여러 부품을 사용하여 기계장치를 만들 듯이, 표준화된 생물학적 부품을 조합하여 새로운 생명시스템이나 생명체를 만든다는 것이다. 일반적으로 생물학자들은 세포의 구성 물질이며 정보물질인 유전자와 단백질의 특성을 조사하고 생물학적 과정을 이해하기 위해 이들을 조작하는 것과는 달리, 합성 생물학자들은 인공 생물 시스템을 구축에 필요한 생물학적 부품 및 장치를 마련하기 위해 이들 생체 물질들을 조작한다. 인공 효모 시스템을 구축하기 위한 가장 대표적인 생물학적 부품 및 장치로는 합성 프로모터, 인공 전사 인자, 또는 유전자 제거 및 삽입 카세트 등을 들 수 있다. 이와 같은 최근의 합성생물학의 발전은 고전적인 유전자 공학의 도구세트를 확장시켰다(Liang et al., 2011).
1) 합성 프로모터
효율적인 전사는 유전자 발현을 초기에 조절할 수 있는 중요한 단계로서, 강력하고 조절 가능한 프로모터는 유용대사산물 및 재조합 단백질 생산에 높은 역가를 확보하기 위한 필수적인 도구이다. 효모의 경우 특징이 잘 밝혀진 구성적 발현이나 유도 조절하는 다양한 프로모터들이 이종 유전자의 발현을 위해 사용되고 있다. 최근에는 자연적 프로모터뿐만 아니라 향상된 발현을 유도하고 맞춤형 조절 프로필을 보여주는 합성 프로모터에 대한 관심이 증가되고 있다(Blount et al., 2012).
다양한 활성과 서열을 가진 많은 세트의 구성적 프로모터들은 광범위한 생합성 경로의 제작에 매우 중요한 부품이다. 기존에는, 강한 구성적 프로모터인 TEF1과 GPD (TDH3)가 이종 유전자의 과발현을 위해 우선적으로 사용되었지만, 최근에는 다양한 전사 활성을 나타내는 구성적 프로모터들을 개발하려는 노력이 수행되고 있다(Partow et al., 2010; Fang et al., 2011). 예를 들면, 무작위로 추출된 올리고뉴클레오티드 라이브러리의 활용을 통한 합성 프로모터 라이브러리를 제작하거나 (Jeppsson et al., 2003), 또는 오류-유발 PCR(error-prone PCR)을 통해 프로모터 부위에 돌연변이를 유발시킴으로써(Alper et al., 2005), 사용 가능한 구성적 프로모터의 선택 범위를 확장시켰다. 또는 연관된 종(species)으로부터 프로모터 서열을 발굴하여 사용 가능한 효모 프로모터의 범위를 확장시키는 시도도 있다(Naesby et al., 2009).
유도 프로모터 시스템은 유도 인자의 첨가에 반응하여 발현 수준을 조절할 수 있다는 장점을 가진다. S. cerevisiae의 경우 GAL1, CUP1, 그리고 MET25 프로모터와 같은 내부적으로 존재하는 조절 네트워크나, 또는 세균에 적용되는 Tet 프로모터와 같은 외래의 합성 요소들이 활용되어 왔다. 이들 내부 프로모터 시스템은 특징이 잘 밝혀지고 예측 가능한 발현 프로파일을 실행하고 제공하기 쉽지만, 이 시스템에 대한 유도 물질들이 다면 발현적 효과를 가진다는 난점이 있다(Mumberg et al., 1994; Labbe & Thiele, 1999). 즉, 갈락토오스와 메티오닌과 같은 유도물질들은 영양소로 소모되므로, 유전자 발현 조절을 한층 더 복잡하게 만든다. 이종의 Tet 조절 프로모터 시스템의 경우는 발현 조절을 위한 부가적인 외적 기구들의 도입이 필요하다. 특히, 유도 분자들은 일반적으로 대량 생산 규모를 위해 사용하기에는 많은 비용이 들기 때문에, 유도 프로모터들은 산업용 생산 균주 개발을 위한 생합성 경로 설계에 대해서는 제한된 유용성을 가진다. 그럼에도 불구하고, 유도 프로모터 시스템은 대사 네트워크에서 특정 효소의 최적 발현 수준을 알아내는데 유용한 도구가 될 수 있다(Hawkins & Smolke, 2008).
최근 미국의 Botstein 교수가 이끄는 연구팀에 의해 전통효모 S. cerevisiae에서 보다 신속하게 유전자 발현 정도를 조절할 수 있는 인공 프로모터가 개발되었다(McIsaac et al., 2014). 6 개의 Zif268 결합 부위를 포함한 변형된 GAL1 프로모터는 Zif268 결합 도메인, 인체 에스트로겐 수용체의 리간드 결합 부위와 바이러스 단백질 V16로 구성된 인공전사 인자에 의해 베타-에스트라디올(β-estradiol) 존재 여부에 따라 발현이 매우 정교하게 조절되었다. Zif268 결합 부위의 위치가 전사개시 지역에 가까울수록, 또한 결합 부위의 카피 수가 많을수록 발현이 증가되었으며, 흥미롭게도 에스트로겐 수용체 도메인의 아미노산 치환을 통해 호르몬이 아닌 저분자 물질에 의해 발현이 조절 가능함이 관찰되었다.
메탄올자화 효모 P. pastoris의 경우, 메탄올로 유도 가능한 AOX1 프로모터(PAOX1)가 가장 많이 활용되고 있어 산업 공정에서 독성 및 인화성 문제가 있는 메탄올을 사용하지 않으면서도 강력한 발현능이 조절 가능한 합성 프로모터 변이체를 개발하는 연구가 집중적으로 수행되고 있다(Vogl & Glieder, 2013). PAOX1 전사인자 조절 결합 부위에 대한 in silico 분석에 근거하여 짧은 염기서열 부분을 삭제, 삽입, 또는 그 조합을 통해 활성을 최적화하거나(Xuan et al., 2009), PAOX1 부분단편과 자연적 핵심 프로모터 단편이 융합된 합성 PAOX1 변이체(Ruth et al., 2010) 개발을 통한 활성 증대 효과도 보고되었다. 한편 무작위 돌연변이 유발 방법에 의해 변형된 P. pastoris 글리세르알데히드-3-인산 탈수소효소 유전자(PGAP)의 구성 프로모터는 발현을 미세 조정할 수 있는 추가적인 프로모터의 가능성을 보여주었으며(Qin et al., 2011), 이러한 합성 프로모터를 이용하여 생산성 향상 효과가 관찰되었다(Mellitzer et al., 2012).
2) 유전자 제거, 치환 및 도입 장치
유전자 치환 및 표지 재활용을 위한 효율적인 전략은 많은 수의 유전자를 제거하고 도입하는 과정이 수반되는 합성생물학적 연구 수행에 절대적으로 필요하다. 그동안 전통적으로 효모를 대상으로 활용되어온 URA3 표지를 활용한 반대선택(counter-selection) 기법 외에도, Cre/loxP과 Flp/FRT 재조합효소를 이용하여 표지 유전자 절편을 제거함으로써 표지를 재활용하는 보다 진보된 장치가 개발되고 있다(Pan et al., 2011, Naatsaari et al., 2012). 이 경우, 재조합효소 발현을 갈락토오스 또는 메탄올 유도 프로모터의 엄격한 조절을 받도록 설계하면, 표지 유전자 제거 공정을 조절할 수 있는 장점이 있다.
효모 균주 제작을 위한 특정 유전자 제거 및 도입은 내인성 상동 재조합(homologous recombination, HR) 기작을 활용하여 수행된다. 전통효모 S. cerevisiae에서는 HR이 매우 효율적으로 일어나는 반면, P. pastoris를 포함한 대부분의 다른 사상 진균류 및 효모의 경우 비상동성 말단 접합(NHEJ)이 DNA 절단에 의한 손상을 수선하는 주된 경로이다. 여러 비전통 효모들을 대상으로 수행된 최근 연구들은 NHEJ에 관여하는 단백질인 Ku70 동족체를 제거함으로써 HR 비율을 보다 효율적으로 일어날 수 있음을 보고하고 있다(de Jong et al., 2010, Naatsaari et al., 2012, Verbeke et al., 2013). 일반적으로 KU70 유전자 결손 균주는 유전자 불안정성을 보이지 않았지만, 성장 저하 및 자외선 민감성이 증가되는 표현형을 보이고 있어 균주 설계를 완성한 후에 야생형 KU70 유전자를 보완해 주는 것이 바람직하다.
3) 논코딩(non-coding) RNA 조절
최근 연구들은 발현효율 조절을 위한 비전사 조절 장치 개발에 관심이 부상되고 있다. 원핵생물에서는 논코딩(non-coding) RNA가 번역 과정을 조절함으로써 유전자 발현을 활성화하거나 억제할 수 있음이 잘 알려져 있고 이를 활용하는 기술 개발도 활발하게 진행되고 있다(Isaacs et al., 2004). 합성 생물학자들은 논코딩 조절 RNA를 진핵세포에도 활용할 수 있는 기술을 개발하고 있다. 예로써, S. cerevisiae에 도입된 합성 논코딩 RNA는 특정 리간드들에 결합하는 앱타머(aptamer) 도메인과 mRNA를 타깃으로 하는 안티센스(antisense) 도메인으로 구성되어 있다. 논코딩 RNA는 리간드와 결합 시 형태가 변화되어, 안티센스 도메인이 타깃 mRNA와 결합할 수 있도록 함으로써 단백질 번역을 조절하게 되므로 안티스위치(antiswitch)로서 작동을 하게 된다(Bayer and Smolke, 2005). 이와 같은 안티스위치는, 세포내 대사물질의 농도 및 환경적인 신호를 감지하여 유전자 발현을 조절하는 새로운 합성 유전자 조절회로 제작에 매우 유용하게 사용될 것으로 기대된다.
4) 카피 수(Copy number)
효모에서 이종 유전자 발현을 조절하는 또 다른 중요한 도구세트는 원하는 발현 카세트의 카피 수를 조절할 수 있는 장치이다. 전통효모 S. cerevisiae의 경우, 2 마이크론-기반의 플라스미드는 한 세포 당 약 5-30개 카피 정도 존재하는 반면, 효모 동원체(centromere)와 연계된 복제기점 염기서열(CEN/ARS)-기반의 플라스미드는 매우 낮은 복제 수(세포 하나 당 약 1개)로 존재한다(Mumberg et al., 1995; Fang et al., 2011). 고 복제 수 플라스미드는 암호화된 유전자를 강하게 발현시켜, 세포에 큰 부담을 줄 수 있고 구조물의 불안정성을 초래할 수 있다. 저 복제 수 플라스미드는 더욱 안정한 발현 플랫폼을 제공하지만, 더 낮은 유전자 발현 수준과 결부된다. 큰 이종 발현 카세트는 인공 염색체(Yeast Artificial Chromosome, YAC)를 활용하여 도입할 수 있다(Kouprina & Larionov, 2008). 또한 상동 재조합을 통한 효모 숙주 염색체 상의 표적 유전자 부위로 발현 카세트를 삽입하는 경우 지속적인 선택압이 없는 조건에서도 발현카세트가 안정되게 유지될 수 있는 장점이 있다. 최근에는 교차와 교체가 가능한 선택 마커를 활용하여 연속적인 표적 부위로의 통합을 통해 조합적 라이브러리를 제작하는 ‘반복적 재조합’ 방법이 개발되었다(Wingler & Cornish, 2011). 또한 효모 염색체에 반복적인 카피로 존재하고 있는 리보솜 요소(Lopes et al., 1996), 델타 요소(Oliveira et al., 2007), 그리고 시그마 요소 서열(Kudla & Nicolas, 1992) 등을 표적으로 활용하여 발현 카세트가 다중 삽입되는 장치가 개발되었으며 활성이 제어된 선별표지를 함께 사용하여 다양한 카피수로 삽입되는 정도를 제어할 수 있는 기술로 발전되어왔다(Lopes et al., 1989). 이와 같은 다중 부위 삽입 및 조절 제어 장치는 효모에서의 생합성 경로 구축에 필요한 다수의 유전자 부품을 최적의 카피수로 도입시킬 수 있는 매우 유용한 도구를 제공할 것이다.
3. 재조합 단백질 의약품 생산 균주 개발을 위한 효모 합성생물학
현대의약품의 필수 품목인 생물의약품의 추정 시장가치는 700 억에서 800 억이며, 연간 성장률은 7에서 15% 사이로 예상된다(Goodman, 2009, Walsh, 2010a, Walsh, 2010b). 생물의약품은 재조합 치료용 단백질과 핵산 기반 생산물을 일컬으며, 치료용 단백질은 전형적으로 동물 세포주와 Escherichia coli에서 생산된다. 박테리아 시스템은 단순한 배지를 사용하는 생물 반응기에서 빠르고 왕성한 성장을 보여주는 반면, 동물 세포는 당화와 같은 전형적인 진핵세포의 번역 후 수식(Post-Translational Modification, PTM) 면에서 인간과 유사하다(Walsh, 2010b, Demain & Vaishnav, 2009, Palmberger et al., 2013, Berlec & Strukelj, 2013). 하지만, 동물 세포 배양 과정은 상대적으로 느리고, 복잡한 배지를 필요로 하며, 바이러스 오염에 취약하다. 효모를 이용하면 쉬운 유전자 조작과 원하는 PTM의 도입과 함께 단순한 배지에서의 빠른 성장의 조합이 가능하다는 장점이 있다(Mattanovich et al., 2012). 산업적인 관점에서 재조합 단백질 품질, 생산 소요 시간, 규모 확대 능력, 하위 분리 정제 공정 과정 등을 고려해 볼 때, 동물 세포, E. coli과 S. cerevisiae가 생물의약품 발현 시스템으로 가장 흔히 사용되며, 각각 43%, 31%, 15%의 생물의약품을 생산하고 있다(Nielsen, 2013, Berlec & Strukelj, 2013).
단세포 진핵 미생물인 효모는 박테리아와 진행생물의 특징을 모두 가지고 있어 쉬운 배양, 빠른 성장, 높은 생산성과 고밀도 발효를 할 수 있고, 적절한 단백질 접힘과 번역 후 수식 과정을 보장할 수 있으며, 생성물을 세포외 배지로 분비시킬 수 있어 정제가 간편하다(Martinez et al., 2012, Hong & Nielsen, 2012, Ilmen et al., 2011, Jonikas et al., 2009). 더욱이 일반적으로 안전하다고 알려진 GRAS(Generally Recognized As Safe) 생물이라는 점에서 효모는 생물의약품 생산 숙주로서 유리하다. 전통 효모 S. cerevisiae는 가장 연구가 잘 된 진핵생물 중 하나이고 유전자 공학과 재조합 단백질 생산의 초기 이후로 생물의약품을 위한 발현 숙주로써 사용되어 왔다(Martinez et al., 2012). 최근에는 비전통 효모 P. pastoris에서 생성된 첫 번째 생물의약품(Dyax Crop의 Kalbitor, 칼리크레인 억제제)이 FDA에 승인되었다(Walsh, 2010a). 재조합 단백질 생산을 위한 맞춤형 발현 시스템은 전사, 번역, PTM의 조작과 합성 조절 네트워크를 설계함으로써 구축된다(Lynch & Gill, 2012, Krivoruchko et al., 2011).
1) 단백질 분비 경로 재설계
진핵세포로서의 특징을 지닌 효모가 재조합 단백질을 분비 생산하기 위한 좋은 플랫폼일지라도 몇몇의 특성은 상업적 생산 요구를 충족시키기 위해 향상되어야 한다. 생물학적 관점에서 보면 DNA 암호화 서열을 성숙된 단백질로 전환시키는 다단계를 동반하는 S. cerevisiae에 의한 단백질 분비는 복잡하고, 전사, 번역, 전좌, 번역 후 수식, 접힘, 펩티드 절단, 추가적인 당화, 선별 및 분비 등의 많은 다양한 수준의 공정이 포함되어 있다(Hou et al., 2012b). 각각의 단계는 발현을 향상시키기 위한 조작 대상임을 내포한다(그림 2).
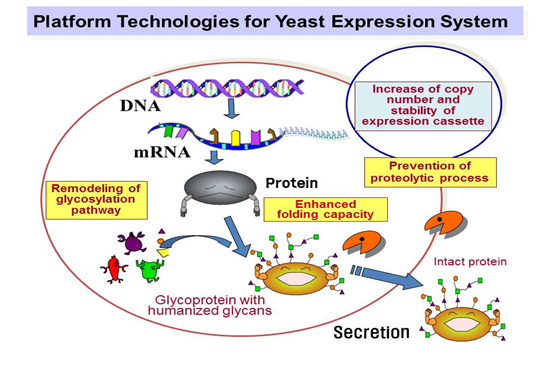
그림 2. 효모에서의 재조합 단백질 분비 생산 기술 플랫폼 개발 타겟
효모 소포체에서의 정확한 형태로의 단백질 접힘은 단백질이 분비 경로로 들어갈 것인지 또는 소포체 연관 분해(ER-ERAD) 과정으로 배정될 것인지를 결정하는 데 매우 중요한 역할을 한다(Schroder, 2008). 만약 발생기의 펩티드가 너무 빠르게 합성되면 그것이 정확하게 접히는 데에 필요한 시간이 충분하지 않아 오접힘을 초래한다(Zhang & Ignatova, 2011). 이것은 내부적 부담으로 소포체 스트레스를 야기하여 미접힘 단백질 반응(Unfolded Protein Response, UPR)을 조절한다(Payne et al., 2008, Patil & Walter, 2001). 효모의 단백질 분비능을 증가시키기 위한 몇몇의 소포체에 존재하는 단백질 폴딩 인자들의 유전자인 IL1, LHS1, EM1, KAR2, ERO1, PDI1 등의 발현을 증폭시키려는 일련의 노력들이 수행되어 왔다(Payne et al., 2008, Kim et al., 2003, Kim et al., 2007, Kim et al., 2009). 최근에는 UPR 전사조절 인자인 Hac1의 과발현을 통해 이들 소포체 단백질 폴딩인자들의 발현을 동시에 총체적으로 증폭시키려는 시도가 수행되어 왔는데, 그 효과는 타겟 단백질에 따라 차이가 있는 것으로 보고되었다(Guerfal et. al., 2010).
한편으로 충격 반응(HSR)은 환경 및 생리적 스트레스에 대응하는 질서정연한 반응이다. 단백질 접힘을 도와주는 분자 샤페론을 암호화하는 수백 개의 유전자가 일차적인 HSR 전사조절인자인 열충격 인자(Hsf1p)의 활성을 통해 증가된다(Hou et al., 2013). 지속적인 HSR 활성화를 위해 돌연변이 Hsf1 전사인자를 과발현시킴으로써 인슐린 전구체 생산을 향상시킬 수 있다(Hou et al., 2013). 다른 세포기관 사이에서의 이동을 향상시키는 것 또한 단백질 분비를 증대시킬 수 있다. 수용성 N-호중구-감수성 인자 부착 단백질 수용체(SNARE) 복합체를 조절하는 단백질인 Sec1p의 과발현은 골지체에서 세포막으로의 소낭 이동을 도와줌으로써 인슐린 분비를 향상시킨다(Hou et al., 2012a).
일부 재조합 단백질은 효모 세포표면에 위치하고 있는 단백질 분해효소에 민감하여 분비된 후 단백질이 분해되어 단백질의 최종 산물이 감소한다. 이런 경우 단백질 분해효소 결손 균주 제작이 필요한 데, 경우에 따라서는 관련된 유전자들을 다중으로 제거해야 단백질 절단 문제를 해결하게 된다. 예로써, S. cerevisiae을 이용한 인간 부갑상선 호르몬(hPTH)의 생산의 경우, 재조합 단백질의 잠재적 절단을 더욱 줄이기 위하여 세포표면에 존재하는 앱신이라는 단백질 분해효소에 대한 유전자 YPS1 뿐만 아니라 그 상동 유전자들인 YPS2, YPS3, YPS6 및 YPS7을 결손시켜 다중-앱신-결손 균주를 제작하였다. 단일 결손 균주와 비교하여 오중 결손 S. cerevisiae 균주는 유가 배양식 발효에서 hPTH의 단백질 분해를 막는 효율이 현저히 증가하였다(Cho et al., 2010).
이와 같이 최근에는 단일 표적 유전자 부품 조절에서 나아가 다중 유전자 부품 결손 및 과발현에 의한 효모세포 재설계 시도가 일반적으로 수행되고 있다. 대표적인 예로서, YPS1, PMT1과 HSP150의 결손과 함께 PDI1의 과발현이 포함된 다양한 표적들의 동시 변형은 인간 트랜스페린의 생산과 질 향상을 위한 S. cerevisiae 엔지니어링에 적용되었다(Finnis et al., 2010). Pdi1p은 단백질 접힘을 도와 정확한 단백질 생성을 향상시키고, YPS1 결손은 표적 단백질의 분해를 감소시키고, PMT1 결손은 O-당화 수식을 피하여 불활성 생성물 대신에 정확한 형태로 단백질을 만든다. Hsp150p는 분비 단백질로 재조합 단백질과 함께 동시 정제될 수 있다. HSP150의 결손은 잠재적 오염 물질을 제거할 수 있어 하위 단계 정제에 유리하다. 향상된 단백질 생산을 위한 이러한 다중 표적 전략은 유용하며 이것은 각기 다른 단계에서의 제약을 풀어주고 결합 효과가 액세스될 것이다. 효모에서의 인간 헤모글로빈 생산의 괄목할 만한 증대는 헴(heme) 생합성 경로의 대사 공학과 두 개의 글로빈 단위체 비율의 변화를 동시에 수행함으로써 헴 그룹 공급과 글로빈 생산의 최적 조합으로 확보되었다(Liu et al., 2014).
최근에 S. cerevisiae을 대상으로 분비 기구 및 반응의 핵심 구성 요소를 포함한 단백질 분비 기구에 대한 유전체-규모 모델이 상향식 접근 방법을 이용하여 제작되었다(Feizi et al., 2013). 전체 효모 단백질체(5,882 단백질)의 모든 단백질에 대한 7가지 분비 관련 특징을 추출하여 효모 단백질 특정 정보 매트릭스(y-PSIM)로 요약하였다. 1,197개의 잠재적인 ER-Golgi 분비 단백질을 산출하였고, 이들 단백질에 대한 유전체-규모 단백질 특이적 반응 목록(전체 11,684개의 반응)을 얻었다. 이 모델은 효모뿐만 아니라 다른 진핵생물에서도 단백질 분비 기구를 이해하는 데 도움이 될 것으로 기대된다. 더욱 중요한 것은, 분비 기구에서의 에너지 및 대사 수요를 추정하고, 이로써 단백질 분비를 향상시키기 위한 합성생물학적 설계 및 제작에 도움을 줄 수 있다는 것이다.
2) 합성 당생물학(Synthetic glycobiology)
의약용 단백질들의 대부분은 가장 복잡하면서도 동시에 가장 흔한 과정인 당화와 함께 번역 후 수식을 포함한다(Walsh, 2010b). 효모는 전형적인 진핵생물의 PTM을 수행할 수 있지만, 효모와 인간의 최종 당화 양식은 확연히 다르다. 특히 S. cerevisiae로부터 만들어진 당단백질과 결부되어 있는 과만노실화와 말단의 α-1,3-만노오스 연결은 혈청 반감기가 낮아지거나 심지어 치료용 단백질의 면역원성 유발을 초래할 수도 있다(Walsh, 2010b, De Pourcq et al., 2010). 이와 같은 효모 특이적 당사슬을 제거하고 인체형 당사슬을 합성해 낼 수 있는 지능형 효모 균주 개발에 대한 노력이 S. cerevisiae를 대상으로 우선적으로 수행되어 왔다. ALG3와 ALG11 이중 돌연변이 S. cerevisiae는 분비 단백질에 본래의 다량의 만노오스 수식을 수행하지 못하는 데, 이 이중 유전자 결손 변이주에 지질-결합 올리고당(LLO) 생합성 경로를 도입하여 새로운 합성 N-당화 경로를 조립하여 인간화 N-글리칸을 가진 단일클론 항체 HyHEL-10의 생산을 하였다(Parsaie Nasab et al., 2013). 최근에는 S. cerevisiae에 비해 과만노실화가 덜 일어나며, 면역유발성이 강한 말단의 α-1,3-만노오스 결합을 없는 비전통효모들을 대상으로 효모 당화 경로를 인간화시키는 시도가 활발하게 진행되고 있다(Cheon et al., 2012, De Pourcq et al., 2010, Hamilton & Gerngross, 2007, Park et al., 2011).
효모에서 인간화 당화를 달성하려면 한편으로는 적절한 효모 유전자 삭제에 의한 과당화의 제거가 요구되고 또 다른 한편으로는 효모에는 존재하지 않는 시알산과 같은 당에 대한 운반체와 결핍된 생합성 경로를 포함하는 추가적인 당분해효소와 당전이효소를 도입해주어야 한다(De Pourcq et al., 2010, Hamilton & Gerngross, 2007). 성공적인 외래 당화 경로 조립을 위해서는 단순한 유전자 발현뿐만 아니라 소포체(ER)과 골지체에서의 정확한 공간적 위치 선정은 필수적이다. 이러한 효모에서 세포 조립 라인에 필요한 요소들의 적합한 위치 선정을 이루기 위해서는 합성 당생물학적 접근이 필요하다(Czlapinski & Bertozzi, 2006). 진핵생물의 당전이효소와 당분해효소는 N-말단의 세포질 꼬리와 막 고정 도메인, 줄기 영역, 그리고 C-말단의 촉매 영역을 가진 유형 2 막단백질로서 골지체 막에 위치하고 있다. C-말단의 촉매 영역은 'CTS'(세포질, 막관통, 줄기)라고도 불리는 N-말단 부분을 부여하는 위치 선정과 독립적으로 작동한다. 따라서 P. pastoris를 대상으로 인간화된 당화경로 조립에 성공한 미국의 GlycoFi 연구팀의 경우 타 생물체 유래의 당전이효소와 당분해효소를 효모의 골지체 막에 정확하게 발현시키기 위해서는 C-말단의 촉매 영역과 CTS 단편들의 다양한 조합으로 구성된 합성 당전이효소와 당분해효소 라이브러리를 제작하였다. 고속 대량 선별법과 병행한 합성 당전이/분해 효소 라이브러리 활용 접근 방식은 최적의 외래 당화 경로가 조립된 형질전환체를 선별할 수 있었다(Choi et al., 2003). 최근 연구논문을 통해 미국의 GlycoFi사는 서로 다른 진핵생물(진균류, 벌레, 초파리, 생쥐, 쥐, 인간)의 33가지 촉매 영역과 66가지 진균류의 선도 서열, 촉매 영역과 CTS를 다양한 길이로 융합시켜 합성 당전이/분해 라이브러리를 제작하였으며, P. pastoris에서 만들어진 인공 당화 경로를 따라 최대 600개의 변이 최적의 원하는 활성과 위치 선정에 대한 선별을 하였음을 보고하였다(Nett et al., 2011). 최종적으로 효모와 곰팡이에는 결여되어 있는 시알산 생합성 경로를 P. pastoris에 조립하기 위해 부가적으로 9개의 합성 외래 유전자를 도입하여 시알산이 말단에 첨가된 인체형 복합형 당사슬이 부착된 당단백질 생산에 성공하였다(Hamilton et al., 2006).
이와 같은 연구결과는 합성 당생물학적 연구를 통해 인간화 당화 경로 조립뿐만 아니라 다양한 형태의 당사슬이 부착되도록 복잡한 당화 경로를 직접 제어할 수 가능성을 제시해 주고 있다. 즉, 당사슬 구조가 조정된 인공 효모 균주를 활용하여 다양한 약력학을 나타내는 맞춤형 당구조가 부착된 당단백질를 만들어 냄으로써, 자연보다 더 나은 기능을 지닌 새로운 당단백질 의약품 생산을 위한 길을 열어준다.
4. 유용 대사산물 대량생산을 위한 효모합성 생물학
다양한 생명체들이 생산하는 이차대사산물은 고부가가치 화학화합물의 주요 공급처로서 많은 경우 중요한 의약적인 성질을 지니고 있다. 특히 식물의 대사산물들은 항산화제 또는 항생제 기능을 통해 박테리아, 바이러스, 곰팡이 등의 감염을 막아주며, 또한 인체 건강에 유익한 기능성 물질로 치료제로서의 활용도가 높아 미생물을 활용한 대량생산 기술에 대한 수요가 높다. 그 중 효모는 다양한 이차대사산물 생합성 경로를 도입하여 발현시킬 수 있는 숙주로 관심을 받고 있다. 이차대사 생합성 경로가 자체적으로 제한적인 효모는 유전공학을 통해 도입된 외래 대사 경로를 방해하거나 경쟁하지 않으며, 무엇보다 다양한 오믹스 분석 시스템이 잘 구축되어 있어 전사체와 대사체 분석을 통해 효모 숙주의 생리 상태에 대한 총체적인 정보를 확보할 수 있는 장점이 있다. 또한 대사과정에 대한 상세한 모델이 개발되어 변형된 대사 네트워크의 행동을 예측할 수 있는 인실리코(in silico) 효모가 구축되어 있어 이를 활용한 인공 세포 디자인 및 제작이 더욱 용이한 점이 큰 장점이다(Duarte et al., 2004). 더 나아가 단세포 진핵 미생물로서 효모는 소포체 및 미토콘드리아 같은 소기관에서 발현되어야만 활성을 갖게 되는 cytochrome P450 같은 외래 효소 발현에 적합한 숙주이며, 식물 및 동물 유래의 효소 활성에 필수적인 단백질 번역 후 변형능이 있는 점도 원핵 미생물 숙주에 비해 갖는 장점이다.
이소프레노이드는 식품, 의학, 약학, 바이오 연료 및 화학 등 다양한 분야의 산업체에 활용되고 있는 유기 합성체의 큰 그룹으로서, 식물 이차 대사산물 중 가장 큰 그룹을 차지하고 있다. 이소프레노이드류 대표적 중간물질로는 파네솔(farnesol), 게라닐게라니올(geranylgeraniol), 아모파디엔(amorphadiene) 등이 포함되며, 이러한 다양한 구조의 이소프레노이드는 생합성 전구체인 Isopentenyl diphosphate (IPP)와 이성질체인 dimethylallyl diphosphate (DMAPP)로부터 합성되며, 이들의 전구체는 효모의 경우 메발론산(mevalonate, MVA) 생합성 대사 경로를 통해 생산된다(그림 3). 효모를 이소프레노이드 생산 균주로 개발하는 경우 불리한 점은 새로이 도입된 생합성 경로와 원래 효모 생합성 경로, 예를 들면 세포막 형성에 필요한 에르고스테롤(ergosterol)과 교배에 관여하는 a-요소(a-factor) 생합성 경로가 전구체를 두고 경쟁을 하게 되는 것이다. 따라서 효모 합성생물학자들은 주요 전구체 합성 경로를 증폭시키면서 새로이 도입된 외래 경로로 대사 흐름을 조절하는 전략을 구상해야 한다. 이를 위해 공통적으로 활용하는 전략은 이소프레노이드 생합성의 대사흐름 증가를 위해 메발론산 생산에 있어 가장 중요한 율속 단계로 알려져 있는 HMG-CoA 효소(hydroxymethylglutaryl CoA reductase) 반응을 HMG 유전자를 과발현시키는 장치를 개발하는 것이며, 경쟁 관계에 있는 효모 특이적 대사 흐름을 조절 가능한 유전자 부품을 사용하여 차단하는 것이다.
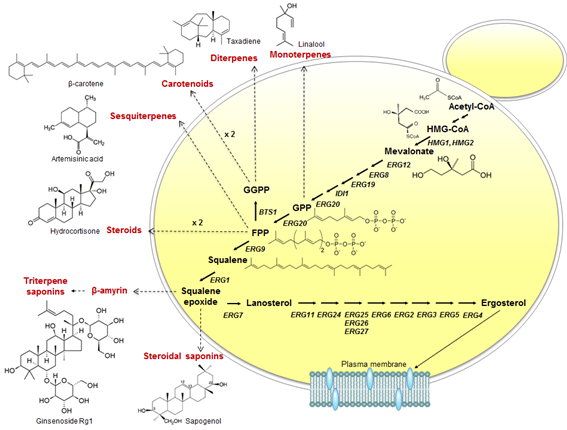
그림 3. 효모의 이소프레노이드 생합성 경로 기반 유용 이차 대사산물 생산. GPP. geranyl pyrophosphate; FPP, farnesyl
pyrophosphate; GGPP, geranylgeranyl pyrophosphate.
1) 아르테미시닌(artemisinin) 생산 합성 효모 개발
효모 합성생물학의 가장 대표적인 성공사례는 빌 게이츠 재단의 연구지원을 받아 합성생물학을 통해 미국 Keasling 그룹이 개발한 아르테미시닌 생산 효모 균주이다. 아르테미시닌은 개똥쑥(Artemisia annua)에서 추출되던 고가의 말라리아 치료제로서 이소프레노이드(isoprenoid)의 일종으로, 이들은 효모 S. cerevisiae로 약 10개 이상의 외래 유전자를 도입하여 성공적으로 아르테미시닌 전구체 아모파디엔(amorpha-4,11-diene)을 생산하였다(Ro et al., 2006). 초기의 연구에서는 효모에서 메발론산 생산 증대를 위해 조절 억제에 관여하는 N-말단이 제거된 HMG에 대한 유전자 tHMG1를 과발현시키고 파네실 다이포스페이트(farnesyl diphosphate, FPP)가 스퀘알렌(squalene)으로 전환되는 단계를 차단하기 위해 ERG9 유전자 발현을 MET3 프로모터를 활용하여 억제하고 FPP가 아모파디엔으로 전환되도록 아모파디엔 합성 유전자 ADS를 도입하였다. 식물 유래의 아모파디엔 옥시다제 시토크롬 P450과 환원제에 대한 유전자인 CYP71AV1와 CPR1, cytochrome b5 유전자를 효모에 도입하여 아르테미시닉산까지 생산되는 합성 효모를 제작하였다(Ro et al., 2006). 후속 연구에서는 보다 상업적으로 활용 가능한 아르테미시닌 생합성 균주개발을 위해 ERG9 유전자 발현을 메티오닌에 비해 저렴한 억제제인 구리를 사용할 수 있도록 CTR3 프로모터로 대체하였으며, 아르테미시닉 알데하이드로 축적되는 것을 막기 위해 식물 유래의 artemisinic aldehyde dehydrogenase (ALDH1)와 알콜디하이드로젠(ADH1) 유전자들을 함께 추가로 도입시켜 아모파디엔이 아르테미시닌으로 전환시키는 단계를 성공적으로 완성시켰다(Paddon et al., 2013).
2) 진세노사이드 생산 합성 효모 개발
아시아 인삼(Panax ginseng C.A.Meer) 및 미국 인삼(Panax quinquefolius L.)이 지닌 다양한 생리활성의 주역을 담당하고 있는 진세노사이드를 효모에서 대량생산하기 위한 기술 개발도 최근 들어 주목을 받기 시작하였다. 진세노사이드는 식물체에서 메발론산 생합성 경로를 포함한 이소프레노이드 합성경로를 통해 생합성(Cristensen, 2008)되므로, 효모의 에르고스테롤 생합성 경로를 재설계하여 진세노사이드 생산 균주를 개발하려는 합성생물학 연구가 시도되고 있다. 최근 중국의 Huang과 Zhang 공동 연구팀에서 인삼의 프로토파나사디올 다마레네디올-II 합성효소(protopanaxadiol dammarenediol-II synthase) 프로토파나사디올 합성 효소(protopanaxadiol synthasegenes) 유전자를 Arabidopsis thaliana에서 확보한 NADPH-cytochrome P450 reductase 유전자와 함께 효모 S. cerevisiae에 발현시켜 프로토파나사디올을 생산하는 데 성공하였음을 보고하였다. 이들은 스퀘알렌과 2, 3-옥시스퀘알렌(2,3-oxidosqualene) 공급을 증폭시키기 위해 N-말단 HMG 유전자 tHMG1를 과발현시켰으며, FPP 합성효소 유전자(ERG20), 스퀘알렌 합성 효소 유전자(EFG9), 2, 3-옥시스퀘알렌 합성 효소 유전자(ERG1)도 동시에 과발현시켜 프로토파나사디올 생산에 필요한 전구체 공급을 증폭시켰다. 또한 효모 코돈에 맞추어 프로토파나사디올 합성 유전자를 합성하여 더욱 프로토파나사디올 전환 효율을 높이고 최종적으로 우리딘 다이포스페이트 글라이코실 트라스퍼라제(uridin diphosphate glycosyl-transferase) 유전자를 도입해서 진세노사이드 생합성 경로를 완성하였다(Dai et al., 2013). 추후, 보다 최적화 작업을 통해 진세노사이드 생산 합성 효모는 식물체로부터 추출해내는 복잡한 공정 과정을 대체할 수 있는 경제적인 생산 공정을 제공할 것으로 기대되고 있다.
5. 고찰
합성생물학은 최근 주목 받고 있는 녹색산업과 헬스케어의 두 유망사업을 지원하는 핵심 기반 기술로서 자리매김을 하면서 21세기 산업에 미치는 그 파급효과는 매우 클 것으로 예상되며, 합성생물학을 통해 제작된 인공 생명체를 사용하여 바이오 연료 및 화학제품, 신약 개발 분야의 일대 혁신을 일으킬 것으로 기대되고 있다. 특히, 단세포 진핵 미생물로서 원핵 미생물이 지니지 못한 장점을 지닌 효모 대상의 합성생물학 연구는 고등생물체 유래의 치료용 단백질과 고기능성 대사물질을 대량으로 저렴하게 생산하는 효모 세포공장(cell factory) 개발을 통해 국민 보건 복지 증진 및 삶의 수준 향상에 크게 기여할 것이다.
인용문헌
Alper H, Fischer C, Nevoigt E, Stephanopoulos G (2005) Tuning genetic control through promoter engineering. Proceedings of the National Academy of Sciences of the United States of America 102: 12678-12683
Bayer TS, Smolke CD (2005) Programmable ligand-controlled riboregulators of eukaryotic gene expression. Nature biotechnology 23: 337-343
Berlec A, Strukelj B (2013) Current state and recent advances in biopharmaceutical production in Escherichia coli, yeasts and mammalian cells. Journal of industrial microbiology & biotechnology 40: 257-274
Blount BA, Weenink T, Vasylechko S, Ellis T (2012) Rational diversification of a promoter providing fine-tuned expression and orthogonal regulation for synthetic biology. PloS one 7: e33279
Cheon SA, Kim H, Oh DB, Kwon O, Kang HA (2012) Remodeling of the glycosylation pathway in the methylotrophic yeast Hansenula polymorpha to produce human hybrid-type N-glycans. J Microbiol. 50:341-348
Cho EY, Cheon SA, Kim H, Choo J, Lee DJ, Ryu HM, Rhee SK, Chung BH, Kim JY, Kang HA (2010) Multiple-yapsin-deficient mutant strains for high-level production of intact recombinant proteins in Saccharomyces cerevisiae. Journal of biotechnology 149: 1-7
Choi BK, Bobrowicz P, Davidson RC, Hamilton SR, Kung DH, Li H, Miele RG, Nett JH, Wildt S, Gerngross TU (2003) Use of combinatorial genetic libraries to humanize N-linked glycosylation in the yeast Pichia pastoris. Proceedings of the National Academy of Sciences of the United States of America 100: 5022-5027
Czlapinski JL, Bertozzi CR (2006) Synthetic glycobiology: Exploits in the Golgi compartment. Current opinion in chemical biology 10: 645-651
Dai Z, Liu Y, Zhang X, Shi M, Wang B, Wang D, Huang L (2013) Metabolic engineering of Saccharomyces cerevisiae for production of ginsenosides. Metabolic engineering 20: 146-156
De Pourcq K, De Schutter K, Callewaert N (2010) Engineering of glycosylation in yeast and other fungi: current state and perspectives. Applied microbiology and biotechnology 87: 1617-1631
Demain AL, Vaishnav P (2009) Production of recombinant proteins by microbes and higher organisms. Biotechnology advances 27: 297-306
Fang F, Salmon K, Shen MW, Aeling KA, Ito E, Irwin B, Tran UP, Hatfield GW, Da Silva NA, Sandmeyer S (2011) A vector set for systematic metabolic engineering in Saccharomyces cerevisiae. Yeast 28: 123-136
Feizi A, Osterlund T, Petranovic D, Bordel S, Nielsen J (2013) Genome-scale modeling of the protein secretory machinery in yeast. PloS one 8: e63284
Finnis CJ, Payne T, Hay J, Dodsworth N, Wilkinson D, Morton P, Saxton MJ, Tooth DJ, Evans RW, Goldenberg H, Scheiber-Mojdehkar B, Ternes N, Sleep D (2010) High-level production of animal-free recombinant transferrin from Saccharomyces cerevisiae. Microbial cell factories 9: 87
Goodman M (2009) Market watch: Sales of biologics to show robust growth through to 2013. Nature reviews Drug discovery 8: 837
Guerfal M, Ryckaert S, Jacobs PP, Ameloot P, Van Craenenbroeck K, Derycke R, Callewaert N (2010) The HAC1 gene from Pichia pastoris: characterization and effect of its overexpression on the production of secreted, surface displayed and membrane proteins. Microbial cell factories 9: 49
Hamilton SR, Davidson RC, Sethuraman N, Nett JH, Jiang Y, Rios S, Bobrowicz P, Stadheim TA, Li H, Choi BK, Hopkins D, Wischnewski H, Roser J, Mitchell T, Strawbridge RR, Hoopes J, Wildt S, Gerngross TU (2006) Humanization of yeast to produce complex terminally sialylated glycoproteins. Science 313: 1441-1443
Hamilton SR, Gerngross TU (2007) Glycosylation engineering in yeast: the advent of fully humanized yeast. Curr Opin Biotechnol 18: 387-392
Harbison CT, Gordon DB, Lee TI, Rinaldi NJ, Macisaac KD, Danford TW, Hannett NM, Tagne JB, Reynolds DB, Yoo J, Jennings EG, Zeitlinger J, Pokholok DK, Kellis M, Rolfe PA, Takusagawa KT, Lander ES, Gifford DK, Fraenkel E, Young RA (2004) Transcriptional regulatory code of a eukaryotic genome. Nature 431: 99-104
Hawkins KM, Smolke CD (2008) Production of benzylisoquinoline alkaloids in Saccharomyces cerevisiae. Nature chemical biology 4: 564-573
Hou J, Osterlund T, Liu Z, Petranovic D, Nielsen J (2013) Heat shock response improves heterologous protein secretion in Saccharomyces cerevisiae. Applied microbiology and biotechnology 97: 3559-3568
Hou J, Tyo K, Liu Z, Petranovic D, Nielsen J (2012a) Engineering of vesicle trafficking improves heterologous protein secretion in Saccharomyces cerevisiae. Metabolic engineering 14: 120-127
Hou J, Tyo KE, Liu Z, Petranovic D, Nielsen J (2012b) Metabolic engineering of recombinant protein secretion by Saccharomyces cerevisiae. FEMS yeast research 12: 491-510
Ilmen M, den Haan R, Brevnova E, McBride J, Wiswall E, Froehlich A, Koivula A, Voutilainen SP, Siika-Aho M, la Grange DC, Thorngren N, Ahlgren S, Mellon M, Deleault K, Rajgarhia V, van Zyl WH, Penttila M (2011) High level secretion of cellobiohydrolases by Saccharomyces cerevisiae. Biotechnology for biofuels 4: 30
Isaacs FJ, Dwyer DJ, Ding C, Pervouchine DD, Cantor CR, Collins JJ (2004) Engineered riboregulators enable post-transcriptional control of gene expression. Nature biotechnology 22: 841-847
Jeppsson M, Johansson B, Jensen PR, Hahn-Hagerdal B, Gorwa-Grauslund MF (2003) The level of glucose-6-phosphate dehydrogenase activity strongly influences xylose fermentation and inhibitor sensitivity in recombinant Saccharomyces cerevisiae strains. Yeast 20: 1263-1272
Jonikas MC, Collins SR, Denic V, Oh E, Quan EM, Schmid V, Weibezahn J, Schwappach B, Walter P, Weissman JS, Schuldiner M (2009) Comprehensive characterization of genes required for protein folding in the endoplasmic reticulum. Science 323: 1693-1697
de Jong JF, Ohm RA, de Bekker C, Wosten HA, Lugones LG (2010) Inactivation of ku80 in the mushroom-forming fungus Schizophyllum commune increases the relative incidence of homologous recombination. FEMS microbiology letters 310: 91-95
Kellis M, Patterson N, Endrizzi M, Birren B, Lander ES (2003) Sequencing and comparison of yeast species to identify genes and regulatory elements. Nature 423: 241-254
Kim EJ, Park YK, Lim HK, Park YC, Seo JH (2009) Expression of hepatitis B surface antigen S domain in recombinant Saccharomyces cerevisiae using GAL1 promoter. Journal of biotechnology 141: 155-159
Kim MD, Han KC, Kang HA, Rhee SK, Seo JH (2003) Coexpression of BiP increased antithrombotic hirudin production in recombinant Saccharomyces cerevisiae. Journal of biotechnology 101: 81-87
Kim MD, Park EH, Cho JW, Kim JC, Cho SM, Han MR, Seo JH (2007) Enhanced production of antithrombotic hirudin by coexpression of Pdi1 and Ero1 in recombinant Saccharomyces cerevisiae. Journal of biotechnology 131: S147-S147
Kouprina N, Larionov V (2008) Selective isolation of genomic loci from complex genomes by transformation-associated recombination cloning in the yeast Saccharomyces cerevisiae. Nature protocols 3: 371-377
Krivoruchko A, Siewers V, Nielsen J (2011) Opportunities for yeast metabolic engineering: Lessons from synthetic biology. Biotechnology journal 6: 262-276
Kudla B, Nicolas A (1992) A multisite integrative cassette for the yeast Saccharomyces cerevisiae. Gene 119: 49-56
Labbe S, Thiele DJ (1999) Copper ion inducible and repressible promoter systems in yeast. Methods in enzymology 306: 145-153
Liang J, Luo YZ, Zhao HM (2011) Synthetic biology: putting synthesis into biology. Wiley Interdiscip Rev Syst Biol Med 3: 7-20
Liu L, Martinez JL, Liu Z, Petranovic D, Nielsen J (2014) Balanced globin protein expression and heme biosynthesis improve production of human hemoglobin in Saccharomyces cerevisiae. Metabolic engineering 21: 9-16
Lopes TS, de Wijs IJ, Steenhauer SI, Verbakel J, Planta RJ (1996) Factors affecting the mitotic stability of high-copy-number integration into the ribosomal DNA of Saccharomyces cerevisiae. Yeast 12: 467-477
Lopes TS, Klootwijk J, Veenstra AE, van der Aar PC, van Heerikhuizen H, Raúe HA, Planta RJ (1989) High-copy-number integration into the ribosomal DNA of Saccharomyces cerevisiae: a new vector for high-level expression. Gene 15: 199-206
Lynch SA, Gill RT (2012) Synthetic biology: new strategies for directing design. Metabolic engineering 14: 205-211
Martinez JL, Liu L, Petranovic D, Nielsen J (2012) Pharmaceutical protein production by yeast: towards production of human blood proteins by microbial fermentation. Curr Opin Biotechnol 23: 965-971
Mattanovich D, Branduardi P, Dato L, Gasser B, Sauer M, Porro D (2012) Recombinant protein production in yeasts. Methods in molecular biology 824: 329-358
McIsaac RS, Gibney PA, Chandran SS, Benjamin KR, Botstein D (2014) Synthetic biology tools for programming gene expression without nutritional perturbations in Saccharomyces cerevisiae. Nucleic acids research
Mellitzer A, Weis R, Glieder A, Flicker K (2012) Expression of lignocellulolytic enzymes in Pichia pastoris. Microbial cell factories 11: 61
Mumberg D, Muller R, Funk M (1994) Regulatable promoters of Saccharomyces cerevisiae: comparison of transcriptional activity and their use for heterologous expression. Nucleic acids research 22: 5767-5768
Mumberg D, Muller R, Funk M (1995) Yeast vectors for the controlled expression of heterologous proteins in different genetic backgrounds. Gene 156: 119-122
Murooka Y, Imanaka T (1993) Recombinant Microbes for Industrial and Agricultural Applications: MARCEL DEKKER, INC.
Naatsaari L, Mistlberger B, Ruth C, Hajek T, Hartner FS, Glieder A (2012) Deletion of the Pichia pastoris KU70 homologue facilitates platform strain generation for gene expression and synthetic biology. PloS one 7: e39720
Naesby M, Nielsen SV, Nielsen CA, Green T, Tange TO, Simon E, Knechtle P, Hansson A, Schwab MS, Titiz O, Folly C, Archila RE, Maver M, van Sint Fiet S, Boussemghoune T, Janes M, Kumar AS, Sonkar SP, Mitra PP, Benjamin VA et al (2009) Yeast artificial chromosomes employed for random assembly of biosynthetic pathways and production of diverse compounds in Saccharomyces cerevisiae. Microbial cell factories 8: 45
Naesby M, Nielsen SV, Nielsen CA, Green T, Tange TO, Simon E, Knechtle P, Hansson A, Schwab MS, Titiz O, Folly C, Archila RE, Maver M, van Sint Fiet S, Boussemghoune T, Janes M, Kumar AS, Sonkar SP, Mitra PP, Benjamin VA et al (2009) Yeast artificial chromosomes employed for random assembly of biosynthetic pathways and production of diverse compounds in Saccharomyces cerevisiae. Microbial cell factories 8: 45
Nett JH, Stadheim TA, Li H, Bobrowicz P, Hamilton SR, Davidson RC, Choi BK, Mitchell T, Bobrowicz B, Rittenhour A, Wildt S, Gerngross TU (2011) A combinatorial genetic library approach to target heterologous glycosylation enzymes to the endoplasmic reticulum or the Golgi apparatus of Pichia pastoris. Yeast 28: 237-252
Nielsen J (2013) Production of biopharmaceutical proteins by yeast: advances through metabolic engineering. Bioengineered 4: 207-211
Oliveira C, Teixeira JA, Lima N, Da Silva NA, Domingues L (2007) Development of stable flocculent Saccharomyces cerevisiae strain for continuous Aspergillus niger beta-galactosidase production. Journal of bioscience and bioengineering 103: 318-324
Paddon CJ, Westfall PJ, Pitera DJ, Benjamin K, Fisher K, McPhee D, Leavell MD, Tai A, Main A, Eng D, Polichuk DR, Teoh KH, Reed DW, Treynor T, Lenihan J, Fleck M, Bajad S, Dang G, Dengrove D, Diola D et al (2013) High-level semi-synthetic production of the potent antimalarial artemisinin. Nature 496: 528-532
Palmberger D, Klausberger M, Berger I, Grabherr R (2013) MultiBac turns sweet. Bioengineered 4: 78-83
Pan R, Zhang J, Shen WL, Tao ZQ, Li SP, Yan X (2011) Sequential deletion of Pichia pastoris genes by a self-excisable cassette. FEMS yeast research 11: 292-298
Park JN, Song Y, Cheon SA, Kwon O, Oh DB, Jigami Y, Kim JY, Kang HA (2011) Essential role of YlMPO1, a novel Yarrowia lipolytica homologue of Saccharomyces cerevisiae MNN4, in mannosylphosphorylation of N- and O-linked glycans. Appl Environ Microbiol. 77:1187-1195
Partow S, Siewers V, Bjorn S, Nielsen J, Maury J (2010) Characterization of different promoters for designing a new expression vector in Saccharomyces cerevisiae. Yeast 27: 955-964
Patil C, Walter P (2001) Intracellular signaling from the endoplasmic reticulum to the nucleus: the unfolded protein response in yeast and mammals. Current opinion in cell biology 13: 349-355
Payne T, Finnis C, Evans LR, Mead DJ, Avery SV, Archer DB, Sleep D (2008) Modulation of chaperone gene expression in mutagenized Saccharomyces cerevisiae strains developed for recombinant human albumin production results in increased production of multiple heterologous proteins. Applied and environmental microbiology 74: 7759-7766
Qin X, Qian J, Yao G, Zhuang Y, Zhang S, Chu J (2011) GAP promoter library for fine-tuning of gene expression in Pichia pastoris. Applied and environmental microbiology 77: 3600-3608
Ro DK, Paradise EM, Ouellet M, Fisher KJ, Newman KL, Ndungu JM, Ho KA, Eachus RA, Ham TS, Kirby J, Chang MC, Withers ST, Shiba Y, Sarpong R, Keasling JD (2006) Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature 440: 940-943
Ruth C, Zuellig T, Mellitzer A, Weis R, Looser V, Kovar K, Glieder A (2010) Variable production windows for porcine trypsinogen employing synthetic inducible promoter variants in Pichia pastoris. Systems and synthetic biology 4: 181-191
Schroder M (2008) Engineering eukaryotic protein factories. Biotechnology letters 30: 187-196
Verbeke J, Beopoulos A, Nicaud JM (2013) Efficient homologous recombination with short length flanking fragments in Ku70 deficient Yarrowia lipolytica strains. Biotechnology letters 35: 571-576
Vogl T, Glieder A (2013) Regulation of Pichia pastoris promoters and its consequences for protein production. New biotechnology 30: 385-404
Walsh G (2010a) Biopharmaceutical benchmarks 2010. Nature biotechnology 28: 917-924
Walsh G (2010b) Post-translational modifications of protein biopharmaceuticals. Drug discovery today 15: 773-780
Wingler LM, Cornish VW (2011) Reiterative Recombination for the in vivo assembly of libraries of multigene pathways. Proceedings of the National Academy of Sciences of the United States of America 108: 15135-15140
Xuan Y, Zhou X, Zhang W, Zhang X, Song Z, Zhang Y (2009) An upstream activation sequence controls the expression of AOX1 gene in Pichia pastoris. FEMS yeast research 9: 1271-1282
Zhang G, Ignatova Z (2011) Folding at the birth of the nascent chain: coordinating translation with co-translational folding. Curr Opin Struct Biol 21: 25-31





