박테리아 ncRNA를 통한 스트레스의 조절

한국과학기술원 이영훈 교수
1. 개요
박테리아는 생존을 위협하는 여러 가지 환경스트레스에 노출 되고 있고, 이를 극복하기 위한 다양한 방법을 사용하고 있다. 환경변화를 감지하여 다양한 유전자의 발현과 단백질들의 활성을 조절하는 신호전달 체계를 조정한다. 세포내의 RNA 유전체 (RNomics) 분석을 통해서, 단백질 합성정보를 암호화하고 있는 messenger RNAs (mRNAs)외에 세포 대사조절에 관여하는 다양한 타입의 RNA가 존재하고 있음이 알려졌다. 다양한 박테리아에서 지속적으로 밝혀지고 있는 이러한 작은 단백질을 코드하지 않는 작은 RNA (small non-coding RNA: ncRNA)들은 기존의 2개-인자 신호전달계 (two-component signal transduction system) 및 다양한 조절단백질과 더불어 스트레스 대응을 위한 핵심요소로써의 기능을 하고 있는 경우가 많았다. (Abu-Qatouseh et al., 2010; Altuvia, 2007; Pichon and Felden, 2008; Raabe et al., 2011). 가장 잘 정립되어 있는 ncRNA의 작용기작은 타겟 mRNA와 염기쌍을 이루거나 타겟 단백질과 상호작용하여 RNA-단백질 복합체 (Ribonucleoprotein; RNP)를 만드는 것이다. ncRNA는 유전체 상에 존재하는 위치에 따라서 시스로 코드되는 (cis-encoded) ncRNA 또는 트랜스로 코드되는 (trans-encoded ncRNA)로 분류하기도 한다. 시스로 코드되는 ncRNA는 타겟 mRNA의 바로 반대가닥에 존재하는 ncRNA로 타겟 mRNA와 완전한 염기쌍을 이룰 수 있는 반면, 타겟 mRNA 유전자와 멀리 떨어진 유전자에서 트랜스로 코드되는 ncRNA는 타겟 mRNA와 부분적인 염기쌍을 이룬다 (Richards and Vanderpool, 2011). 트랜스 코드되는 ncRNA의 경우, 기능을 위해서 샤페론 단백질인 Hfq가 필요한 경우가 많다. 102개 아미노산으로 이루어진 Hfq 단백질은 AU가 많은 단일가닥성 RNA와 상호작용한다고 알려져 있는데 (Geissmann et al., 2006; Moller et al., 2002), 박테리아 종간에 보존성이 높으며 스트레스 대응을 포함한 다양한 세포기능에 영향을 미친다. hfq 돌연변이는 다양한 병원성 박테리아의 병원성을 매우 감소시킨다. mRNA의 5′ 말단 비번역 영역 (5′ untranslated regions; 5′ UTR)은 구조변화 등을 통해서 mRNA의 발현을 조절하는 경우가 많은데, ncRNA가 인식하는 타겟영역 또한 5′ UTR이 되는 경우가 많았다. 본 연구동향에서는 박테리아 ncRNA가 스트레스에 대응하기 위하 위하여 세포대사에 어떻게 관여하고 있는지를 몇가지 실험 결괴들의 예를 통해 살펴보고자 한다.
2. 온도 스트레스와 ncRNA
박테리아는 온도변화에 민감하게 대응하기 위한 복잡한 피드백 조절기작들을 가지고 있다. ‘RNA 온도계 (RNA thermometers)’라고 부르는 RNA 영역의 온도에 따른 구조변화를 통한 피드백 시스템은, 온도변화에 빠르게 대응할 수 있다. RNA 온도계는 주로 5′ UTR 영역에서 리보좀 결합자리를 폐쇄하는 형태로 2차구조를 형성하고 있다가, 높은 온도에서 구조가 변화하여 단백질 발현이 가능하도록 한다 (Kortmann and Narberhaus, 2012). ROSE 요소 (Repression of the heat shock gene expression element: ROSE)는 열충격 단백질에서 흔하게 나타나는 RNA 온도계중 하나로, SD 서열 (Shine-Dalgarno sequence) 근처에 U(U/C)GCU 공통서열을 갖고 60~100 nt의 영역으로 존재한다. 2개 이상의 머리핀 구조가 포함되는데, SD 서열을 포함하는 3′ 머리핀구조가 고온에서 풀어지면서 mRNA 번역을 촉진시킨다 (Waldminghaus et al., 2009). 4U 요소 (fourU element)는 Salmonella agsA (aggregation suppressing A) mRNA의 5′ UTR에 나타나는데, 낮은 온도에서 네 개의 우리딘 염기서열이 SD 서열의 AGGA 염기서열과 함께 머리핀 구조를 형성한다. 마찬가지로 온도가 높아지면 구조가 불안정해지고 mRNA의 발현이 촉진된다.
RNA 온도계는 저온 스트레스에서도 중요한 기능을 한다. 저온에서 발현되어 RNA 샤페론의 기능을 하는 CspA의 경우, 저온에서 완전히 다른 형태의 5′ UTR 구조를 가짐으로써 세포내 mRNA의 안정성이 증가되어 단백질 발현이 증가된다 (Giuliodori et al., 2010).
트랜스로 코드되는 ncRNA인 DsrA는 두가지 전사체의 형태로 존재하는데, 온도변화에 따라 두가지 전사체의 비율이 달라지고, 이를 통해 스트레스 대응을 위한 RpoS의 발현을 촉진시킨다 (Repoila and Gottesman, 2001).
토양미생물인 L. monocytogenes의 prfA mRNA의 경우, 사람의 체온인 37℃에서 5′UTR의 구조가 변화되면 발현이 촉진되고 병원성을 일으키는 여러 가지 유전자를 발현시키게 된다 (Johansson et al., 2002).
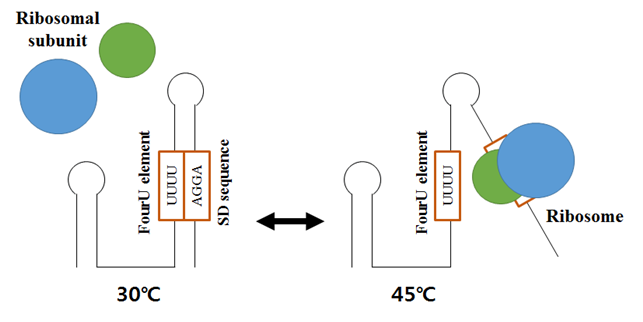
그림 1. 온도 스트레스를 극복하기 위한 ncRNA의 작용기작. agsA mRNA는 RNA 온도계인 4U 요소를 통해서 낮은 온도에서는 리보좀의 결합이 저해되고, 높은 온도에서는 지퍼와 유사한 메커니즘으로 불안정화되어 리보좀의 결합이 가능해지고 유전자가 발현된다.
3. 대사산물/양분 스트레스와 ncRNA
미생물들은 종종 영양결핍으로 인한 성장정체상태로 발견된다. 세포내에 글루코오스-6-인산 (glucose-6-phosphate)이 축적되면 세포성장이 멈추고 죽음에 이르기도 한다 (Englesberg et al., 1962). 이러한 스트레스 상태에서 Hfq-결합 ncRNA 중 하나인 SgrS ncRNA가 발현되고, 글루코오스 운반체 중의 하나를 코드하는 ptsG mRNA와 상호작용하여 리보핵산가수분해효소 E (RNase E) 의존적인 방식으로 ptsG mRNA를 분해한다 (Maki et al., 2010; Morita et al., 2005). SgrS ncRNA는 SgrT라는 짧은 펩타이드를 발현하기도 하는데, SgrT는 PtsG 단백질의 기능을 저해한다 (Wadler and Vanderpool, 2011).
탄소저장 조절시스템의 핵심 역할을 하는 CsrA 단백질의 활성은 BarA/UvrY 2개-인자 신호전달계에 의해서 간접적으로 조절된다. 탄소결핍상황에서 UvrY에 의해 두가지 ncRNA, CsrB와 CsrC가 빠르게 발현되는데, 이 두가지 ncRNA는 다수의 CsrA 단백질 결합자리를 갖고 있어서 CsrA 단백질과 결합함으로써 해당작용에 관여하는 유전자의 발현을 저해하고, 글루코오스 신생합성에 관여하는 유전자들을 발현한다 (Pernestig et al., 2003).
Caulobacter crescentus의 CrfA ncRNA는 탄소결핍 상황에서 발현되어 CC3461 수용기 mRNA의 5′ UTR과 염기쌍을 이루어, mRNA의 세포내 안정성을 증가시켜서 더 다양한 종류의 탄소원이 세포내로 유입될 수 있도록 한다 (Landt et al., 2010).
Staphylococcus, Macrococcus, Bacillus등에서 잘 보존되어 있는 RsaE ncRNA는 양분이 부족해지는 지수성장기 후반부에서 발현이 증가된다. 17 nt의 단일가닥부분 양쪽으로 머리핀구조를 형성하고 있는 이 ncRNA는, opp3 오페론의 처음과 마지막 mRNA인 opp3B, opp3A와 염기쌍을 이루어 발현을 저해시켜서 영양결핍상태에 잘 적응할 수 있도록 한다 (Bohn et al., 2010).
아미노산의 대사는 핵산이나 효소 보조인자등의 생성에 필수적이기 때문에 정확하게 제어될 필요가 있다. Gcv 오페론 (glycine cleavage operon)인 gcvTHP 오페론은 글라이신을 대사시켜 세포내 탄소원생합성에 이용되는 5,10-메틸렌테트라하이드로엽산을 생성하는데 필수적이다. 세포내에 글라이신 농도가 높아지면 Hfq와 상호작용하는 GcvB ncRNA의 발현이 활성화되고 짧은 펩타이드나, 다른 아미노산, 독성물질과 항생제등의 세포유입에 관여되는 유전자들의 발현을 억제한다 (Urbanowski et al., 2000). GcvB ncRNA는 mRNA 타겟들을 조절하는 두가지 안티센스영역을 가지고 있는데, ~1%의 Salmonella mRNA가 G/U가 많은 영역을 통해 조절됨이 보고되었고, cycA mRNA는 두가지 안티센스영역을 모두 사용하여 조절된다. 이는 타겟 mRNA와 염기쌍을 이루는 유연성을 나타내주는 예로 앞으로 더 다양한 타겟 mRNA가 찾아질 수 있음을 의미한다.
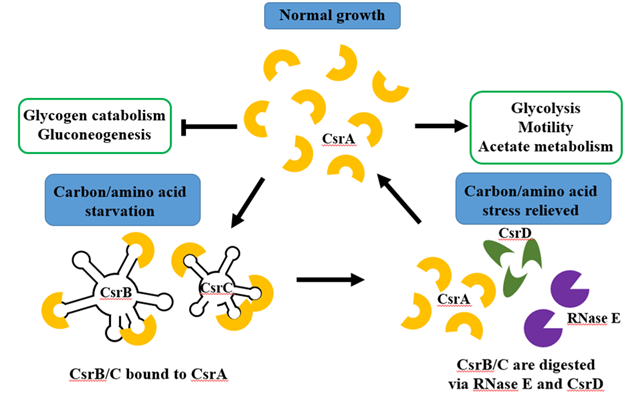
그림 2. 일반적인 성장조건에서 CsrA 단백질은 해당작용에 필요한 유전자를 발현시키고, 글루코오스 신생합성에 관련된 유전자들의 발현을 저해한다. 탄소원이 부족한 스트레스 상황에서 CsrB/CsrC ncRNA는 CsrA에 결합하여 CsrA의 활동을 방해한다. 스트레스상황에서 벗어나면 CsrB/CsrC는 CsrD와 RNase E를 매개로하여 분해된다.
4. 세포막 스트레스와 ncRNA
그람음성 박테리아의 세포외막 (outer membrane; OM)은 독성물질의 유입을 막는 중요한 역할을 한다. 포린 단백질들은 OM에 통로를 만들어 양분을 흡수하고 폐기물을 배출한다. E. coli와 Salmonella에서는 σE 인자 신호전달체계를 통해서 OM 단백질들의 상태가 감시된다. 잘못 형성된 OM 단백질들 (세포막 스트레스를 야기)이 많아지면 σE 인자가 활성화된다. 장내박테리아에서 보존성이 매우 높은 OmpA 단백질은 성장정지기에서 트랜스로 코드되는 ncRNA인 MicA에 의해서 RNase E 의존적으로 급격히 발현이 저해된다 (Rasmussen et al., 2005). σE 인자에 의해 발현되는 또 다른 ncRNA인 RybB ncRNA 또한 다수의 OM 단백질들의 발현을 조절하여 세포막 스트레스 상황에서 OM 단백질들의 양을 변화시킨다 (Papenfort et al., 2010). CyaR ncRNA는 ompX mRNA의 번역을 억제하며, V. cholerae의 VrrA ncRNA도 OmpA의 발현을 저해시키고, OMV (outer membrane vesicle)의 생성도 촉진시켜 세포막 스트레스를 더욱 완화한다.
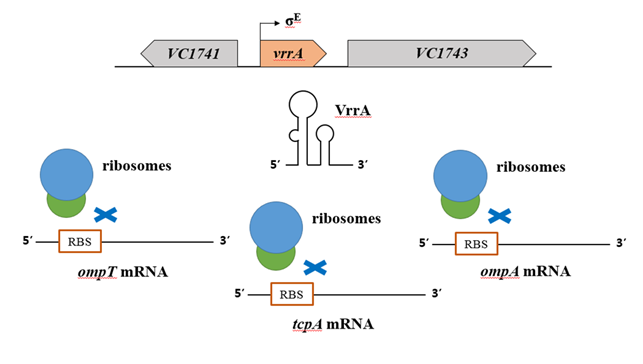
그림 3. 트랜스로 코드되는 ncRNA인 VrrA는 세포막 스트레스에 대응하여 발현이 유도된다. VrrA ncRNA는 ompT, tcpA, ompA mRNA의 리보좀 결합자리영역에 결합하여 유전자 발현을 저해한다.
5. 산화스트레스와 ncRNA
OxyR 전사인자는 H2O2에 의해서 티올-이황화 산화환원 (thiol-disulfide redox) 스위치를 통해 활성화되어, 카탈라아제 (catalase), 수산화과산화물 환원효소, 초산화물 불균등화효소 (superoxide dismutase) 등의 발현을 증가시켜서 산화스트레스에서 세포를 보호한다 (Pomposiello and Demple, 2001). OxyS ncRNA는 산화스트레스로 인해서 발현이 유도되어 많은 유전자들의 발현을 조절하고 돌연변이가 유발되는 것을 억제한다 (Altuvia et al., 1997).
많은 병원성 박테리아들은 숙주의 대식세포안에서 산화스트레스에 노출되게 되는데, B11, B55, F6, ASpks와 같은 ncRNA들이 감염 초기에 발현되어서 다양한 유전자의 발현을 조절한다 (Arnvig and Young, 2009).
6. 철분부족 스트레스와 ncRNA
철분은 세포성정과 분화, 전자전달계, 산소운반, 세포대사, 해독등의 세포활동에 반드시 필요한 요소이다. 하지만 높은 농도의 철분은 활성산소의 형성을 유발하여 세포에 해를 끼치기도 한다. 대부분의 박테리아에서 Fur (ferric uptake regulator) 단백질은 철분의 항상성 유지에 중심기능을 맡고 있다. 철분이 충분할 때는 철분 흡수 유전자들을 억제하고, 철분 저장 단백질들의 발현을 증가시킨다. RyhB ncRNA는 철분부족 스트레스에서 발현되는 ncRNA로 Fur 단백질에 의해 발현이 조절된다. 철분이 풍부할 때 RyhB ncRNA의 발현이 유도되고 sdhCDAB, acnA, fumA, bfr, ftnA, sodB mRNA 등 철분대사와 관여된 18개 오페론, 56개 단백질의 발현을 조절한다 (Masse et al., 2005). RyhB ncRNA는 fur mRNA의 발현도 억제하는 음성피드백시스템도 보여준다는 점이 주목할 만하다 (Vecerek et al., 2007).
V. cholerae, S. flexneri, S. dysenteriae 등에서 RyhB ncRNA의 상동체가 발견되고 있다. 그 기능도 더 다양하게 밝혀져서 바이오필름 형성이나 주화성 움직임에도 관여하고 있고 (Davis et al., 2005; Mey et al., 2005), 내산성의 감소와 병원성 억제에도 관여한다 (Murphy and Payne, 2007).
7. pH 스트레스와 ncRNA
위산의 극한산성조건을 견뎌내는 것은 박테리아들이 장내에 정착하기 위해 중요하다. 몇몇 그람양성균 및 그람음성균은 글루탐산을 이용한 내산성 시스템 (gad system)을 가지고 있어서 높은 농도의 H+ 스트레스로부터 세포를 보호할 수 있다. GadA, GadB 두 개의 글루탐산 탈카르복실화효소는 글루탐산을 이용해 γ-아미노부틸산을 만들면서 H+를 제거한다. GadE, GadX, GadW, CRP, H-NS, σS 등이 복잡하게 관여하여 내산성시스템을 조절한다. E. coli에서 Hfq와 상호작용하는 GadY ncRNA는 gadX mRNA의 3′ 말단부위에 시스로 코드되는 ncRNA로 gadX mRNA의 3′ 부위에 상보적으로 결합하여 mRNA를 RNase E의 분해로부터 보호한다 (Opdyke et al., 2004). GadX의 축적은 하위의 내산성 관련유전자를 발현시켜 낮은 pH에서 생존성을 증가시킨다 (Opdyke et al., 2011). GcvB ncRNA 또한 알 수 없는 기작으로 σS 의 발현을 증가시켜 낮은 pH에서의 생존성을 증가시키며 (Jin et al., 2009), ArcZ, DsrA, RprA ncRNA를 통한 σS 의 발현증가 역시 pH 2.0에서의 세포의 생존성에 큰 영향을 미친다 (Bak et al., 2014). 높은 pH에서는 아미노산 탈아민화와 당발효를 통한 산 생성증가, 세포막의 특성변화 등을 통해 스트레스를 극복한다. alx mRNA의 5′ UTR에는 PRE 리보 스위치 (pH responsive RNA element)가 있어서 높은 pH에서만 Alx 단백질을 발현시킨다 (Nechooshtan et al., 2009).
8. CRISPR: 스트레스 관리를 위한 새로운 기작
박테리아는 바이러스, 파지, 플라스미드 등의 침입을 견뎌내기 위해서 다양한 전략들을 사용해왔다. 그중 CRISPR/Cas 시스템 (clustered regularly interspaced short palindromic repeats/CRISPR-associated proteins)은 ncRNA를 기반으로 한 방어시스템이다 (Barrangou and Horvath, 2012; Makarova et al., 2011; Westra et al., 2012). CRISPR 유전자 자리는 많게는 수백개의 간격 띄우게 염기서열들 (spacer sequences)이 반복서열 (repeat sequence) 사이로 규칙적으로 배열되어 있는 특성을 보인다. CRISPR 영역에서 전사된 선구 crRNA는 가공되어서 완성된 crRNA가 되고, 진행생물의 RNAi 시스템과 유사한 방식으로 Cas 단백질들과 함께 외부에서 유래한 RNA 또는 DNA를 절단한다. CRISPR/Cas 시스템은 고세균류에서는 항상 지속적으로 발현되고 있으나 (Hale et al., 2008; Tang et al., 2002), 특정 신호에 의존적인 경우도 많다. Thermus thermophilus의 CRISPR/Cas 전사체는 파지에 감염된 후에 발현이 증가되고, 대장균에서는 H-NS에 의해 casA의 발현이 조절되고 있기도 하다 (Pul et al., 2010). 세포막 스트레스에서 유도되는 BaeSR 2개-인자 조절시스템에 의해서 CRISPR/Cas 발현을 촉진시키는 경우도 보고되었다 (Perez-Rodriguez et al., 2011). 어떤 다른 스트레스들이 CRISPR/Cas 시스템의 발현조절과 어떤 기작으로 연결되어있는지 아직 밝혀지지 않았지만, 앞으로 스트레스와 CRISPR/Cas 발현사이에 많은 연관이 밝혀질 것으로 기대된다.
9. 고찰
최근 ncRNA 연구분야의 성과들은 스트레스 조절과 박테리아 병원성 등에 ncRNA가 어떻게 관여하고 있는지를 짐작케 한다. RNA 온도계, 시스 또는 트랜스로 코드되는 ncRNA 및 CRISPR 등은 다양한 스트레스에 대응하여 구조적으로 변화되거나, 타겟 mRNA로 부터 발현 또는 단백질의 활성을 조절한다. ncRNA를 활용한 스트레스의 대응은 단백질 전사인자를 이용한 스트레스 대응과 비교되는 점들이 있다. ncRNA는 짧고 단백질로 번역되지 않기 때문에 세포내 에너지 소모가 적고, 합성과 분해가 빠르다. 또한 ncRNA들은 전사후 단계에서 타겟들을 조절하는 경우가 많아서 스트레스 대응에 더 신속할 수 있다. 또한 ncRNA들은 추가적인 조절단계를 갖게 하는 기능도 한다. ncRNA와 전사인자단백질이 모두 관여하는 조절시스템의 경우 발현의 누수가 최소화되고 동역학적으로도 전사인자 하나만 사용하는 것보다 장점이 많다 (Levine and Hwa 2008).
대용량 실험기술과, 생물정보학적 기법들의 발달과 함께 박테리아 스트레스조절에 관여하는 다양한 ncRNA들이 더 많이 발견될 것으로 보인다. 약물 유사체에 대한 리보스위치를 활용하여 쥐에서 S. aureus의 병원성을 감소시키거나 (Mulhbacher et al., 2010), Cas9과 합성 가이드 ncRNA를 이용한 CRISPR 시스템을 염색체 조작에 활용하거나 (Cong et al., 2013; Jinek et al., 2012; Mali et al., 2013)하는 새로운 기술들로도 많이 적용되고 있다. 더 나아가 ncRNA를 진단 마커나 약물 타겟으로 하여 박테리아의 병원성을 조절하는 연구가 새로운 도전이 될 것으로 생각된다.
참고문헌
1. Abu-Qatouseh, L.F., Chinni, S.V., Seggewiss, J., Proctor, R.A., Brosius, J., Rozhdestvensky,
T.S., Peters, G., Von Eiff, C., Becker, K., 2010. Identification of differentially expressed small
non-protein-coding RNAs in Staphylococcus aureus displaying both the normal and the
small-colony variant phenotype. J. Mol. Med. 88, 565–575.
2. Altuvia, S., 2007. Identification of bacterial small non-coding RNAs: experimental
approaches. Curr. Opin. Microbiol. 10, 257–261.
3. Altuvia, S., Weinstein-Fischer, D., Zhang, A., Postow, L., Storz, G., 1997. A small, stable
RNA induced by oxidative stress: role as a pleiotropic regulator and antimutator.
Cell 90, 43–53.
4. Arnvig, K.B., Young, D.B., 2009. Identification of small RNAs in Mycobacterium tuberculosis.
Mol. Microbiol. 73, 397–408.
5. Bak, G., Han, K., Kim, D., Lee, Y., 2014. Roles of rpoS-activating small RNAs in pathways
leading to acid resistance of Escherichia coli. MicrobiologyOpen. 3, 15-28.
6. Barrangou, R., Horvath, P., 2012. CRISPR: new horizons in phage resistance and strain
identification. Annu. Rev. Food Sci. Technol. 3, 143–162.
7. Bohn, C., Rigoulay, C., Chabelskaya, S., Sharma, C.M., Marchais, A., Skorski, P., Borezée-
Durant, E., Barbet, R., Jacquet, E., Jacq, A., Gautheret, D., Felden, B., Vogel, J., Bouloc, P.,
2010. Experimental discovery of small RNAs in Staphylococcus aureus reveals a
riboregulator of central metabolism. Nucleic Acids Res. 38, 6620–6636.
8. Cong, L., Ran, F.A., Cox, D., Lin, S., Barretto, R., Hsu, N.H.P.D., Wu, X., Jiang, W.,
Marraffini, L.A., Zhang, F., 2013. Multiplex genome engineering using CRISPR/Cas systems.
Science.
9. Davis, B.M., Quinones, M., Pratt, J., Ding, Y., Waldor, M.K., 2005. Characterization of the
small untranslated RNA RyhB and its regulon in Vibrio cholerae. J. Bacteriol. 187, 4005–4014.
10. Englesberg, E., Anderson, R.L., Weinberg, R., Lee, N., Hoffee, P., Huttenhauer, G., Boyer,
H., 1962. L-arabinose-sensitive, l-ribulose 5-phosphate 4-epimerase-deficient mutants of
Escherichia coli. J. Bacteriol. 84, 137.
11. Geissmann, T., Possedko, M., Huntzinger, E., Fechter, P., Ehresmann, C., Romby, P.,
2006. Regulatory RNAs as mediators of virulence gene expression in bacteria. Handb. Exp.
Pharmacol., 9–43.
12. Giuliodori, A.M., Di Pietro, F., Marzi, S., Masquida, B., Wagner, R., Romby, P., Gualerzi,
C.O., Pon, C.L., 2010. The cspA mRNA is a thermosensor that modulates translation of the
cold-shock protein CspA. Mol. Cell 37, 21–33.
13. Hale, C., Kleppe, K., Terns, R.M., Terns, M.P., 2008. Prokaryotic silencing (psi)RNAs in
Pyrococcus furiosus. RNA 14, 2572–2579.
14. Hoe, C.-H., Raabe, C.A., Rozhdestvensky, T. S., Tang, T.-H., 2013. Bacterial sRNAs:
Regulation in stress. Int J Med Microbiol. 303, 217-229.
15. Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J.A., Charpentier, E., 2012. A
programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science
337, 816–821.
16. Johansson, J., Mandin, P., Renzoni, A., Chiaruttini, C., Springer, M., Cossart, P., 2002. An
RNA thermosensor controls expression of virulence genes in Listeria monocytogenes. Cell
110, 551–561.
17. Jin, Y., Watt, R.M., Danchin, A., Huang, J., 2009. Small noncoding RNA GcvB is a novel
regulator of acid resistance in Escherichia coli. BMC Genomics 10, 165.
18. Kortmann, J., Narberhaus, F., 2012. Bacterial RNA thermometers: molecular zippers and
switches. Nat. Rev. Microbiol. 10, 255–265.
19. Landt, S.G., Lesley, J.A., Britos, L., Shapiro, L., 2010. CrfA, a small noncoding RNA
regulator of adaptation to carbon starvation in Caulobacter crescentus. J. Bacteriol. 192,
4763–4775.
20. Levine, E., Hwa, T., 2008. Small RNAs establish gene expression thresholds. Curr. Opin.
Microbiol. 11, 574–579.
21. Makarova, K.S., Haft, D.H., Barrangou, R., Brouns, S.J.J., Charpentier, E., Horvath, P.,
Moineau, S., Mojica, F.J.M., Wolf, Y.I., Yakunin, A.F., Van der Oost, J., Koonin, E.V., 2011.
Evolution and classification of the CRISPR-Cas systems. Nat. Rev. Microbiol. 9, 467–477.
22. Maki, K., Morita, T., Otaka, H., Aiba, H., 2010. A minimal base-pairing region of a bacterial
small RNA SgrS required for translational repression of ptsG mRNA. Mol. Microbiol. 76, 782–
792.
23. Mali, P., Yang, L., Esvelt, K.M., Aach, J., Guell, M., Dicario, J.E., Norville, J.E., Church, G.M.,
2013. RNA-guided human genome engineering via Cas9. Science.
24. Massé, E., Vanderpool, C.K., Gottesman, S., 2005. Effect of RyhB small RNA on global iron
use in Escherichia coli. J. Bacteriol. 187, 6962–6971.
25. Mey, A.R., Craig, S.A., Payne, S.M., 2005. Characterization of Vibrio cholerae RyhB: the
RyhB regulon and role of ryhB in biofilm formation. Infect. Immun. 73, 5706–5719.
26. Morita, T., Maki, K., Aiba, H., 2005. RNase E-based ribonucleoprotein complexes:
mechanical basis of mRNA destabilization mediated by bacterial noncoding RNAs. Genes
Dev. 19, 2176–2186.
27. Mulhbacher, J., Brouillette, E., Allard, M., Fortier, L.-C., Malouin, F., Lafontaine, D.A., 2010.
Novel riboswitch ligand analogs as selective inhibitors of guanine-related metabolic
pathways. PLoS Pathog. 6, e1000865.
28. Murphy, E.R., Payne, S.M., 2007. RyhB, an iron-responsive small RNA molecule, regulates
Shigella dysenteriae virulence. Infect. Immun. 75, 3470–3477.
29. Møller, T., Franch, T., Højrup, P., Keene, D.R., Bächinger, H.P., Brennan, R.G., Valentin-
Hansen, P., 2002. Hfq: a bacterial Sm-like protein that mediates RNA–RNA interaction. Mol.
Cell 9, 23–30.
30. Nechooshtan, G., Elgrably-weiss, M., Sheaffer, A., Westhof, E., Altuvia, S., 2009. A pH-
responsive riboregulator. Gene Dev. 23, 2650–2662.
31. Opdyke, J.A., Fozo, E.M., Hemm, M.R., Storz, G., 2011. RNase III participates in GadY
dependent cleavage of the gadX–gadW mRNA. J. Mol. Biol. 406, 29–43.
32. Opdyke, J.A., Kang, J., Storz, G., 2004. GadY, a small-RNA regulator of acid response
genes in Escherichia coli. J. Bacteriol. 186, 6698–6705.
33. Papenfort, K., Bouvier, M., Mika, F., Sharma, C.M., Vogel, J., 2010. Evidence for an
autonomous 5ꠗ target recognition domain in an Hfq-associated small RNA. Proc. Natl.
Acad. Sci. U. S. A. 107, 20435–20440.
34. Perez-Rodriguez, R., Haitjema, C., Huang, Q., Nam, K.H., Bernardis, S., Ke, A., DeLisa,
M.P., 2011. Envelope stress is a trigger of CRISPR RNA-mediated DNA silencing in
Escherichia coli. Mol. Microbiol. 79, 584–599.
35. Pernestig, A., Georgellis, D., Romeo, T., Suzuki, K., Tomenius, H., Normark, S., Melefors,
Ö., 2003. The Escherichia coli BarA-UvrY two-component system is needed for efficient
switching between glycolytic and gluconeogenic carbon sources. J. Bacteriol. 185, 843–853.
36. Pichon, C., Felden, B., 2008. Small RNA gene identification and mRNA target predictions in
bacteria. Bioinformatics (Oxford, England) 24, 2807–2813.
37. Pomposiello, P.J., Demple, B., 2001. Redox-operated genetic switches: the SoxR and OxyR
transcription factors. Trends Biotechnol. 19, 109–114.
38. Pul, U., Wurm, R., Arslan, Z., Geissen, R., Hofmann, N., Wagner, R., 2010. Identification and
characterization of E. coli CRISPR-cas promoters and their silencing by H-NS. Mol.
Microbiol. 75, 1495–1512.
39. Raabe, C.A., Hoe, C.-H., Randau, G., Brosius, J., Tang, T.-H., Rozhdestvensky, T.S., 2011.
The rocks and shallows of deep RNA sequencing: Examples in the Vibrio cholerae RNome.
RNA 17, 1357–1366.
40. Rasmussen, A.A., Eriksen, M., Gilany, K., Udesen, C., Franch, T., Petersen, C., Valentin-
Hansen, P., 2005. Regulation of ompA mRNA stability: the role of a small regulatory RNA in
growth phase-dependent control. Mol. Microbiol. 58, 1421–1429.
41. Repoila, F., Gottesman, S., 2001. Signal transduction cascade for regulation of RpoS:
temperature regulation of DsrA. J. Bacteriol. 183, 4012–4023.
42. Richards, G.R., Vanderpool, C.K., 2011. Molecular call and response: The physiology of
bacterial small RNAs. Biochim. Biophys. Acta 1809, 525–531.
43. Tang, T.-H., Bachellerie, J.-P., Rozhdestvensky, T., Bortolin, M.-L., Huber, H., Drungowski,
M., Elge, T., Brosius, J., Hüttenhofer, A., 2002. Identification of 86 candidates for small non-
messenger RNAs from the archaeon Archaeoglobus fulgidus. Proc. Natl. Acad. Sci. U. S.
A. 99, 7536–7541.
44. Urbanowski, M.L., Stauffer, L.T., Stauffer, G.V., 2000. The gcvB gene encodes a small
untranslated RNA involved in expression of the dipeptide and oligopeptide transport
systems in Escherichia coli. Mol. Microbiol. 37, 856–868.
45. Veˇcerek, B., Moll, I., Bläsi, U., 2007. Control of Fur synthesis by the non-coding RNA RyhB
and iron-responsive decoding. EMBO J. 26, 965–975.
46. Wadler, C.S., Vanderpool, C.K., 2007. A dual function for a bacterial small RNA: SgrS
performs base pairing-dependent regulation and encodes a functional polypeptide. Proc.
Natl. Acad. Sci. U. S. A. 104, 20454–20459.
47. Waldminghaus, T., Gaubig, L.C., Klinkert, B., Narberhaus, F., 2009. The Escherichia coli
ibpA thermometer is comprised of stable and unstable structural elements. RNA Biol. 6,
455–463.
48. Westra, E.R., Swarts, D.C., Staals, R.H.J., Jore, M.M., Brouns, S.J.J., Van der Oost, J.,
2012. The CRISPRs, they are a-changin’: how prokaryotes generate adaptive immunity.
Annu. Rev. Genet. 46, 311–339.





