합성생물학 부품 개량을 위한 단백질공학의 연구동향
건국대학교 이정걸
1. 개요
효소는 전 세계적으로 대대적인 연구개발 및 산업화가 추진되고 있는 White Biotechnology 분야의 핵심으로 부각되었으며, 그로인해 세계적 화학 및 제약, 생명공학 기업들이 각각의 목적에 맞는 효소의 개발에 집중적으로 투자하고 있으며, 그 시장은 급속도로 팽창하고 있다 (Fig. 1).
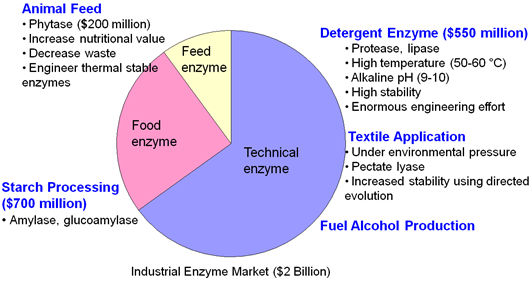
Fig. 1. 산업용 효소 시장의 현황. Animal feed, starch processing, detergent, textile, biofuel 등 등 다양한 분야에서 산업용 효소가 사용되고 있으며, 대규모 시장을 형성하고 있음 (Cherry and Fidantsef 2003, Walsh 2006).
과거에는 자연계에 존재하고 있는 다양한 미생물들이 효소의 다양성을 확보하는 중요한 자원이었다. 즉, 목적에 맞게 효소를 설계하기보다는 목적에 부합되는 효소를 방대한 생태계에서 탐색하는 bioprospecting이 주된 효소 발견 방법이었다. 그러나, 자연계에서 발굴한 유전자원을 실제 산업 현장에 바로 이용하기에는 여러 가지 제약이 따른다. 즉, 분리한 효소의 비활성, pH 및 열 안정성, 기질특이성, 리간드와의 친화성 등이 실제 의약용이나 산업적으로 사용하기에는 많은 한계점을 나타낸다. 1990년대부터 이를 해결하기 위해 목적에 맞는 특성이나 새로운 기능을 지닌 단백질을 설계하는 방향으로 paradigm shift가 일어나 효소 활성, 안정성, 기질 특이성 등을 원하는 방향으로 설계하여 맞춤형 효소를 제작하는 단백질공학에 의해 주로 연구가 진행되고 있다. 고전적 효소 설계로는 단백질 수준에서 chemical에 의해 특정 아미노산 잔기를 변형하는 화학적 변형법인 atom replacement, segment reassembly 등이 사용되었다. 그러난 단지 몇몇 잔기만 chemical에 의해 변형될 수 있고, 기법 자체가 복잡하고, time-consuming 하여 널리 사용되지 못하였다. 2000년대 이후로 유전자 관련 기술 및 구조생물학이 급속히 발달하면서 유용한 새로운 생체 분자를 만들기 위한 많은 접근 방법이 개발되었다. 이 접근 방법들은 크게 3가지로 분류될 수 있다: directed evolution, rational design 및 semi-rational design 또는 designed evolution과 같은 하이브리드 시스템. 이들 기법을 통해 자연계에 존재하지 않았던 우수한 촉매들이 제작되었으며, 본 보고에서는 단백질 공학을 위한 이들 진화적 접근 방법들의 새로운 동향과 몇몇 흥미로운 응용에 대해서 논의한다.
2. 단백질 공학 연구 기법 및 동향
효소들은 매우 다재다능하고 능숙한 촉매이다. 수백만 년 동안의 다윈 진화에 의해 최적화된 이들은 화학 반응을 크게 가속시킬 수 있고 높은 기질 특이성뿐 아니라 정교한 거울상 선택성과 입체 선택성을 확보할 수 있다. 이러한 성능 특성들이 생체 촉매를 실험실 및 상업 공정에서 비대칭적 합성을 위한 매력적인 후보로 만든다. 그러나 자연에서의 효소의 기능과 과학자들 및 기술자들이 생각하는 생체 외 용도를 위한 구체적 요건들 사이에 종종 유의한 차이가 있다. 이러한 차이의 극복을 위해 다음과 같은 효소 조작이 표적 단백질의 촉매 특성, 생물 물리학적 및 분자 인식 특성의 조절을 위해 선택되는 전략이 되고 있다.
1) Directed evolution
Directed evolution은 구조에 대한 깊이 있는 이해 없이 초고속 스크리닝 시스템에 기반하여 비교적 빠르게 효소를 개량할 수 있게 한다. Directed evolution은 대표적으로 두 가지 접근 방법에 의해 진행된다; i. 관련된 서열의 조합을 무작위로 재조합 (예. 유전자 shuffling), ii. 단일한 단백질 서열에 무작위의 변화 도입 (예. error prone PCR). 이러한 directed evolution의 장점은 구조 정보가 필요 없으며 활성 부위에서 먼 예상치 않은 위치에서의 변이가 도입될 수 있다는 것이다. 즉, Directed evolution은 실험실 수준에서 자연 진화 과정을 모사하는 기법으로 반복적인 단백질 변이체 library 구축과 초고속 스크리닝법에 의한 목적 단백질의 스크리닝/selection으로 구성되어 있다. 또한 목적 효소의 구조 및 기능에 대한 자세한 정보를 요구하지 않음에도 불구하고, 확보된 변이체의 규명을 통해 단백질 구조와 기능에 대한 정보를 얻을 수 있는 top-down approach에 해당한다. 그러나 한 차례의 돌연변이 및 스크리닝에 의해 확보되는 변화는 대개 그 효과가 작다. 그로인해 수차례의 추가적인 진화가 적용되어야 하며, 많은 수의 변종이 선별되어야 하므로 많은 시간과 노동력의 소모를 요구하는 경우가 대부분이다. 그와 더불어 신뢰성이 높고 대용량 처리가 가능한 초고속 분석법을 필수적으로 요구한다. 또한 단순한 효소의 기능 개선 목적이 아니라 신규 기질특이성, 초고활성 등과 같은 새로운 기능을 갖는 효소를 얻기 위해서는 다중 돌연변이를 요구하는데, 현 기술로는 다중 돌연변이가 도입된 개체의 스크리닝이 용이하지 않다는 문제점이 있다. 즉, Fig. 2와 같이 quadruple mutant만 되더라도 현재의 기술로는 필요한 library의 규모를 확보할 수 없기 때문에 정상적인 스크리닝이 불가능하다.
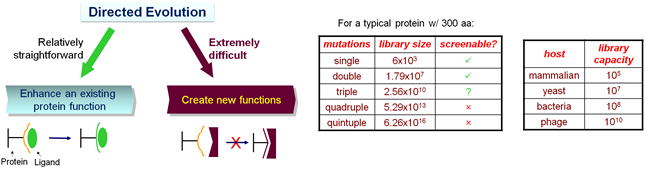
Fig. 2. Limitation of directed evolution. 다중 돌연변이를 스크리닝 하기에는 라이브러리 규모에 한계가 있으며, 그로인해 신기능을 갖는 효소의 탐색이 매우 어려움.
2) Rational design
단백질 기능은 보통 3차원 구조와 긴밀하게 연계되어서 고분자에서의 하나 또는 그 이상의 아미노산의 치환을 단지 서열 맥락에서의 함수만이 아닌 구조적 위상관계에서의 함수로도 만든다. PDB 단백질 구조 숫자 증가와 상동관계 모델링의 진보는 단백질 공학자들이 활성 부위와 영역 인터페이스 또는 경첩 영역에 가까운 핵심 잔기를 보다 효과적으로 찾아내는데 가치 있는 도움을 제공하여 뛰어난 라이브러리 설계를 가능하게 한다. Rational design에서는 생화학적 자료 및 단백질 구조 자료에 근거하여 단백질 특성 개량에 필요한 돌연변이 위치를 제안하며 돌연변이는 위치 특이성 돌연변이 유발에 의해 도입된다. Rational design 접근 방법의 장점들 중 하나는 유익한 돌연변이의 가능성이 증가되고 라이브러리 크기가 유의하게 감소되므로 라이브러리의 선별에 더 적은 노력과 시간이 쓰인다는 것이다. 이것은 처리량이 큰 분석법을 이용할 수 있을 경우에 특히 유리하다. 최근 rational design에 많이 적용되고 있는 computational 단백질 설계의 분야는 두 가지 목표를 추구한다. 하나는 실제적인 것으로 목적 기능이 강화된 단백질 산물을 확보하는 것이고, 두 번째 목표는 공학적 목표보다는 과학적 목표로서, 단백질의 특성에 영향을 미치는 원리에 대한 정량적 설명을 추구하는 것이다. 이와 같이 rational design은 단백질의 구조정보에 근거하여 목적 단백질의 특정 위치에 rational alteration을 가하여 단백질의 기능에 원하는 변화를 추구하는 방법이다. 따라서 이 방법은 효소 구조, 기능, 촉매 기작 등 의 자세한 정보를 요구하는 bottom-up approach에 해당하는 기법이다. 그러나 구조생물학 기법이 비교적 최근에 보편화되었으며, 그로인해 다양한 효소의 구조가 충분히 밝혀져 있지 않기 때문에 구조에 근거한 단백질공학 연구, 즉 rational design에 의한 연구가 모든 효소를 대상으로 체계적으로 수행되는 데 한계가 있다.
3) Semi-rational design 또는 designed evolution
Semi-rational design은 합리적 및 무작위 단백질 설계의 장점들을 결합하여 생화학적, 그리고/또는 구조적 자료에서 도출된 지식에 근거하여 더 작고 스마트한 라이브러리를 생성한다 (Lutz S and Bornscheuer U, 2009). 한 예는 구조적 자료에서 도출된 정보를 사용하여 관심 부위(예, 활성 부위)에 있는 아미노산들을 확인하고 그 아미노산들이 무작위로 또는 부위 포화 돌연변이 유발에 의해서 하나씩 또는 함께 돌연변이가 되는 CASTing (combinatorial active site saturation test)이다. 표적 위치에서 돌연변이의 무작위 조합 또는 상관 돌연변이는 단일 부위에 특이적인 돌연변이 유발에서 누락되었을 수 있는 시너지 효과를 가져올 수 있다. 그러나, 이 조합적 접근 방법들은 라이브러리 크기를 크게 증가시키므로 최근 몇 년간 가상 라이브러리를 검사하여 단백질 주름에 불리할 것으로 예상되는 돌연변이를 배제함으로써 라이브러리 크기를 줄이도록 돕는 여러 가지 computational 방법들이 개발되었다. 다음은 근래 사용되고 있는 다양한 semi-rational design 또는 designed evolution 기법을 소개한다.
- 서열기반 효소 재설계
단백질 서열 space에서 기능성의 ‘islands’를 보다 효과적으로 찾고 확인하는 흔한 전략은 진화 정보를 사용하는 것이었다. 다중 서열 정렬 (MSAs) 및 계통 발생 분석이 아미노산 보존과 상동 단백질 서열 및 구조 집단들 간 조상의 관계를 탐색하는 표준 도구가 되었다. 이 통계가 큰 자연 서열 풀에서 도출되었든 실험실에서 중성 흐름 실험을 통해서 도출되었든, 이 자료는 기능적 핫 스팟을 확인하고 국소 아미노산 변동성 평가 및 합의 설계를 위해 단백질 공학자에게 중요하다. 이 목적을 위한 많은 수의 자료 분석 소프트웨어 중에서 (Damborsky J and Brezovsky J 2009, Pei J 2008), 2가지의 새로운 기반 전산 도구가 주목할 만하다. 즉, HotSpot Wizard 서버 (http://loschmidt.chemi.muni.cz/hotspotwizard)는 광범위한 서열 및 구조 데이터베이스 검색에서 얻은 정보를 기능적 자료와 결합하여 표적 단백질에 대한 돌연변이 성 맵을 생성한다 (Pavelka A 2009). 이 시스템의 성능이 Rhodococcus rhodochrous로부터 유래한 haloalkane dehalogenase (DhaA) 제작의 일부로 제시되었다 (Pavlova et al. 2009). 이 자료들은 상관 돌연변이 및 보존과 같은 진화 특징에 대한 일련의 내장된 필터를 통하여 효과적으로 검색될 수 있다. 이 데이터베이스의 단백질 공학에 대한 잠재적인 편익이 활성도 및 기질 특이성의 변화뿐 아니라 (Kuipers et al. 2009, Joosten et al. 2008), 거울상 선택성의 개선 (Jochens et al. 2010)을 위한 모델 효소를 탐사하는 3건의 연구에서 제시되었다. 서열기반 정보와 전산 모델링 도구의 결합을 통해서 효소 재설계의 추가적인 발전이 가능하다. 무작위 돌연변이 유발과 DNA shuffling 라이브러리에서 이전에 확인된 DhaA 변종에서의 중등도의 기능적 개선을 설명하기 위해서 분자 역학 시뮬레이션을 수행하는 도중에, Damborsky와 그의 동료들이 유익한 돌연변이들이 활성 부위에 직접 영향을 미치지 않고 효소의 2개의 접근 터널의 배좌 변화를 통해서 촉매 성능을 개선한다는 것을 발견했다 (Banas et al. 2006). HotWizard에 의해 유도된 특정한 치환이 위치들 중 2개에 도입되었고 향상된 dehalogenase 활성에 대한 나머지 3개 아미노산 잔기의 영향이 작은 부위포화 돌연변이 유발 라이브러리에 의해서 쉽게 평가될 수 있었다. 이 전략은 유의하게 더 높은 활성이 있는 효소 변종을 분리하는데 유용할 뿐 아니라 가장 유익한 아미노산 치환을 확인하는데 매우 중요한 것으로 확인되었다.
- 구조 기반 효소 재설계
활성 부위에 가까운 잔기 뿐만아니라 멀리 떨어진 잔기의 돌연변이 유발에 근거한 혁신적인 구조 기반 효소 재설계가 다음과 같이 수행되었다. 즉, Reetz 와 그의 동료들은 활성 부위에 가까운 잔기의 돌연변이 유발을 넘어서 소위 유도된 알로스테릭 효과를 통해서 원위치 돌연변이의 잠재적인 기능적 편익을 탐구했다 (Wu et al. 2010). 이들은 Thermobifida fusca로부터 유래한 열 안정적인 Bayer-Villiger monooxygenase의 FAD 와 NADP 결합 영역의 인터페이스에 초점을 두었다 (Malito et al. 2004, Fraaije et al. 2005, 66:393-400). 구조 분석이 포화 돌연변이가 유발된 2개 영역의 경첩 영역 가까이 위치한 나선 부분에서 2개의 아미노산 위치를 확인했다. 이들 위치들에서의 치환이 촉매 활성을 증가시켰을 뿐 아니라 수용 가능한 기질의 범위를 유의하게 넓혔다. 또 다른 성공적인 생체 촉매 재설계 중의 하나는 JanuviaTM (Sitagliptin phosphate) 공정을 위한 ATA-117 D-아미노산 아미노 전이효소의 개발이다 (Savile et al. 2010). Savile 등은 transition state 도킹을 통해서 semi-rational design을 사용하여 목적 기질에 대한 활성을 개선하여 표적 기질을 아미노화 할 수 있는 효소를 만들어 냈다. ProSAR을 통한 몇 차례의 추가적인 표적 및 무작위 돌연변이 유발 후에 산업적 산출 및 운영 조건을 위해 설계된 효소가 개발되었다. 궁극적으로 최종 효소는 활성 부위 주위 및 이량체 인터페이스 모두에 27개의 돌연변이를 함유하였다. 또한 Codexis는 성공적으로 semi-rational design을 적용하여 몇 가지 케토 환원 효소의 활성과 거울상 선택성을 향상시켜서 산업적으로 적절한 기질을 제조하였으며, 이 과정 중에 결과로 나오는 촉매도 원하는 산업적 반응 조건을 향해서 최적화 되었다 (Ma et al. 2010, Liang et al. 2010, Huisman et al. 2010).
- Computational 효소 재설계
Computational 단백질 설계 알고리즘의 진보는 가상 환경에서의 모델링을 생체 촉매의 조절을 위한 매우 유망한 전략으로 만들었다. 벤치에서의 서열 변경과 조합적 라이브러리 준비에 대한 안내로 진화적 정보에 의존하는 대신 computational 방법은 회전 이성질체 라이브러리와 백본 재편성을 사용하는 것을 통해서 전반적 단백질 구조에 대한 아미노산 변이의 에너지학을 효과적으로 추산할 수 있으므로 실험에 의한 단백질 공학을 단지 몇 개의 합리적 설계의 평가로 축소한다. 효소의 기질 특이성을 개조할 수 있는 이 예측 틀의 능력은 두 그룹에 의해 최근에 제시되었다. Chen 등은 자신들의 K* 알고리즘을 사용하여 비 리보솜 펩티드 합성 효소 gramicidin S synthetase A의 페닐알리닌 아데닐화 영역의 기질 특이성을 변경했다 (Chen et al. 2009). 새로운 기질로 Leu 이나 전하를 띠는 아미노산 Arg, Lys, Glu 또는 Asp 중 하나를 표적으로 하는 최초의 시뮬레이션은 결합 포켓의 안쪽에 깔려 있는 7개의 잔기에 집중했다 (Fig. 3A). Leu에 대해서는 7개의 위치들 중 2개에서의 돌연변이가 kcat/KM을 20배 더 높게 하고, Phe에 대한 촉매 효율을 30배 감소시켜서 아데닐화 영역의 기질 특이성을 효과적으로 변경하였다. 이후에, 단백질 구조의 다른 부분에서의 돌연변이가 탐구되었으며, 그를 통해 효소 활성을 현저히 높인 결과를 얻었다. 또한 computational 효소 재설계가 개별적인 아미노산 치환에 제한될 필요가 없고 루프 영역 전체의 교체에 적용될 수 있다는 훌륭한 설명이 Murphy 등에 의해 보고되었다. (Murphy et al. 2009). 박테리아 시토신 탈아미노 효소에서 약간의 영감을 얻은 저자들은 자살 유전자 요법에서의 잠재적인 적용을 위해서 시토신 특이성 인간 구아닌 탈아미노 효소 (hGDA) 변종을 생성하는 것을 목표로 hGDA의 활성 부위 루프 영역의 리모델링을 위해서 Rosetta 설계 알고리즘을 사용하였다. 루프 길이와 아미노산 구성의 동시적 변이 가상 환경에서의 모델링을 통해 새로운 서열이 확인되었는데, 그것은 ammelide (시토신에 대해 고안된 중간체)에 대한 활성을 100 배 높이고 구아닌 탈아미노화를 네 자릿수 배 만큼 감소시켰다 (Fig. 3B). 또 다른 성공적인 결과는 Baker 실험실에 의한 활성 부위의 computational 설계를 포함한다. 그들은 Rosetta 알고리즘을 사용하여 Kemp 배제 효소 (Khersonsky et al. 2010, Alexandrova et al. 2008)와 Diels-Alderase (Siegel et al. 2010)를 포함하는 새로운 생체 촉매를 성공적으로 설계했다. 이러한 촉매들은 구조–기능 관계에 대한 현재까지의 이해에 이의를 제기하며 자연에서 발견되지 않는 촉매를 설계하는 단백질공학자의 능력을 유의하게 향상시킨다.
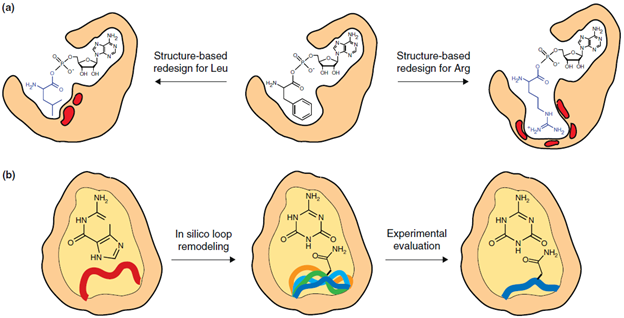
Fig. 3. Computational 효소 재설계. (A) 돌연변이들 (붉은색)은 작은 소수성 (Leu) 또는 전하를 띤 측쇄를 가진 아미노산(Arg)에 대한 phenylalanine adenylation domain의 기질특이성을 변화시킴. (B) 인간 guanosine deaminase 활성부위 내 loop 영역(붉은색)의 engineering은 ammelide-specific deaminase를 제조함.
3. 고찰
가상환경 또는 구조 기반 방법의 활용이 효과적 단백질 개량을 위한 예측을 가능하게 하며 이것은 시스템의 복잡성을 유의하게 줄일 수 있고 더 작고 기능적으로 풍부한 라이브러리의 제작을 가능하게 한다. Directed evolution이 아직도 이 집중된 라이브러리에서 성능이 좋은 후보들을 확인하는 가장 효과적인 실험 전략이지만 이미 새로운 설계 전략과 기술적 진보가 전통적인 프로토콜로부터 벗어나기 시작한 것을 의미한다. 즉, 최근 semi-rational design 또는 designed evolution 에서의 방법론적 진보는 단백질 연구자들에게 생체 촉매를 조작하는 강력하고 효과적인 새로운 전략을 제공하고 있다. 이와같이 구조 기반 rational design 또는 computational 단백질 설계 기법이 개별 기술로서 단백질 공학자들이 생체 고분자를 조작하는 방법을 근본적으로 변화시켰지만 단백질 공학을 위한 방법으로서 directed evolution을 완벽하게 대체하지는 못할 것으로 전망되고 있다. 그 대신에 단백질 공학 연구자들은 각 전략의 보완성을 인식하고 효소의 보다 효과적인 조작을 위해서 이들의 통합 또는 융합을 수용해야 할 것으로 사료된다. 궁극적으로는 대규모 선별 장비에 덜 의존하는 것과 더 높은 소모된 자원 단위 당 개선율 또는 적중률이 단백질공학 연구의 향후 방향을 유도할 것으로 전망된다.
4. 참고문헌
Alexandrova AN, Rothlisberger D, Baker D, Jorgensen WL: Catalytic mechanism and performance of computationally designed enzymes for kemp elimination. J Am Chem Soc 2008, 130:15907-15915.
Banas P, Otyepka M, Jerabek P, Petrek M, Damborsky J: Mechanism of enhanced conversion of 1,2,3-trichloropropane by mutant haloalkane dehalogenase revealed by molecular modeling. J. Comput Aided Mol Des 2006, 20:375-383.
Chen CY, Georgiev I, Anderson AC, Donald BR: Computational structure-based redesign of enzyme activity. Proc Natl Acad Sci U S A 2009, 106:3764-3769.
Damborsky J, Brezovsky J: Computational tools for designing and engineering biocatalysts. Curr Opin Chem Biol 2009, 13:26-34.
Fraaije MW, Wu J, Heuts DP, van Hellemond EW, Spelberg JH, Janssen DB: Discovery of a thermostable Baeyer-Villiger monooxygenase by genome mining. Appl Microbiol Biotechnol 2005, 66:393-400.
Gary Walsh. Biopharmaceutical benchmarks. The rate of biopharmaceutical approvals has leveled off, but some milestones bode well for the future. Nature Biotechnology 24, 769 - 776 (2006)
Huisman GW, Liang J, Krebber A: Practical chiral alcohol manufacture using ketoreductases. Curr Opin Chem Biol 2010, 14:122-129
Jochens H, Bornscheuer UT: Natural diversity to guide focused directed evolution. ChemBioChem 2010, 11: 1861–1866. Validation of higher functional content in designer enzyme libraries derived from consensus sequence over random or non-consensus sequence pools.
Joel R Cherry and Ana L Fidantsef. Directed evolution of industrial enzymes: an update. Current Opinion in Biotechnology 2003, 14:438–443.
Joosten HJ, Han Y, Niu W, Vervoort J, Dunaway-Mariano D, Schaap PJ: Identification of fungal oxaloacetate hydrolyase within the isocitrate lyase/PEP mutase enzyme superfamily using a sequence marker-based method. Proteins 2008, 70:157-166.
Khersonsky O, Rothlisberger D, Dym O, Albeck S, Jackson CJ, Baker D, Tawfik DS: Evolutionary optimization of computationally designed enzymes: kemp eliminases of the KE07 series. J Mol Biol 2010, 396:1025-1042.
Kuipers RK, Joosten HJ, Verwiel E, Paans S, Akerboom J, van der Oost J, Leferink NG, van Berkel WJ, Vriend G, Schaap PJ: Correlated mutation analyses on super-family alignments reveal functionally important residues. Proteins 2009, 76:608-616.
Liang J, Mundorff E, Voladri R, Jenne S, Gilson L, Conway A, Krebber A, Wong J, Huisman G, Truesdell S et al.: Highly enantioselective reduction of a small heterocyclic ketone: biocatalytic reduction of tetrahydrothiophene-3-one to the corresponding (R)-alcohol. Org Process Res Dev 2010, 14:188-192.
Lutz S, Bornscheuer U (Eds): The Protein Engineering Handbook. Weinheim: Wiley-VCH; 2009.
Ma SK, Gruber J, Davis C, Newman L, Gray D, Wang A, Grate J, Huisman GW, Sheldon RA: A green-by-design biocatalytic process for atorvastatin intermediate. Green Chem 2010, 12:81-86.
Malito E, Alfieri A, Fraaije MW, Mattevi A: Crystal structure of a Baeyer-Villiger monooxygenase. Proc Natl Acad Sci U S A 2004, 101:13157-13162.
Murphy PM, Bolduc JM, Gallaher JL, Stoddard BL, Baker D: Alteration of enzyme specificity by computational loop remodeling and design. Proc Natl Acad Sci U S A 2009, 106:9215-9220.
Pavelka A, Chovancova E, Damborsky J: HotSpot Wizard: a web server for identification of hot spots in protein engineering. Nucleic Acids Res 2009, 37:W376-383.
Pavlova M, Klvana M, Prokop Z, Chaloupkova R, Banas P, Otyepka M, Wade RC, Tsuda M, Nagata Y, Damborsky J: Redesigning dehalogenase access tunnels as a strategy for degrading an anthropogenic substrate. Nat Chem Biol 2009, 5:727-733.
Pei J: Multiple protein sequence alignment. Curr Opin Struct Biol 2008, 18:382-386.
Savile CK, Janey JM, Mundorff EC, Moore JC, Tam S, Jarvis WR, Colbeck JC, Krebber A, Fleitz FJ, Brands J et al.: Biocatalytic asymmetric synthesis of chiral amines from ketones applied to sitagliptin manufacture. Science 2010, 329:305-309.
Siegel JB, Zanghellini A, Lovick HM, Kiss G, Lambert AR, Clair JLS, Gallaher JL, Hilvert D, Gelb MH, Stoddard BL et al.: Computational design of an enzyme catalyst for a stereoselective bimolecular Diels-Alder reaction. Science 2010, 329:309-313.
Wu S, Acevedo JP, Reetz MT: Induced allostery in the directed evolution of an enantioselective Baeyer-Villiger monooxygenase. Proc Natl Acad Sci U S A 2010, 107:2775-2780.





