Genome engineering을 통한 지능형 인공미생물 개발
이성국
울산과학기술대학교
최근 몇 년간 미국의 CNN1 , 영국의 BBC2 와 국영방송3 등 여러 권위 있는 미디어매체를 통해 합성생물학의 잠재성이 보도되었다. 현재 가장 뜨거운 관심이 모아지고 있는 분야인 합성생물학은 기존 몇 개의 유전자를 대상으로 하는 유전공학적 실험 방법이나 기술들에 국한된 것이 아닌, 특정 유전체의 전반적인 이해를 바탕으로 한다. 특히 새로운 생체대사경로의 제작뿐 아니라 나아가 유전체 설계 및 인공 유전체 합성 등을 가능하게 하여 지금까지 다루어진 적이 없는 새로운 분야를 다루는 학문으로 여겨지고 있다.
지난 반세기 동안 생물학분야의 한 줄기에 불과했던 유전공학은 급격한 성장을 바탕으로 생물체를 기반한 목적 대사산물의 생산량을 상승시키거나 다른 생물체B로부터 생물체A에는 없는 물질을 생산하게 하는 등의 일들을 실현 가능하게 하였다. 지난 고전적 생물학적 방법을 통한 생물체의 유전자 변형은 몇 가지가 있어 왔으나, 초기의 연구자들은 물리•화학적인 방법을 사용하여 유전자에 돌연변이를 도입하였다.4 5 비록 유전자에 돌연변이를 도입시켜 새로운 유전자형(genotype)은 얻었지만 원하는 표현형(phenotype)을 제작하기는 쉽지 않은 일이었다. Biomolecule의 생산 및 분해 그리고 그들의 상호작용을 이해하기 위한 방대한 양의 지식의 결여와 함께 모델링을 위한 computational source의 약한 기반은 생명체의 이해를 바탕으로 공학적인 개념을 도입하는고자 하는 합성생물학의 발전의 큰 장애물이었다. 하지만 무엇보다도 유전자 서열의 변화를 위한 분자생물학적인 기술의 부재는 원하는 표현형을 얻기 위해 계획적 변경(rational alteration)보다 진화적 최적화(evolutionary optimization)에 의존하도록 하였다. 하지만 지난 몇 년간 수천 종의 생명체 유전자 서열분석을 통해서 생물 정보량이 기하급수적으로 증가하고 그와 동시에 계산적 생물학적 도구(computational tool)의 발전으로 생명시스템을 이해하게 되고 이를 바탕으로 새로운 생명 시스템 및 생명체를 제작하고자 하는 합성생물학이 급속적으로 발전하게 된다. 실질적으로 인공 생명 시스템을 제작하기 위해서는 다양한 지놈엔지니어링(genome engineering) 기술의 도움이 꼭 필요하다.
최근 개발되고 있는 지놈엔지니어링 기법들은 합리적인 유전체의 이해를 바탕으로 한 유전자 지도의 설계와 합성을 통해 대사산물의 생산량을 증가시킬 수 있는 전략임과 동시에 다양한 지능형 인공 생명체를 제작할 수 있는 기반을 만들고 있다. 이에 최근에 개발된 인공 미생물 제작 기술인 지놈엔지니어링 기술들과 합성된 새로운 생물체들 중 우량균주의 선별을 효과적으로 도울 수 있는 bioMEMS 기술에 대해서 간략히 설명하고자 한다.
가. 지놈 엔지니어링 기술들
1. Transcriptional activator-like effector genome editing
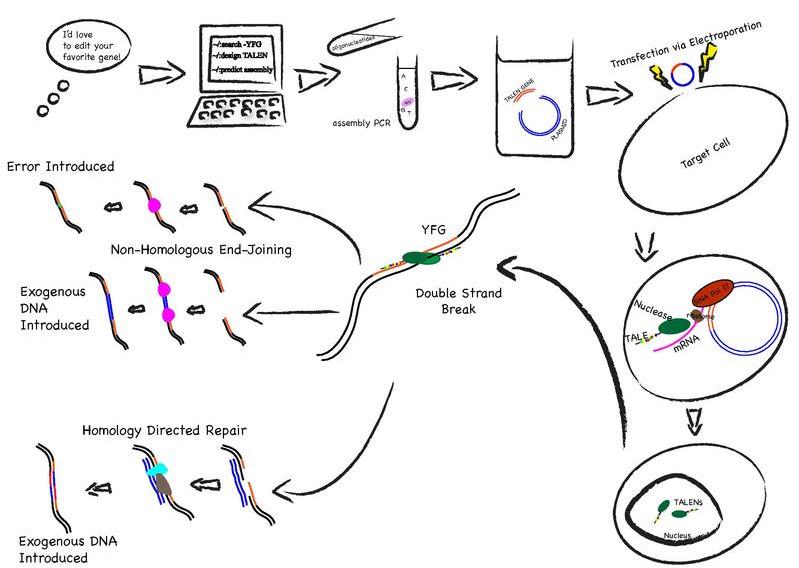
그림 1. Transcriptional activator-like effector nuclease를 이용한 genome engineering (http://en.wikipedia.org/wiki/Tal_effector_nuclease)
지놈엔지니어링이 유전자 서열의 특정부분을 인식하고 표적하는 것을 전제로 하는 특성덕분에 zinc-finger(ZF)나 transcriptionl activator-like(TAL) effector와 같은 DNA-결합 단백질이 하나의 지놈엔지니어링 기술이 될 수 있었다. 제한효소와 같은 DNA 변형효소와 연결된 DNA-결합 단백질은 특정 DNA서열에 결합한 후 잘라줌으로써 이중가닥절단(double strand break; DSB)를 형성 할 수 있고 상동적 재조합(homologous recombination)이나 비상동 말단 봉합(non-homologous end joining; NHEJ)을 통하여 mutation을 도입할 수 있었다. 초기에 각광을 받았던 ZF-nuclease는 motif당 약 3개의 base pairs (bp)를 인식할 수 있었지만 motif를 stacking하더라도 상대적으로 낮은 특이성과 실험실수준에서 얻기엔 비싼 단점으로 인해 최근에는 TALEN(TAL effector nuclease)를 이용하여 지놈엔지니어링을 하는 추세이다. 비록 한 motif당 하나의 bp를 인식하고 큰 size로 인해 assembly가 복잡한 편이지만 긴 DNA서열을 인식하기 위한 stacking이 비교적 쉽기 때문에 지놈엔지니어링 기술로 많이 사용되고 있다. Iowa state university의 Yang`s 팀은 최근 TALEN를 이용하여 효모(Saccharomyces cerevisiae)의 유전체에 있는 3개의 유전자제거(gene knockout)에 성공하였다.6 23bp까지 인식가능한 TALEN을 인위적으로 제작하여 효모의 특정 유전자를 제거하였고 예상되어지는 표현형을 얻을 수 있었다. 또한 상동적 재조합을 통한 유전자 교체도 34%의 효율을 보였다. 이러한 TALEN 활용 지놈엔지니어링은 저비용과 높은 효율로 원하는 표현형을 얻을 수 있는 새로운 기법으로 진핵 생물의 유전자 변형에 상당한 역할을 할 것으로 기대되어진다.7 8
2. Genome editing using an engineered type II CRISPR system
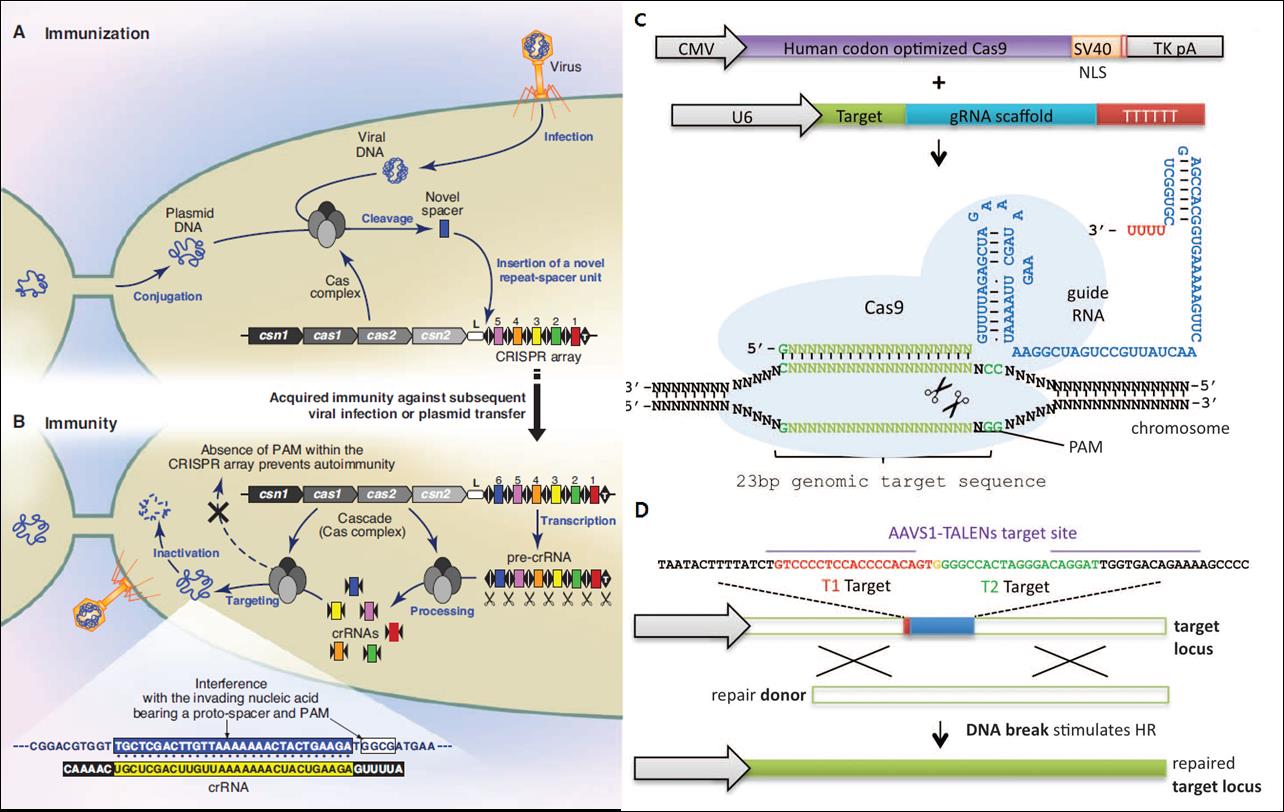
그림 2. (a,b) Mechanism of CRISPR system. (Philippe H et al, Science, 2010) (c,d) Human genome editing with
engineered type II CRISPR system. (Mali P et al, Science, 2013.)
원핵생물의 적응면역시스템 역시 좋은 지놈엔지니어링 도구가 될 수 있다. 파지감염(phage infection)이나 세포간 접합(conjugation)에 의해 세포내로 유입된 외래 유전자와 결합 할 수 있는 RNA와 Cas9 nuclease가 복합체를 이뤄 DSB를 야기하는 CRISPR(clustered regularly interspaced short palindromic repeats)의 원리를 기본으로 하는 새로운 방법이 바로 그 예이다. 최근 하버드 메디컬 스쿨의 Church`s 팀은 CRISPR의 시스템을 이용하여 포유동물세포(mammalian cell)에서 non-sense GFP를 active한 형태로 바꾸었다.9 Cas9 nuclease와 결합할 수 있는 gRNA(guide RNA)와 타겟 유전자를 인식하는 RNA를 결합시켜 세포내로 삽입시키고 그와 동시에 코돈 최적화된 Cas9을 제작하여 세포내로 도입시키게 되면 RNA가 타겟 DNA서열과 RNA-DNA base pairing을 형성하고 Cas9의 nuclease활성이 DSB를 형성하게 된다. 그로 인해 상동적 재조합이 촉진되게 되므로 원하는 유전자형을 도입할 수 있게 된다. 더욱 신기한 것은 Cas9 nuclease가 발현될 때와 그렇지 않을 때의 상동적 재조합 확률이 거의 8배나 차이가 난다는 것이다. 이는 지속적인 Cas9 nuclease의 발현이 상동적 재조합을 촉진한다는 것을 암시하기도 한다. 이러한 CRISPR 시스템은 낮게는 2%에서 높게는 25%까지 유전자 변형 효율을 보였다. 이 결과는 RNA-guided editing은 빠르고 multiplexible한 지놈엔지니어링 도구로써 가능성을 보여줬고 이에 따라 추가적인 연구들이 진행되고 있는 추세이다.10
3. Trackable multiplex recombineering(TRMR) method
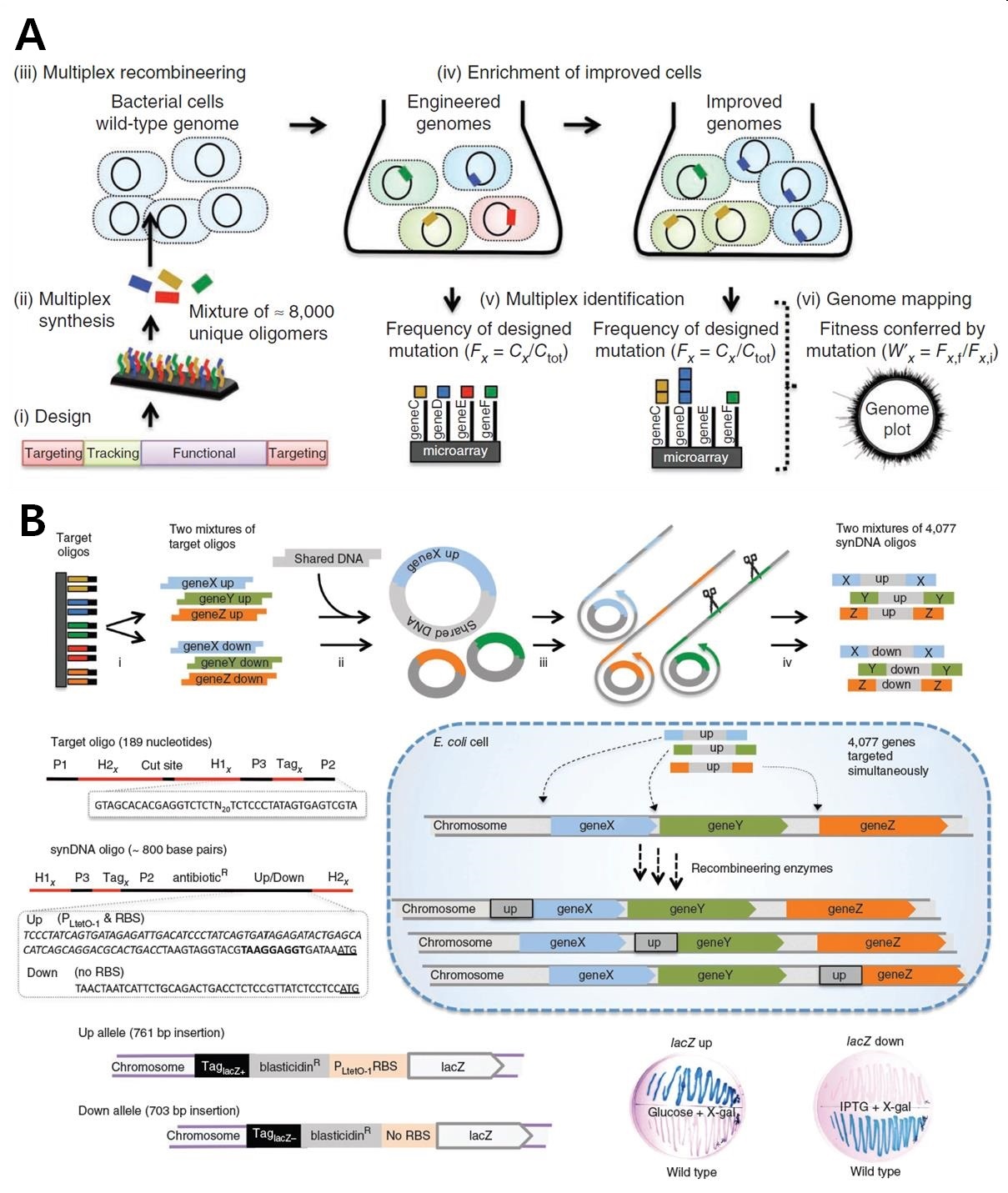
그림 3. (a) Trackable multiplex recombineering(TRMR) method (b) Multiple strategy to rapidly generate cell mixtures with
defined genetic modification. (Warner JR et al, Nat Biotechnol, 2010)
미생물 지놈을 이해하기 위해서는 많은 수의 library가 필요하다는 것을 감안한다면 적절한 유전자변이(genetic variation)을 제작한 후에 그로부터 파생되는 연구를 하는 것이 시간적으로나 비용적으로나 훨씬 경제적이라는 것은 누구도 부정하지 못할 것이다. 최근의 지놈학(genomics)의 발달과 multiplex DNA 합성 기술, 그리고 상동적 재조합 기술의 발달은 이러한 일들이 가능하게 하고 있다. 지놈학의 발달은 유전자 제거와 같은 방법을 통하여 유전자의 특성을 파악하는데 도움을 주었고 이는 유전자의 역활을 확인할 수 있는 강력한 도구가 되었지만 다수 유전자들의 돌연변이에 의해 야기되는 복합적인 변화에서 표현형을 예측하고 분석하는 것에는 아직 아쉬운 성과를 보이고 있었다. 최근 개발된 TRMR은 기존의 문제점을 보완하고 쉽고 빠르게 다수의 돌연변이를 제작할 수 있는 방법으로 소개되었다. 단 1일만에 전체 대장균의 유전자의 95%를 변형하였고 이로부터 원하는 표현형의 미생물을 선별할 수 있었다.11 기존에 흔히 사용되던 on-off방식이 아닌 up-down방식으로 유전자의 발현을 조절함으로써 타겟 유전자에 대해 보다 폭 넓은 이해를 제공하였다. 인위적으로 합성된 dsDNA는 rolling circle amplification에 의해 증폭되고 세포내에서 지놈으로 삽입됨으로써 타겟 유전자의 발현을 조절하게 되는데 이때 만들어지는 mutant library는 선별과정을 거치면서 원하는 표현형의 세포만을 선별할 수 있다. 이러한 과정을 통해 단 일주일만에 원하는 형질에 영향을 주는 유전자들을 확인할 수 있었다. 대다수의 유전자들은 새로이 밝혀진 유전자들이었으며 일부는 기존에 이미 밝혀져 있던 유전자였고 그 중에서는 새로운 기능이 밝혀지기도 하였다. TRMR을 통해서 무수히 많은 mutant library를 제작할 수 있으며 이를 통해 switches, oscillators 혹은 sensors와 같은 추가적인 역할을 하는 유전자들에 대해서도 규명할 수 있을 것이다.12
4. Multiplex automated genome engineering
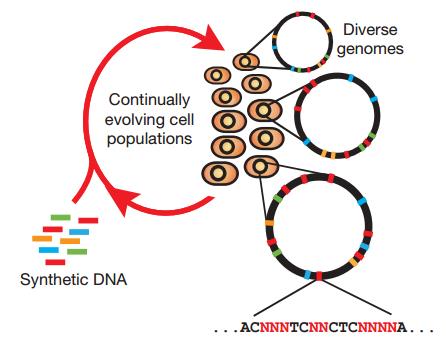
그림 4. Rapid and continuous generation of sequence diversity by MAGE (Wang HH et al, Nature, 2009)
많은 지놈엔지니어링 기술이 하나의 유전자 돌연변이에 주로 초점을 맞춘 데에는 이유가 있다. ZFN이나 TALEN, 그리고 group II intron system을 이용한 기법은 모두 DSB를 야기하기 때문에 다수의 유전자의 돌연변이에는 적절치 않다. 이러한 DSB가 다수 생성되게 된다면 전체 지놈을 파괴할 뿐 아니라 NHEJ에 의해 의도치 않은 rearrangement를 발생시킨다. 또한 λ-red system에 의해 매개되는 dsDNA 재조합 역시 낮은 효율을 보이기 때문에 다수의 돌연변이 제작에는 적절치 않다. 이러한 문제점을 보완하기 위해서 최근에는 dsDNA가 아닌 ssDNA를 이용하는 방법이 소개되었다. MAGE (multiplex automated genome engineering)은 작은 ssDNA가 lagging strand의 Okazaki fragment 합성에 프라머(primer)로 작용하여 딸세포 지놈에 도입되면서 유전자 변이를 유도한다.13 MAGE는 ssDNA를 세포내로 도입시킴으로써 아주 간단하게 다수의 돌연변이를 제작할 수 있었다. 약 30~100bp의 ssDNA는 상동의 말단만 있으면 타겟 유전자 부위에 상보적으로 결합함으로써 우리가 원하는 유전자 서열을 딸세포로 도입시킬수 있다.14 비슷한 원리로 MAGE는 mismatch repair에 관여하는 mutS 유전자를 제거시킴과 동시에 λ-red system의 발현을 통해 ssDNA 삽입을 1000배까지도 증가시킬수 있었다. MAGE와 같이 ssDNA-mediated recombination의 경우 다른 plasmid나 dsDNA보다 유전자 도입(transformation)확률이 높기 때문에 보다 높은 효율의 유전자 재조합을 기대할 수 있을 뿐만 아니라 한 cycle당 소요되는 시간이 상대적으로 다른 기법들보다 짧아 많은 수의 돌연변이 균주를 만들 수 있다. 또한 간단한 원리덕분에 다양한 균주에서도 적용가능할 것으로 예상된다. ssDNA-mediated recombination의 많은 장점 덕분에 현재도 많은 연구들이 추가적으로 진행되고 있는 상황이다.15 16 17 18
나. 미세유체장치를 이용한 우수 균주 선별
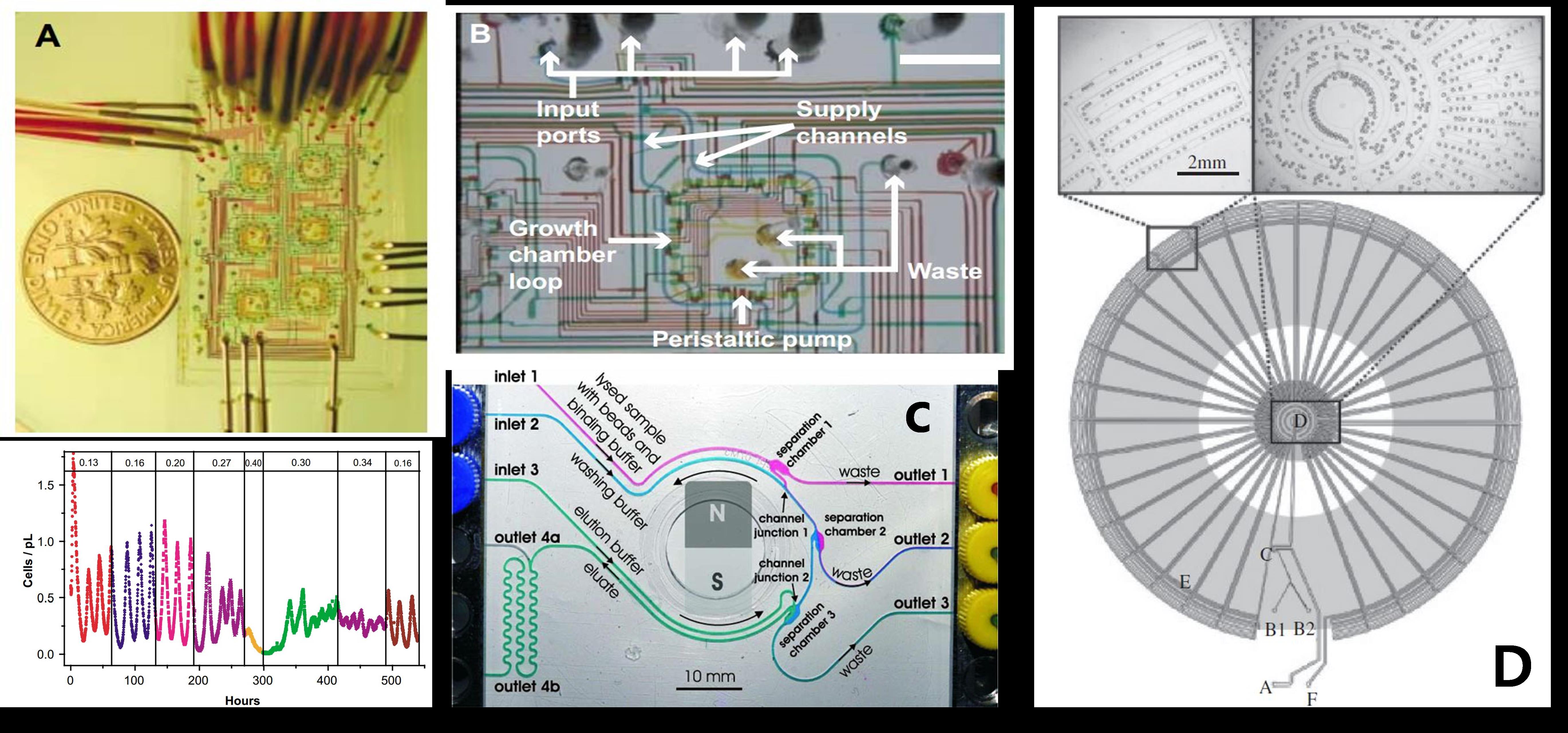
그림 5. 다양한 미세유체장치가 응용된 사례들
다양한 합성생물학적 기술을 사용해서 재구성된 새로운 유전체는 각각의 개체들이 모두 다른 활성을 가지게 한다. 예를 들어 위에 설명된 MAGE와 같은 방법으로 재구성된 DNA를 가진 균주들이 만들어지면, 이를로부터 원하는 표현형의 균주를 선택적으로 분리하기 위해서는 고전적 방법으로는 많은 노동력과 시간이 필요했었다. 하지만 최근 미세유체공학(microfluidics)등과 같은 새로운 기술이 등장하면서 좀 더 손쉽고, 빠르고 효과적으로 고속대량스크리닝 (high-throughput screening; HTS)을 가능하게 하였다. 합성생물학과는 완전히 다른 연구분야인 미세유체공학이 함께 협업(collaboration)되어 성공적일수 있다는 가능성을 보여주었던 사례는 2005년 스탠포드의 퀘이크 연구그룹이 자동화된 미세유체장치 내에서 미생물의 생장을 촉진, 저해, 유도 등과 같이 다양한 방법을 통해 무려 40일간 관찰했던 것이 잘 알려져 있다.19 또한, CIT의 Leadbetter 그룹은 microfluidic 장치를 이용해 기존에 유전정보가 전혀 알려져있지 않았던 미생물 Traponema의 대량 106개의 PCR을 해냈다. 이는 bioMEMs 기술의 장점인 대량의 sample 처리가능능력과 반응에 극소량의 chemical만이 필요한 점을 십분살려, target DNA 자세한 정보를 알지 못해도 특정 gene의 존재여부를 가려낼 수 있는 PCR등의 기술에 적용 할 수 있다는 것을 보여준다.20
위에 설명된 두가지 미세유체 장치를 기점으로 한 다양한 지놈엔지니어링기술은 미세유체장치를 이용한 기술들과 함께 최근까지 적용되어져 왔다. 예를 들면, 가장 기본적인 PCR과 같은 기술들이 미세유체장치내에서 재현되었고21. 위에 설명된 퀘이크 그룹의 bioreactor는 여전히 많은 관심을 받고 있으며, DNA sequence22, 특정된 단백질의 검량, 검출이 가능한 자동화된 장치23, 크기나 다른 외부 요인을 사용한 생물체의 separation위한 미세유체장치24, 단시간내에 107~9개의 샘플 분석이 가능한 구획화된 미세방울 생성 및 저장장치25 등등 여러가지 방법들이 합성생물학 분야 연구에 활용되고 있다.
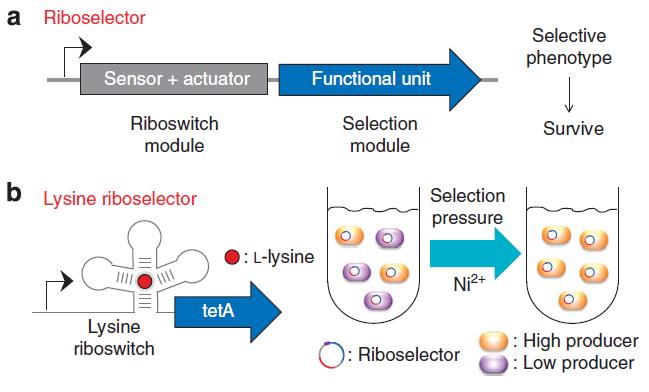
그림 6. High-throughput screening of strains (Jina Y et al, Nat commun, 2013)
이러한 지놈엔지니어링을 접목한 합성생물학과 대사공학은 굉장히 많은 platform 균주들을 만들어냈고 ethanol26, higher alcohol27, fatty acids28 등이 많은 재조합 균주들로부터 생산되고 있다. 최근에는 MAGE와 TRMR과 같이 다중엔지니어링 기술이 발달함에 따라 수많은 미생물군집에서 적절한 균주를 선별하기 위한 스크리닝 기술이 필요하게 되었다. 하지만 형광물질을 통한 특정 균주 선별29이나 enrichment와 같은 선택압(selective pressure)을 사용하는 기존의 HTS30는 많은 시간이 소모된다는 단점을 가지고 있다. 이에 따라 생물학적 스크리닝보다 좀 더 쉽고 빠른 방법으로 bioMEMs가 떠오르고 있고 관련 연구가 수행되고 있다. 이처럼 지놈 엔지니어링은 합성생물학 및 대사공학의 발전을 위한 충실한 도구로써 그리고 bioMEMs는 새로운 파트너로써 인공미생물의 개발에 중요한 역할을 하고 있다.
참고문헌
1. Synthetic biology inches toward the mainstream, CNN, Cherise Fong,
http://edition.cnn.com/2008/TECH/10/17/digitalbiz.synbio/
2. Playing God, BBC Documentary, http://www.youtube.com/watch?v=AKxmqMH4w_A
3. Eight great technologies, UK speeches, https://www.gov.uk/government/speeches/eight-great-technologies
4. Altenburg, E (1928) The limit of radiation frequency effective in producing mutations. The American Naturalist
5. Auerbach C, Robson JM, Carr JG (1947) Chemical Production of Mutations. Science
6. Li T, Huang S, Zhao X, Wright DA, Carpenter S, Spalding MH, Weeks DP, Yang B (2011) Modularly assembled designer
TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes. Nucleic Acids Res.
7. Sander JD, Cade L, Khayter C, Reyon D, Peterson RT, Joung JK, Yeh JR (2011) Targeted gene disruption in somatic
zebrafish cells using engineered TALENs. Nat. Biotechnol.
8. Hockemeyer D, Wang H, Kiani S, Lai CS, Gao Q, Cassady JP, Cost GJ, Zhang L, Santiago Y, Miller JC, Zeitler B,
Cherone JM, Meng X, Hinkley SJ, Rebar EJ, Gregory PD, Urnov FD, Jaenisch R (2011) Genetic engineering of human
pluripotent cells using TALE nucleases. Nat. Biotechnol.
9. Mali P, Yang L, Esvelt KM, Aach J, Guell M, DiCarlo JE, Norville JE, Church GM (2013) RNA-guided human genome
engineering via Cas9. Science
10. Cong L, Ran FA, Cox D, Lin S, Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA, Zhang F (2013) Multiplex
genome engineering using CRISPR/Cas systems. Science
11. Warner JR, Reeder PJ, Karimpour-Fard A, Woodruff LB, Gill RT (2010) Rapid profiling of a microbial genome using
mixtures of barcoded oligonucleotides. Nat. Biotechnol.
12. Lu TK, Khalil AS, Collins JJ (2009) Next-generation synthetic gene networks. Nat.Biotechnol.
13. Wang HH, Isaacs FJ, Carr PA, Sun ZZ, Xu G, Forest CR, Church GM (2009) Programming cells by multiplex genome
engineering and accelerated evolution. Nature
14. Ellis HM, Yu D, DiTizio T, Court DL (2001) High efficiency mutagenesis, repair, and engineering of chromosomal DNA
using single-stranded oligonucleotides. Proc. Natl. Acad. Sci. USA
15. Swingle B, Markel E, Costantino N, Bubunenko MG, Cartinhour S, Court DL (2010b) Oligonucleotide recombination in
Gram-negative bacteria. Mol. Microbiol.
16. van Pijkeren JP, Britton RA (2012) High efficiency recombineering in lactic acid bacteria. Nucleic Acids Res.
17. Sawitzke JA, Costantino N, Court DL (2011) Probing cellular processes with oligo-mediated recombination; using
knowledge gained to optimize recombineering. J. Mol. Biol.
18. Esvelt KM , Wang HH (2013) Genome-scale engineering for systems and synthetic biology. Mol. Sys. Biol.
19. Balagaddé FK, You L, Hansen CL, Arnold FH, Quake S (2005) Long-term monitoring of bacteria undergoing
programmed population control in a microchemostat. Science
20. Ottesen E A, Hong JW, Quake SR, Leadbette JR (2006) Microfluidic digital PCR enables multigene analysis of
individual environmental bacteria. Science
21. Lagallya ET, Simpson PC, Mathies RA (2000) Monolithic integrated microfluidic DNA amplification and capillary
electrophoresis analysis system. Sensor and Acuators
22. Hahm J. Lieber CM (2004) Direct ultrasensitive electrical detection of DNA and DNA sequence variations using
nanowire nanosensors. Nano Lett.
23. Hellmich W, Pelargus C, Leffhalm K (2005) Single cell manipulation, analytics, and label‐free protein detection in
microfluidic devices for systems nanobiology. Electrophoresis
24. Inglis DW, Riehn R, Austin RH (2004) Continuous microfluidic immunomagnetic cell separation. Appl. Phy. Lett.
25. Huebner A, Bratton D, Whyte G, Yang M (2009) Static microdroplet arrays: a microfluidic device for droplet trapping,
incubation and release for enzymatic and cell-based assays. Lab on a Chip
26. Ingram LO, Gomez PF, Lai X, Moniruzzaman M, Wood BE, Yomano LP, York SW (1998) Metabolic engineering of
bacteria for ethanol production. Biotechnol. Bioeng.
27. Atsumi S, Hanai T, Liao JC (2008) Non-fermentative pathways for synthesis of branched-chain higher alcohols as
biofuels. Nature
28. Steen EJ, Kang Y, Bokinsky G, Hu Z, Schirmer A, McClure A, Del Cardayre SB, Keasling JD (2010) Microbial
production of fatty-acid-derived fuels and chemicals from plant biomass. Nature
29. Hoover SW, Tyler Youngquist J, Angart PA, Withers ST, Lennen, RM, Pfleger BF (2012), Isolation of improved free fatty
acid overproducing strains of Escherichia coli via nile red based high-throughput screening. Environ. Prog.
Sustainable Energy
30. Yang J, Seo SW, Jang S, Shin S-I, Lim CH, Roh T-Y, Jung GY (2013) Synthetic RNA devices to expedite the evolution
of metabolite-producing microbes. Nat. Commun.





