합성 생물학에서의 무세포 단백질 합성 시스템의 응용

이경호 박사, 김동명 교수
충남대학교
1. 개요
합성 생물학은 새로운 생물학적 시스템의 구현을 위한 생물학적 부품들의 합성, 엔지니어링 그리고 조합에 기초를 두고 있다. DNA, RNA그리고 단백질 등의 생물학적 부품들로 새롭게 설계된 모듈들은 단계적 확장을 통해 목적 산물의 최적화된 생산을 위한 시스템으로 이어진다. 세포 내에서 대부분의 생물학적 기능과 화학 반응 등을 수행하는 단백질은 합성생물학 시스템을 구성하는 기본적인 구성 요소 중의 하나이다. 한편, 생물학적 부품으로 사용될 수 있는 단백질들을 암호화하는 유전자 서열은 유전자 서열분석기술의 급속한 발전과 함께 기하급수적으로 증가하고 있으나 이에 비하여 단백질의 구조 분석 및 기능을 밝히는 작업은 유전정보의 증가속도를 따르지 못하고 있다 (Figure 1). 이는 기존의 세포 기반의 단백질 합성법으로 유전정보를 번역하는 속도 및 쓰루풋의 한계에 기인한다고 할 수 있으며, 생물학적 부품의 고속개발을 위해서는 대안적인 단백질 합성법이 필요한 실정이다.1이러한 맥락에서 무세포 단백질 합성 시스템은 살아 있는 세포의 사용에서 비롯되는 세포 기반 단백질 합성법의 한계를 벗어나, 유전자 발현의 분자생물학적 접근을 용이하게 하도록 다양한 형태의 유연성을 제공한다. 본 란에서는 유전 정보의 번역에만 국한되지 않고 목적 단백질의 3차 구조 형성 및 유지를 위하여 다양하게 표준화된 무세포 단백질 합성법을 소개하고, 더 나아가 무세포 단백질 합성 시스템이 다른 영역의 과학 분야에서 행해지는 다양한 실험 기법들과의 조합을 통하여 게놈 서열의 신속한 기능 분석을 위한 새로운 개념의 단백질 합성 및 분석 시스템으로 발전되는 내용을 담고 있는 연구들을 소개하고 고찰하고자 한다.
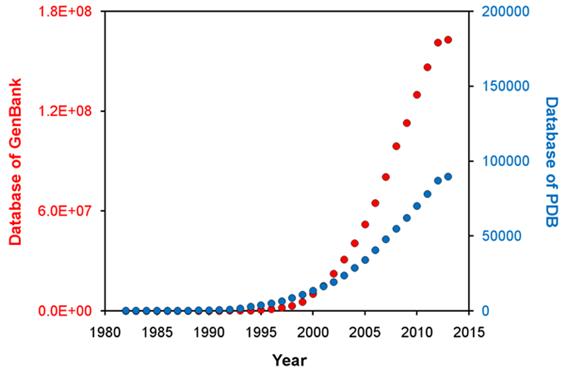
Figure 1. Widening gap between the amounts of sequence information (red circles)
and of protein structures (blue circles).
2. 목적 단백질의 특성에 따라 표준화된 무세포 단백질 합성 시스템
무세포 단백질 합성 시스템은 살아 있는 세포를 이용한 세포 기반의 합성법과 차별화된 접근 방식을 채택하고 있다. 기본적으로 세포 기반의 단백질 합성법은 목적 단백질을 암호화하는 유전자가 형질전환된 세포를 배양하고 세포 파쇄 과정을 거쳐 수거하여 단백질을 확보한다. 반면에 무세포 단백질 합성법은 배양된 세포를 파쇄하여 리보오좀을 포함한 단백질 합성 기구들이 존재하는 세포 추출액을 우선적으로 확보하고, 준비된 세포 추출액에 유전자와 함께 추가적으로 아미노산, 에너지원 및 완충용액 등을 함께 첨가하여 목적 단백질을 합성하도록 한다. 살아 있는 세포를 사용하지 않고, 세포로부터 추출된 단백질 생합성 기구를 이용하여 세포 바깥에서 단백질을 생산하는 무세포 단백질 합성 시스템의 가장 큰 특징은 인위적으로 합성 조건을 조작할 수 있다는 것이다. 세포의 생장에 필요한 복잡한 대사 네트워크의 유지가 필요하지 않기 때문에 단백질 합성에 영향을 미치지 않는 다면 세포 독성을 갖거나 치명적인 영향을 주는 화합물의 사용이 가능하다.
인위적으로 반응 조건을 조작할 수 있는 무세포 단백질 합성 시스템의 대표적인 모델로는 Ueda연구팀에 의해 개발된 “PURE (Protein synthesis Using Recombinant Elements)” 시스템을 들 수 있다.2 이 연구팀은 대장균의 31가지의 단백질 생합성 번역 인자들을 개별적으로 준비하고, 46종 tRNA, 리보조옴 및 단백질 합성 기질들을 따로 준비한 후 목적 유전자와의 혼합물을 만들어 단백질을 합성할 수 있는 시스템을 구축하였다. 이러한 PURE 시스템은 각각의 인자들을 쉽게 대체할 수 있기 때문에 목적 단백질 합성뿐만이 아니라mRNA display, 비천연 아미노산의 도입 등에 적합하게 개선되어 활용되기도 하였다. 하지만, 단백질 발현 인자들의 개별적인 발현 및 분리/정제 과정이 필수적인 PURE 시스템은 스케일-업을 통한 목적 단백질의 대량 생산에는 적합하지 않다.
낮은 수율의 한계를 갖는 PURE시스템과 비교하여 세포 추출액을 사용하는 무세포 단백질 합성 시스템은 수 mg/ml의 발현량으로 높은 수율로 단백질을 합성할 수 있는 장점을 가지고 있으며,3 이러한 시스템을 이용하여 목적 단백질의 높은 발현 수율을 유지하면서도 목적에 맞게 단백질 합성 인자들을 재구성하고자 하는 연구들이 진행되어왔다. 세포 추출액에 존재하는 tRNA를 제거하여 유전자적 리프로그래밍을 이용하여 단백질을 엔지니어링 하거나,4-6 세포 파쇄액내의 저해 인자를 선택적으로 제거하여 단백질 합성 효율을 증가시키고자 하는 시도들이 진행되어왔으며,7 이렇듯 목적에 맞게 단백질 합성 반응 환경을 조절할 수 있는 무세포 단백질 합성 시스템은 더 나아가 목적 단백질의 3차 구조를 고려하여 안정적인 접힘을 유도하고자 다양한 형태로 활성형 단백질 합성을 위한 표준화가 진행되었다.
단백질 발현 수율을 높이는 시도와 더불어 효과적인 단백질의 접힘과 번역 후 수식과정을 위하여 표준화된 무세포 단백질 합성 시스템이 개발되었다 (Figure 2). 단백질의 접힘은 그 기능과 밀접한 관계를 가지고 있고, 환원 조건의 대장균 유래의 무세포 단백질 합성 시스템은 이황화결합을 갖는 목적 단백질의 합성을 제한한다. 이황화결합은 주로 세포질 구획에서 일어나는데, 원핵세포에서는 periplasm이나 진핵세포의 endoplasmic reticulum의 관상에서 일어난다. 하지만, 무세포 단백질 합성법에서는 본질적으로 이황화결합 형성을 방해하는 요소들이 존재하며, 이를 해결하기 위하여 세포 추출액 내의 이황화결합의 환원을 유도하는 단백질들을 비활성화시키고, 산화/환원 포텐셜을 조절하여 이황화결합을 목적 단백질에 도입할 수 있으며, 그 결과 단백질의 활성 역시 증가하는 것을 확인할 수 있었다.8-11
무세포 단백질 합성 시스템에서 막단백질의 가용적인 발현을 위하여 단백질의 구조적 안정성을 높이고자 계면활성제 혹은 리포오좀과 같은 인위적인 기구들을 사용하기도 한다.12-14 즉, 막단백질의 과발현은 세포의 생장을 저해하는 역할을 하기 쉽지만, 무세포 단백질 합성은 근원적으로 이러한 문제를 피할 수 있다. 하지만, 막단백질의 높은 소수성에 의하여 응집체로 발현되는 경우가 대부분이며, 이러한 문제를 해결하고자 계면활성제 또는 지방산으로 이루어진 리포오좀의 첨가는 세포 내에서의 막댄백질의 구조를 유지할 수 있는 지지체로 작용하여 막단백질의 가용성을 높이면서 활성형 막단백질을 합성할 수 있는 시스템이 개발되었다.
마지막으로 무세포 단백질 합성이 진행되는 동안 번역 후 수식이 가능한 시스템이 구축되었다.15 대장균이나 밀 배아 유래의 세포 추출액은 숙주가 갖는 특성을 이어 받기 때문에 단백질 내로 당의 수식이 가능하지 않다.16 이를 해결하고자 RRL (rabbit reticulocyte), insect cell, Hela cell 등의 다양한 진핵세포를 이용하여 세포 추출액을 마련하여 당 단백질 합성을 가능하게 하였다.17 RRL 기반의 무세포 단백질 합성 시스템은 상대적으로 매우 낮은 단백질 합성 수율을 갖기 때문에 실용성이 떨어지고 insect cell의 경우에는 합성 수율은 상대적으로 높지만 당 수식 효율이 낮은 편이다. 이에 반해 human cell line으로부터 준비된 세포 추출액을 사용할 경우에는 RRL, insect 세포 기반의 시스템에 비하여 단백질 합성 수율도 높을 뿐만 아니라 당 수식의 효율도 가장 높기 때문에 당 단백질의 합성에 적합한 시스템으로 개발되었다.18
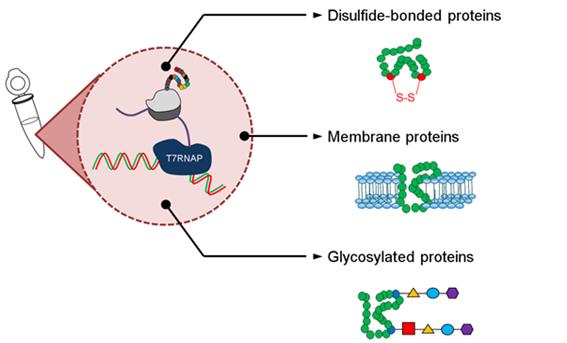
Figure 2. Cell-free protein synthesis coupled with various
post-translational modification reactions
3. 미세유체플랫폼 기반의 무세포 단백질 합성시스템
무세포 단백질 합성 시스템은 목적에 맞게 반응 조성을 조작할 수 있을 뿐만 아니라, 반응기 형태에 구애 받지 않고 인위적인 반응 환경에서 단백질 합성을 수행할 수 있다. 즉, 무세포 단백질 합성 시스템의 가장 큰 장점은 세포 배양이나 클로닝과 같은 단계를 배제하면서 유전 정보를 특정 공간에 구애 받지 않고 번역할 수 있는 것이다. 특히, 마이크로플루이딕스 시스템과의 조합으로 단백질 합성과 분석을 위한 새로운 플랫폼으로 발전되어 왔다 (Figure 3). 미세유체 시스템으로 준비된 수백 나노리터 수준의 공간에서 CECF형식으로 단백질 합성을 진행 함으로써 수 백 가지의 유전자를 동시/병렬적으로 초고속 발현을 가능하게 하는 시스템이 개발되었다.19-21 더 나아가 Quake 연구팀은 단백질-단백질 상호작용 연구에 사용할 수 있는 무세포 단백질 합성법 기반의 미세유체 시스템을 개발하였다.22 유리 슬라이드 크기의 미세유체 반응기에서 약 14,792개의 반응을 동시에 진행할 수 있는 집적도를 갖추었으며, 유전자로부터 발현된 단백질들이 미리 준비된 항체에 고정이 되고 43 종류의 Streptococcus pneumonia 유래의 단백질들이 작은 통로를 통하여 교차반응이 진행된다. 그 후, 단백질-단백질 상호작용이 일어난 단백질 쌍을 같은 회로에서 확인할 수 있었으며, 기존의 연구에서 알려진 상호작용하는 단백질 쌍 이외에도 새로운 종류의 단백질-단백질 상호작용을 일으키는 단백질 쌍을 찾아낼 수 있었다. 이렇게 마이크로플루이딕스 시스템은 초고속/병렬식 단백질 합성을 가능하게 하는가 하면 더 나아가 또 다른 분석 시스템과의 조합을 통하여 새로운 분석 플랫폼을 제공하기도 하였다. Seefeld 연구팀은 마이크로플루이딕스 시스템을 surface plasmon resonance (SPR) 칩 위에서 구현하여 무세포 단백질 합성을 통한 단백질의 합성 과정을 실시간으로 분석할 수 있는 시스템을 개발하였다.23 이러한 분석 시스템은 단백질-단백질, 단백질-DNA 또는 단백질-RNA 등의 상호작용을 실시간으로 분석할 수 있는 새로운 무세포 단백질 합성법 기반의 분석 시스템으로 사용될 수 있다.
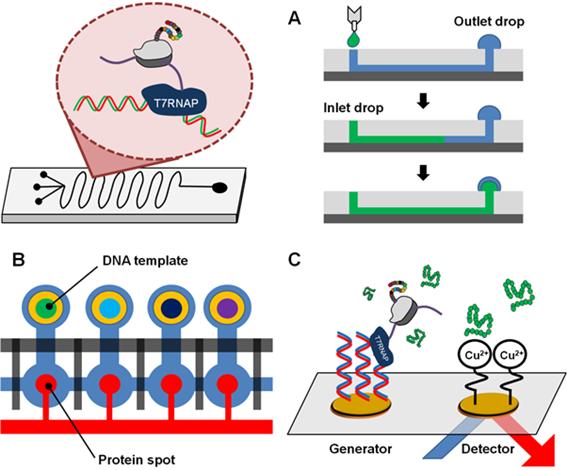
Figure 3. Cell-free protein synthesis based microfluidics system.
A: A cell-free protein synthesis system in a microfluidic device using passive pumping
B: An in vitro microfluidic platform for highthroughput screening of protein interactions,
called protein interaction network generator (PING)
C: A single-step biosensing platform combined cell-free protein synthesis with SPRi.
4. 무세포 단백질 합성 시스템 기반의 in situ 단백질 어레이
단백질 어레이 기술은 수백에서 수천 개의 단백질을 동시에 분석 가능하게 하면서 시간을 단축 시키고 비용을 절감하도록 한다. 하지만, 기존의 세포 기반의 단백질 합성법으로 유전자 클로닝, 세포 배양, 단백질 분리 및 정제를 거쳐 단백질 어레이를 위하여 단백질을 준비하는 데에는 많은 시간과 노동력이 발생한다. 이를 해결하기 위한 대안적인 방법으로 무세포 단백질 합성법 기반의 in situ 단백질 어레이 기법이 개발되어 왔다. Taussig연구팀은 목적 단백질을 인식하는 항체가 바닥에 고정된 24 well 형식의 plate에서 유전자와 함께 무세포 단백질 합성에 필요한 요소들을 함께 반응하여 단백질을 고정하는 ‘protein in situ array (PISA)’ 개념을 도입하였다.24, 25 유전자 어레이와 달리 단백질 어레이는 변성이 일어나기 쉽기 때문에 장기 보존이 힘들다. 이러한 PISA 기법은 필요할 때에 수 시간 내에 단백질 어레이를 준비할 수 있기 때문에 보관 문제를 피할 수 있다. 단백질 어레이가 DNA 어레이 기법과 함께 사용되면서 well plate 형식이 아닌 DNA 어레이로부터 단백질 어레이로 전환하는 기법들이 이어서 개발되었다. LaBaer 연구팀은 먼저 DNA 어레이를 준비한 후 무세포 단백질 합성 동안 발현된 에피톱을 갖는 목적 단백질이 본래의 DNA 어레이 근처에 고정되는 식의 ‘Nucleic acid programmable protein array (NAPPA)’ 기술을 개발하였다.26, 27이러한 기술은 DNA 어레이로부터 무세포 단백질 합성법을 통하여 단백질 어레이로 전환하는 개념을 도입한 것으로 PISA의 기법에 비하여 동시에 처리할 수 있는 유전자의 수를 크게 향상 시킬 수 있었다. 그 후, 유리 슬라이드에서 무세포 단백질 합성법을 기반으로 단백질 어레이의 집적도를 크게 향상시키는 기법이 마련되었다. Hoheisel 연구팀은 ‘multiple spotting technique (MST)’을 통하여 유리 슬라이드 당 13,000개의 달하는 단백질 어레이를 마련할 수 있도록 하였다.28 Ni-NTA가 고정된 슬라이드에 먼저 유전자 어레이를 형성 시키고 유전자가 어레이 되었던 동일한 위치에 단백질 합성을 위한 혼합액을 다시 어레이 하여 무세포 단백질 합성 반응이 진행되고 최종적으로 목적 단백질이 발현되어 고정되도록 하였다.
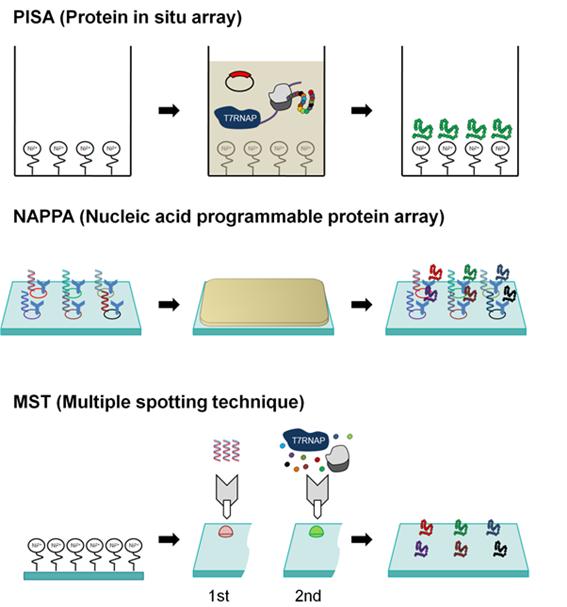
Figure 6. In situ protein array methods based on a cell-free protein synthesis system.
5. 결론 및 고찰
무세포 단백질 합성 시스템은 유전자 클로닝, 배양공정 등 세포 기반의 단백질 합성 시스템이 갖는 한계를 극복하고, 다량의 단백질 부품들을 고속으로 합성 할 수 있는 기술로 사용될 수 있다. 특히, 세포막의 존재 없이 균일한 액상반응 형태로 단백질을 합성할 수 있다는 무세포 단백질 합성기술의 장점은 인해 무세포 단백질 발현 시스템은 단순히 유전정보를 단백질로 번역하는 도구로서의 기능을 뛰어넘어, 인공적인 합성 및 분석 플랫폼과의 융합을 통해 유전자 서열상에 입력된 기능을 효율적으로 해석하고 조작할 수 있는 기술적 기반을 제공한다. 최근 들어, 본론에서 언급된 사례들을 포함하여 세포의 바이오합성기구를 유전자 발현이 요구되는 다양한 응용분야에 적용하고자 하는 연구가 활발히 진행되고 있으며, 무세포 단백질 합성 기술의 활용범위는 향후 지속적으로 확대될 것으로 예상된다.
참고문헌
1. Harris, D.C. and Jewett, M.C. Cell-free biology: exploiting the interface between synthetic biology and synthetic chemistry. Current Opinion in Biotechnology 23, 672-678 (2012).
2. Shimizu, Y., Inoue, A., Tomari, Y., Suzuki, T., Yokogawa, T., Nishikawa, K. and Ueda, T. Cell-free translation reconstituted with purified components. Nature Biotechnology 19, 751-755 (2001).
3. Kim, T.W., Keum, J.W., Oh, I.S., Choi, C.Y., Kim, H.C. and Kim, D.M. An economical and highly productive cell-free protein synthesis system utilizing fructose-1,6-bisphosphate as an energy source. Journal of Biotechnology 130, 389-393 (2007).
4. Jackson, R.J., Napthine, S. and Brierley, I. Development of a tRNA-dependent in vitro translation system. RNA 7, 765-773 (2001).
5. Kanda, T., Takai, K., Yokoyama, S. and Takaku, H. Knocking out a specific tRNA species within unfractionated Escherichia coli tRNA by using antisense (complementary) oligodeoxyribonucleotides. FEBS Letters 440, 273-276 (1998).
6. Kanda, T., Takai, K., Yokoyama, S. and Takaku, H. An easy cell-free protein synthesis system dependent on the addition of crude Escherichia coli tRNA. Journal of Biochemistry 127, 37-41 (2000).
7. Michel-Reydellet, N., Woodrow, K. and Swartz, J. Increasing PCR fragment stability and protein yields in a cell-free system with genetically modified Escherichia coli extracts. Journal of Molecular Microbiology and Biotechnology 9, 26-34 (2005).
8. Goerke, A.R. and Swartz, J.R. Development of cell-free protein synthesis platforms for disulfide bonded proteins. Biotechnology and Bioengineering 99, 351-367 (2008).
9. Kim, D.M. and Swartz, J.R. Efficient production of a bioactive, multiple disulfide-bonded protein using modified extracts of Escherichia coli. Biotechnology and Bioengineering 85, 122-129 (2004).
10. Oh, I.S., Kim, D.M., Kim, T.W., Park, C.G. and Choi, C.Y. Providing an oxidizing environment for the cell-free expression of disulfide-containing proteins by exhausting the reducing activity of Escherichia coli S30 extract. Biotechnology Progress 22, 1225-1228 (2006).
11. Yin, G. and Swartz, J.R. Enhancing multiple disulfide bonded protein folding in a cell-free system. Biotechnology and Bioengineering 86, 188-195 (2004).
12. Bannwarth, M. and Schulz, G.E. The expression of outer membrane proteins for crystallization. Biochimica et Biophysica Acta 1610, 37-45 (2003).
13. Junge, F., Haberstock, S., Roos, C., Stefer, S., Proverbio, D., Dotsch, V. and Bernhard, F. Advances in cell-free protein synthesis for the functional and structural analysis of membrane proteins. New Biotechnology 28, 262-271 (2011).
14. Schwarz, D., Klammt, C., Koglin, A., Lohr, F., Schneider, B., Dotsch, V. and Bernhard, F. Preparative scale cell-free expression systems: new tools for the large scale preparation of integral membrane proteins for functional and structural studies. Methods 41, 355-369 (2007).
15. Endo, Y. and Sawasaki, T. Cell-free expression systems for eukaryotic protein production. Current Opinion in Biotechnology 17, 373-380 (2006).
16. Takai, K., Sawasaki, T. and Endo, Y. The wheat-germ cell-free expression system. Current Pharmaceutical Biotechnology 11, 272-278 (2010).
17. Ezure, T., Suzuki, T., Shikata, M., Ito, M., Ando, E., Utsumi, T., Nishimura, O. and Tsunasawa, S. Development of an insect cell-free system. Current Pharmaceutical Biotechnology 11, 279-284 (2010).
18. Zeenko, V.V., Wang, C., Majumder, M., Komar, A.A., Snider, M.D., Merrick, W.C., Kaufman, R.J. and Hatzoglou, M. An efficient in vitro translation system from mammalian cells lacking the translational inhibition caused by eIF2 phosphorylation. RNA 14, 593-602 (2008).
19. Angenendt, P., Nyarsik, L., Szaflarski, W., Glokler, J., Nierhaus, K.H., Lehrach, H., Cahill, D.J. and Lueking, A. Cell-free protein expression and functional assay in nanowell chip format. Analytical Chemistry 76, 1844-1849 (2004).
20. Kinpara, T., Mizuno, R., Murakami, Y., Kobayashi, M., Yamaura, S., Hasan, Q., Morita, Y., Nakano, H., Yamane, T. and Tamiya, E. A picoliter chamber array for cell-free protein synthesis. Journal of Biochemistry 136, 149-154 (2004).
21. Khnouf, R., Beebe, D.J. and Fan, Z.H. Cell-free protein expression in a microchannel array with passive pumping. Lab Chip 9, 56-61 (2009).
22. Gerber, D., Maerkl, S.J. and Quake, S.R. An in vitro microfluidic approach to generating protein-interaction networks. Nature Methods 6, 71-74 (2009).
23. Seefeld, T.H., Halpern, A.R. and Corn, R.M. On-chip synthesis of protein microarrays from DNA microarrays via coupled in vitro transcription and translation for surface plasmon resonance imaging biosensor applications. J Am Chem Soc 134, 12358-12361 (2012).
24. He, M. and Taussig, M.J. Single step generation of protein arrays from DNA by cell-free expression and in situ immobilisation (PISA method). Nucleic Acids Res 29, e73-73 (2001).
25. He, M. and Taussig, M.J. DiscernArray technology: a cell-free method for the generation of protein arrays from PCR DNA. J Immunol Methods 274, 265-270 (2003).
26. Ramachandran, N., Hainsworth, E., Bhullar, B., Eisenstein, S., Rosen, B., Lau, A.Y., Walter, J.C. and LaBaer, J. Self-assembling protein microarrays. Science 305, 86-90 (2004).
27. Ramachandran, N., Hainsworth, E., Demirkan, G. and LaBaer, J. On-chip protein synthesis for making microarrays. Methods Mol Biol 328, 1-14 (2006).
28. Angenendt, P., Kreutzberger, J., Glokler, J. and Hoheisel, J.D. Generation of high density protein microarrays by cell-free in situ expression of unpurified PCR products. Mol Cell Proteomics 5, 1658-1666 (2006).





