지능형 바이오 시스템에 의한 고품질 항체 의약품 생산성 극대화

한국과학기술원 이균민 교수
1. 개요
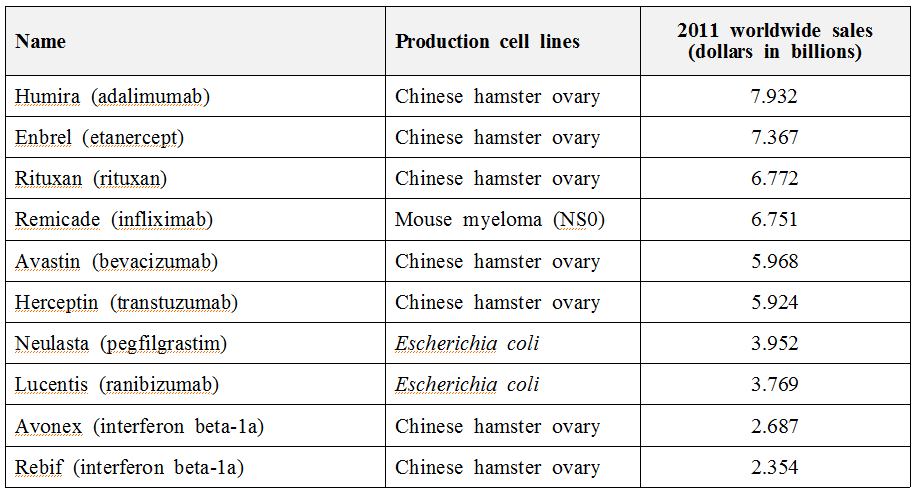
CHO (Chinese Hamster Ovary) 세포주는 치료용 항체 의약품의 생산을 위하여 산업적으로 가장 많이 사용되는 세포주의 하나로, 현재 판매되는 1/3이상의 치료용 단백질이 CHO 세포를 통해 생산되고 있다 (표1). 현재까지 CHO 세포를 이용한 치료용 단백질의 생산은 매년 20-30%의 성장을 거듭하여 시장 규모가 지속적으로 확대되고 있으며 제품 가격도 비싸기 때문에 고부가가치를 창출할 수 있다. 항체 의약품을 비롯한 치료용 단백질은 고용량 투여가 불가피하기 때문에 적정수준의 생산성을 확보하여 제조원가를 낮추는 것이 시장 경쟁력 확보를 통한 제품화 성공의 관건이며, 단순히 생산성을 높이는 것뿐만 아니라 효력이 있는 항체를 손상 없이 균일하게 생산하는 것도 매우 중요하다.
표1. Top selling biologic drugs (Huggett and Lahteenmaki, 2012)
현재까지 고생산성 또는 고품질 항체를 발현하는 CHO 세포주를 구축하기 위해서는 타겟 생산유전자를 유전자 안에 무작위로 삽입하여 과발현 시킨 후 다양한 클론들을 선별하는 과정이 가장 많이 쓰여 왔으며, 이 때 각 후보 클론들의 유전자 증폭 및 배양환경 최적화과정이 필요하다. 하지만 이 과정은 비용, 노동력, 및 시간의 소요가 크고, 각각 클론들의 특성이 예측가능하지 않기 때문에 매 과정마다 각 생산 세포주에 적합한 프로세스를 개발하여야 한다. 따라서 최근에는 최소의 비용과 노동력 및 시간을 소비하면서 항상 예측 가능한 고 생산성 CHO 세포주를 개발 할 수 있는 플랫폼 기술의 확보에 대한 관심이 커지고 있다.
현재 transcriptome, translatome, proteom 등을 관찰하는 -omics 시스템생물학을 이용한 타겟발굴과 세포의 유전자를 직접 조작하는 targeted engineering의 도입이 상당히 진행되고 있다. 이들을 통해 CHO 세포주의 높은 생산성을 도모하고 생산되는 치료용 항체의 품질을 향상시키는 것은 무작위적인 엔지니어링과 비교했을 때 예측 가능한 결과를 얻을 수 있어 실로 지능형 바이오 시스템이라고 할 수 있겠다 (그림1).
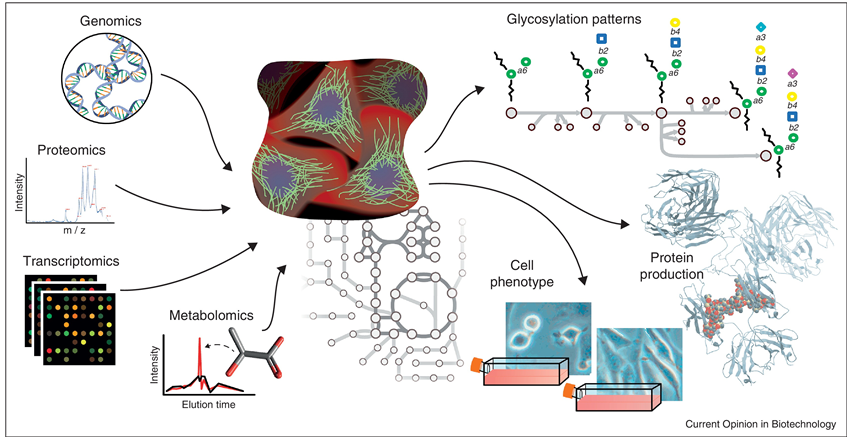
그림1. 시스템 생물학을 이용한 CHO 세포주의 엔지니어링 (Kildegaard et al., 2013)
2. -Omics를 이용한 타겟 발굴
최근 전 세계의 다양한 연구 기관에서 CHO genome을 밝히기 위한 많은 연구가 진행되었으며, genome 정보를 이용한 다양한 분석 방법 및 응용 기술이 개발되고 있다. 대표적인 성과로는 미국의 GT-life sciences가 중국의 BGI-Shenzhen과 합작하여 CHO-K1 세포주의 genome sequence를 밝혀내었다 (Xu et al., 2011). 밝혀진 CHO genome 관련 자료는 온라인상에 database를 구축하여 누구나 무료로 자유롭게 그 소스를 이용할 수 있게 하였고 이 정보를 기반으로 CHO 세포주를 이용한 생물 의약품 생산성 향상을 위한 다양한 연구가 진행될 것으로 전망된다 (Hammond et al., 2012). 또한 CHO 세포주의 기원인 Female Chinese hamster (Cricetulus griseus)의 genome을 sequence하여 그로부터 유래한 다양한 CHO 세포주들의 차이점등을 규명하여 각 세포주들의 이해와 이용에 도움이 될 수 있게 하였다 (그림2) (Lewis et al., 2013).
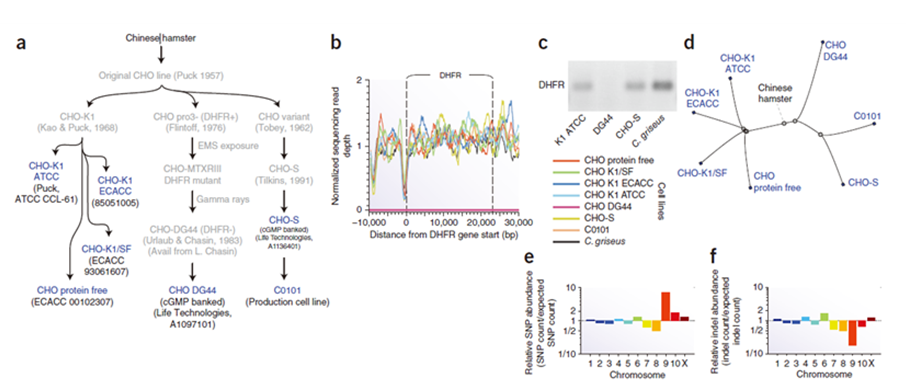
그림2. CHO 세포주들간의 mutation landscape (Lewis et al., 2013)
시스템 생물학을 바탕으로 한 오믹스(omics) 정보 통합 분석 기술은 숙주 세포주의 생산성, 세포 생존율, 생산품의 품질 강화를 위한 타겟의 탐색을 고용량으로 처리(high-throughput) 가능하게 만들었는데, 그 기술은 transcriptome, translatome, proteome 등 유전자 발현의 다양한 단계에 적용되고 있다. 적용 예를 살펴보면, 단백질 고발현 조건인 저온 배양 시와 butyrate 처리 시 CHO 세포내에서 transcriptome과 proteomics가 어떻게 변하는지 살펴보아서 생산성이 높은 세포주 안에서 어떠한 유전자가 많이 발현되는지를 확인한 경우도 있고 세포사멸방지유전자인 Bcl-xL을 발현시킨 고발현 세포주와 대조군인 저발현 세포주를 비교하여 proteome을 확인 한 경우도 있다 (Carlage et al., 2009; Kantardjieff et al., 2010). 또한 nCounter NanoString을 이용하여 세포내 mRNA분석을 통해 세포사멸이 일어나는 스트레스 환경에서 고발현 되는 유전자들을 발굴과 제품 품질을 저해하는 환경에서 고발현 되는 유전자를 발굴하는 연구들도 수행되었다 (Lee et al., 2014; Ha et al., 2015). transcriptome과 proteome간의 차이를 연결하기 위해서는 세포내의 전사효율을 밝히는 translatome의 연구가 진행되어 타겟 유전자 선정을 한층 더 효율적이게 만들 수 있게 되었다 (Courtes et al., 2013). 이렇게 다양한 분석을 통해서 새롭게 발견된 여러 타겟 유전자는 실제 엔지니어링에 적용했을 때 얼마나 효과가 있을지 그리고 얼마나 빠르게 그 screening이 가능할 지가 중요한 포인트가 될 것이다.
3. Targeted engineering의 도입
CHO 세포주의 genome이 밝혀지면서 더욱 활발한 발전이 돋보이는 분야는 targeted engineering이다. Targeted engineering의 경우 원하는 유전자의 염기서열만 있으면 그 유전자를 특이적으로 knockout 할 수도 있고 genome 상의 원하는 부분에 외부유전자를 특이적으로 삽입(knock in) 할 수도 있다. 특히 knockout의 경우 이전에는 RNA interference 기술을 이용한 down regulation만 가능했던 반면 이젠 genome상의 유전자를 mutation시켜서 기능을 완전히 차단함으로서 좀 더 영구적이고 확실하게 특정 유전자를 없애 그 효과를 볼 수 있다. 이러한 방법은 meganuclease, zinc finger nuclease (ZFN), transcriptional activator like effector nuclease (TALEN) 등의 순서로 발달해왔는데, 최근에는 더욱 효율이 좋은 CRISPR/Cas9 기술을 이용한 engineering이 많이 진행되고 있다 (그림3). CHO 세포의 경우 ZFN가 초반기에 knockout을 목적으로 많이 쓰였으며 GS, DHFR, FUT8, Bax, Bak 유전자 등이 knockout 되어서 효과를 보았다 (Cost et al., 2010; Liu et al., 2010; Malphettes et al., 2010; Santiago et al., 2008). 유전자 삽입의 경우에는 아직 CHO 세포에 시도한 예가 없는데, 인간세포주에서는 어느 정도 시도가 되고 있기 때문에 CHO 세포주에도 조만간 도입이 가능할 것으로 예상된다.
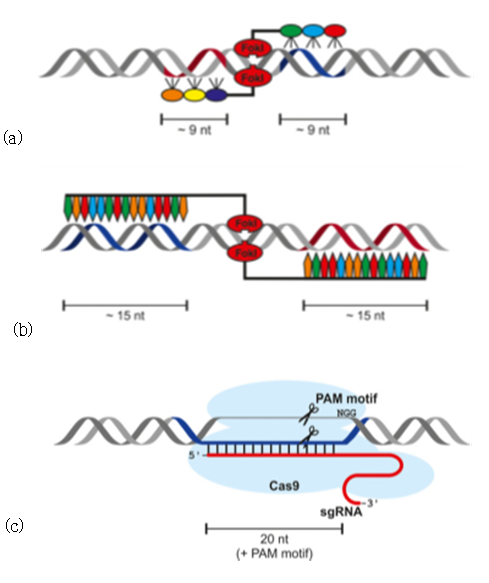
그림3. (a) ZFN, (b) TALEN, (c) CRISPR의 비교
(Puchta and Fauser, 2014)
다양한 targeted engineering 기술 중에서도 CRISPR/Cas9 기술은 보다 간편하고 비용이 낮아서 최신의 연구에 많이 사용되고 있는데, 덴마크의 The Novo Nordisk Foundation Center for Biosustainability에서는 CHO-K1 genome내에 위치하는 single guide RNAs (sgRNAs)를 검색하고 확인할 수 있는 CRISPY라는 web기반의 bioinformatics 툴을 개발하였다. 이 툴은 총 27,553개의 유전자에 달하는 1,970,449개의 타겟 염기서열을 가지고 있으며 genome 상의 off-target sites도 표기하여 CRISPR/Cas9 기술의 사용을 좀 더 용이하게 만들어주었다 (Ronda et al., 2014).
4. 고찰
밝혀진 CHO genome과 오믹스 기술을 이용한 시스템 생물학의 사용은 무작위적인 엔지니어링보다 세포의 본질인 유전체에 좀 더 직접적으로 다가가기 때문에 보다 예측 가능한 엔지니어링을 할 수 있다는 것에 큰 의미가 있다. 아직까지는 여러 연구팀들이 모두 사용이 가능한 baseline reference를 형성하는 단계에 그치지만 이후에는 단백질의 발현과 그 품질에 영향을 끼치는 유전자들을 list up 하고 이를 적용하는 것이 중요해질 것이다. 그러나 유전자의 전사정도 (transcriptome)와 단백질의 발현정도 (proteome)를 비교해보면 항상 그 정도가 비례하지 않아 그 사이에 밝혀지지 않은 무언가가 있을 것으로 예상되며, 어떠한 유전자형(genotype)과 표현형(phenotype)을 변경시켰을 때 그것이 세포가 생산하는 항체의 품질에 어떠한 영향을 끼치는 지도 확실한 이해가 성립되지 않았다. 이러한 것들은 아직까지 직접적인 엔지니어링을 통해 확인하는것이 최선인데, 다행이도 targeted engineering과 이를 위한 web기반 데이타베이스를 이용하여 좀 더 빠르고 손쉬운 knockout 엔지니어링이 가능하게 되었고 이는 현재 CHO 세포주를 연구하는 많은 그룹에서 실행하고 있는 연구이기도 하다. 현재 활발히 진행되고 있는 knockout 기술 외에 knock in 기술도 CHO 세포주에서 적용이 곧 가능하게 될 것인데 이는 주로 genome상에서 외부 유전자를 고발현 할 수 있는 자리를 찾는데 연구의 초점이 맞춰지게 될 것이며, CHO 세포주에 존재하지 않는 유전자를 발현시켜서 생산되는 항체의 품질을 높이는데도 적용될 수 있을 것이다.
참고문헌
1. Carlage, T., Hincapie, M., Zang, L., Lyubarskaya, Y., Madden, H., Mhatre, R., and Hancock, W.S. (2009). Proteomic profiling of a high-producing Chinese hamster ovary cell culture. Analytical chemistry 81, 7357-7362.
2. Cost, G.J., Freyvert, Y., Vafiadis, A., Santiago, Y., Miller, J.C., Rebar, E., Collingwood, T.N., Snowden, A., and Gregory, P.D. (2010). BAK and BAX deletion using zinc-finger nucleases yields apoptosis-resistant CHO cells. Biotechnology and bioengineering 105, 330-340.
3. Courtes, F.C., Lin, J., Lim, H.L., Ng, S.W., Wong, N.S., Koh, G., Vardy, L., Yap, M.G., Loo, B., and Lee, D.Y. (2013). Translatome analysis of CHO cells to identify key growth genes. J Biotechnol 167, 215-224.
4. Ha, T.K., Kim, Y.G., and Lee, G.M. (2015). Understanding of Altered N-glycosylation-Related Gene Expression in Recombinant Chinese Hamster Ovary Cells Subjected to Elevated Ammonium Concentration by Digital mRNA Counting. Biotechnology and bioengineering, Accepted.
5. Hammond, S., Kaplarevic, M., Borth, N., Betenbaugh, M.J., and Lee, K.H. (2012). Chinese hamster genome database: an online resource for the CHO community at www.CHOgenome.org. Biotechnology and bioengineering 109, 1353-1356.
6. Huggett, B., and Lahteenmaki, R. (2012). Public biotech 2011--the numbers. Nature biotechnology 30, 751-757.
7. Kantardjieff, A., Jacob, N.M., Yee, J.C., Epstein, E., Kok, Y.J., Philp, R., Betenbaugh, M., and Hu, W.S. (2010). Transcriptome and proteome analysis of Chinese hamster ovary cells under low temperature and butyrate treatment. J Biotechnol 145, 143-159.
8. Kildegaard, H.F., Baycin-Hizal, D., Lewis, N.E., and Betenbaugh, M.J. (2013). The emerging CHO systems biology era: harnessing the 'omics revolution for biotechnology. Current opinion in biotechnology 24, 1102-1107.
9. Lee, S.M., Kim, Y.G., Lee, E.G., and Lee, G.M. (2014). Digital mRNA profiling of N-glycosylation gene expression in recombinant Chinese hamster ovary cells treated with sodium butyrate. J Biotechnol 171, 56-60.
10. Lewis, N.E., Liu, X., Li, Y., Nagarajan, H., Yerganian, G., O'Brien, E., Bordbar, A., Roth, A.M., Rosenbloom, J., Bian, C., et al. (2013). Genomic landscapes of Chinese hamster ovary cell lines as revealed by the Cricetulus griseus draft genome. Nature biotechnology 31, 759-765.
11. Liu, P.Q., Chan, E.M., Cost, G.J., Zhang, L., Wang, J., Miller, J.C., Guschin, D.Y., Reik, A., Holmes, M.C., Mott, J.E., et al. (2010). Generation of a triple-gene knockout mammalian cell line using engineered zinc-finger nucleases. Biotechnology and bioengineering 106, 97-105.
12. Malphettes, L., Freyvert, Y., Chang, J., Liu, P.Q., Chan, E., Miller, J.C., Zhou, Z., Nguyen, T., Tsai, C., Snowden, A.W., et al. (2010). Highly efficient deletion of FUT8 in CHO cell lines using zinc-finger nucleases yields cells that produce completely nonfucosylated antibodies. Biotechnology and bioengineering 106, 774-783.
13. Puchta, H., and Fauser, F. (2014). Synthetic nucleases for genome engineering in plants: prospects for a bright future. The Plant journal : for cell and molecular biology 78, 727-741.
14. Ronda, C., Pedersen, L.E., Hansen, H.G., Kallehauge, T.B., Betenbaugh, M.J., Nielsen, A.T., and Kildegaard, H.F. (2014). Accelerating genome editing in CHO cells using CRISPR Cas9 and CRISPy, a web-based target finding tool. Biotechnology and bioengineering 111, 1604-1616.
15. Santiago, Y., Chan, E., Liu, P.Q., Orlando, S., Zhang, L., Urnov, F.D., Holmes, M.C., Guschin, D., Waite, A., Miller, J.C., et al. (2008). Targeted gene knockout in mammalian cells by using engineered zinc-finger nucleases. Proceedings of the National Academy of Sciences of the United States of America 105, 5809-5814.
16. Xu, X., Nagarajan, H., Lewis, N.E., Pan, S., Cai, Z., Liu, X., Chen, W., Xie, M., Wang, W., Hammond, S., et al. (2011). The genomic sequence of the Chinese hamster ovary (CHO)-K1 cell line. Nature biotechnology 29, 735-741.





