합성 조절 sRNAs 개발 및 대사공학에서의 응용에 대한 연구 동향


노민호 - KAIST 생명화학공학과 박사과정
이상엽 - KAIST 생명화학공학과 특훈교수
1. 개요
생명활동에 있어서 유전자의 발현 조절은 여러 유전자들의 상호작용에 있어서 매우 중요한 부분이며 RNA 조절자는 원핵생물과 진핵생물의 많은 생리 조절 기작에서 유전자의 발현 조절에 매우 중요한 부분을 차지한다. 미생물의 대표적인 RNA 조절자는 그 작용 기작에 따라 크게 두 가지로 볼 수 있는데, cis-element로 작용하는 riboswitch는 mRNA의 UTR에 위치하여 세포내 환경에 따라서 유전자의 발현을 조절하여, trans-element로 작용하는 small RNAs (이하 sRNAs) 는 mRNA에 결합하여 리보솜에 의한 번역을 억제 혹은 활성화 시키거나 mRNA의 수명에 영향을 주는 것으로 알려져 있다. sRNAs는 mRNA와 분리되어 있기 때문에 하나의 sRNA로 여러 유전자의 발현에 영향을 미칠 수 있으며, 탄소 대사 경로의 조절, 아미노산 생산의 조절 등 대사경로의 미묘한 조절이나, 다양한 환경적인 스트레스에 대응하여 생리적으로 즉시 적응하기 위해서 기민하게 유전자 발현을 조절하는 기작으로 작용한다. 특히 sRNA는 목표유전자를 염색체 상에서 편집하지 않고도 세포가 지닌 유전자의 발현을 억제할 수 있다는 매력 때문에 바이오 분야의 연구에 있어서 강력한 도구로 각광받고 있으며 이를 활용하고자 하는 연구도 활발히 이루어지고 있다. 본래 미생물이 지니는 sRNA를 조작하여 연구자가 원하는 특질을 지니도록 제작한 합성 sRNAs는 적은 비용으로 단시간에 많은 유전자에 대해서 적용할 수 있는 장점이 있으며 따라서 그 응용성이 무궁하여 대사회로를 조절하거나 최적화 시킬 필요가 있는 대사공학에서는 다양한 유전자의 조작하기 위한 도구로 그 응용성이 확장되고 있다. 그리고 앞으로 생물 기반으로 창출되는 새로운 산업 분야의 창출 및 성장에 있어서 매우 중요한 역할을 할 것으로 기대 되며 바이오 의약이나 White BT의 발전을 도울 것이라 생각된다. 본 보고서에서는 sRNAs가 발견된 이래로 합성 sRNAs의 개발 및 대사공학에서의 응용에 대한 연구에 대하여 고찰해보고자 한다.
2. 합성 sRNAs의 발전
sRNAs가 발견된 이래로 이를 이용하여 목표 유전자의 발현을 조절하려는 시도가 활발히 이루어졌다. 30여년 전 대장균에서 최초의 sRNA로 MicF가 발견되었으며 이는 OmpF 유전자의 번역을 억제하는 sRNA였다. 그 당시 이 MicF에 인공적인 DNA 서열을 삽입하여 Ipp, OmpA, OmpC의 유전자 발현을 억제하는 실험에 성공하였다(Coleman et al., 1984). 최근, 기존의 sRNA의 구조를 분석하고 응용하여 인공 sRNA들를 개발하여 외부적으로 넣어준 EGFP나 대장균에 내재되어있는 uidA 유전자의 억제를 가능하게 한 연구가 이루어졌다. 하지만 이렇게 개발된 인공 sRNA들은 그 서열이 일정하지 않고 목표 유전자에 대한 특이성이 떨어지는 문제가 있었다 (Man et al., 2011). 형광단백질을 리포터 유전자로 사용한 고효율의 스크리닝을 통하여 특정 목표 유전자의 발현을 억제하는 sRNA를 발견하는 연구도 이루어졌으며 (Sharma et al., 2012) 자동화된 방법으로 디자인과 실험 병행하여 세포 내에서 목표 유전자의 발현을 억제할 수 있는 합성 sRNA 제작 시스템을 구축되었다. (Rodrigo et al., 2012). 그리고 sRNA의 구조에 대해서 깊이 있는 연구가 진행되면서 sRNA의 각 부위의 역할이 밝혀지면서 어떤 부분이 mRNA의 특이성에 중요한 역할을 하며 합성 sRNA의 제작에 중요한 지가 점차 밝혀졌으며 (Maki et al., 2010;Otaka et al., 2011;Ishikawa et al., 2012;Gottesman et al., 2004), 이에 따라 직관적으로 좀 더 쉽고 편리하게 목표 유전자를 억제하는 sRNA를 제작할 수 있게 되었다.
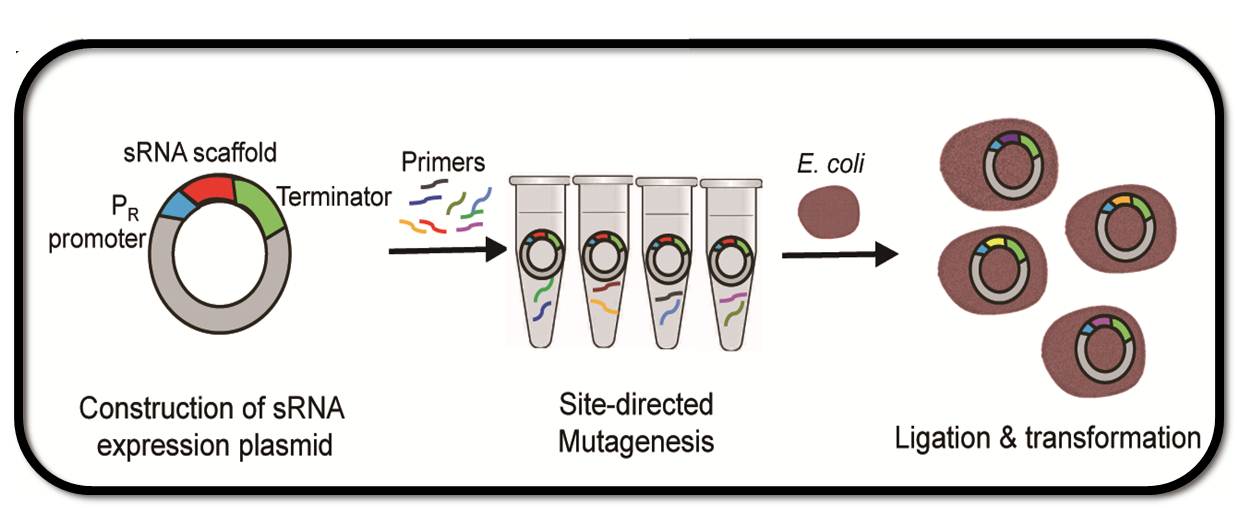
그림1. mRNA 결합 부위에 돌연변이를 일으켜 목표 유전자 발현을 억제하는 sRNA제작 기법
3. 대사공학에 있어서 sRNAs의 응용
대사공학에서 antisense RNA 전략은 Clostridium acetobutylicum에서 쓰여진 바가 있다. antisense RNA를 과발현하여 butyrate kinase, phosphobutyrylase, phosphotransacetylase의 활성을 억제하여 아세톤과 부탄올의 생산성을 높였었다. 이와 비슷하게 대장균에서도 antisense RNA 전략이 사용되었으며 acetate kinase와 phosphotransacetylase의 발현을 억제하여 외래 단백질의 생산량을 높이는 연구가 진행된 바 있다 (Kim and Cha, 2003). 하지만 이러한 antisense RNA 전략은 특이성이나 유전자 발현 억제의 효율이 일정하지 않다는 단점을 지니며 이는 antisense RNA의 길이에서 기인하는 것으로 이 전략의 태생적인 문제점으로 지적받았다 (Sharma et al., 2012). 반면에 sRNAs는 일반적으로 80 ~ 140 bp 정도로 길이가 짧기 때문에 대사 경로의 조작 등에 있어서 antisense RNA보다 매력적인 도구로 관심을 받고 있다. 대장균에서 sRNAs인 RhyB를 과발현하여 부산물을 줄이고 목표로 했던 숙신산의 생산량을 7배까지 늘리는 연구가 진행된 바 있으며 (Kang et al., 2012), 이 연구를 기점으로 sRNAs의 대사공학 도구로서 잠재력을 인정받기 시작하였다. 5-aminolevulinic산을 생산하는 대장균 균주에서 RhyB의 과발현을 통하여 최종 생산 농도를 16% 증가시키는 연구도 발표되었으며 sRNAs인 SgrS의 과발현을 통하여 대장균 K-12균주에서 대표적인 부산물인 아세트산의 생산을 감소시키는 연구를 수행한 바도 있다 (Negrete et al., 2013). 그리고 기존에 발견된 sRNA의 구조 및 목표 유전자와 그 목표 유전자에 결합하는 서열에 대한 연구가 활발히 이루어져 sRNA의 구조체에 목표 유전자의 시작 코돈으로부터 24 mer의 서열만 결합하여 발현하면 목표 유전자의 발현을 억제할 수 있게 되었다. 이러한 기술이 원하는 산물을 생산하기 위하여 관련 대사회로를 조작하는 도구로 활용되었다. 이 연구에서는 MicC라는 sRNA의 구조체를 이용하여 원하는 목표 유전자의 발현을 억제할 수 있도록 목표 유전자의 유전자의 시작코돈 부근의 서열을 포함한 합성 sRNA를 제작하였으며, 이를 이용하여 1) 특정 유전자의 발현 억제를 여러 균주에 적용하여 최적의 생산 균주를 찾아내는 스크리닝 및 2) 130 여개의 sRNA를 제작하여 특정 산물의 생산성을 늘릴 수 있는 발현 억제 목표 유전자를 찾는 스크리닝을 수행하여 타이로신과 카다베린의 생산성을 높일 수 있는 유전자를 성공적으로 찾을 수 있었다 (Na et al., 2013;Yoo et al., 2013).
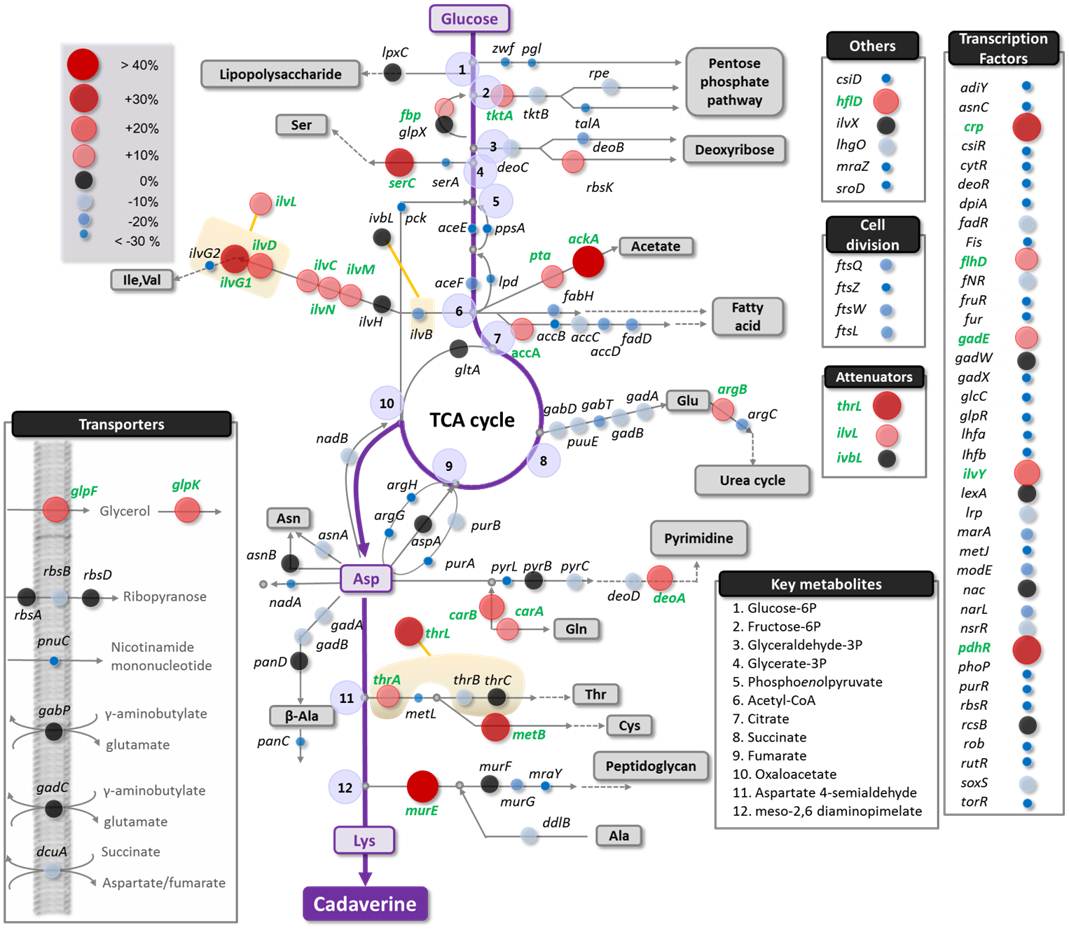
그림2. 130여개의 sRNA를 활용하여 cadaverine 생산성을 높이는 억제 목표 유전자의 screening
이 연구를 기반으로 개발된 sRNA 제작 시스템은 여러 분야에 응용되어 대사공학으로는 대장균으로부터 포도당을 페놀로 만드는 균주의 제작에 활용되며 (Kim et al., 2014), 그람 양성 균주인 Bacillus subtilis 균주에서 글루코사민의 생산성을 높이는 연구에서도 응용된다 (Liu et al., 2014). 또한 파지 기반으로 미생물의 항생제 저항 유전자의 발현 억제를 통해서 미생물에 대한 항생제의 효과를 높이는 전략에서 응용되었다 (Libis et al., 2014). 그리고 기존의 대사공학 연구에서는 유전자의 결실을 기반으로 유전자 조작을 수행하여 세포의 성장에 필수적인 유전자의 조작이 불가능하였지만, 해당 유전자의 발현을 미세하게 억제하여 세포의 성장과 생산하고자 하는 산물의 생산 사이의 균형을 맞추어 최적화하는 연구도 활발히 이루어져 여러 연구 성과가 발표될 전망이다.
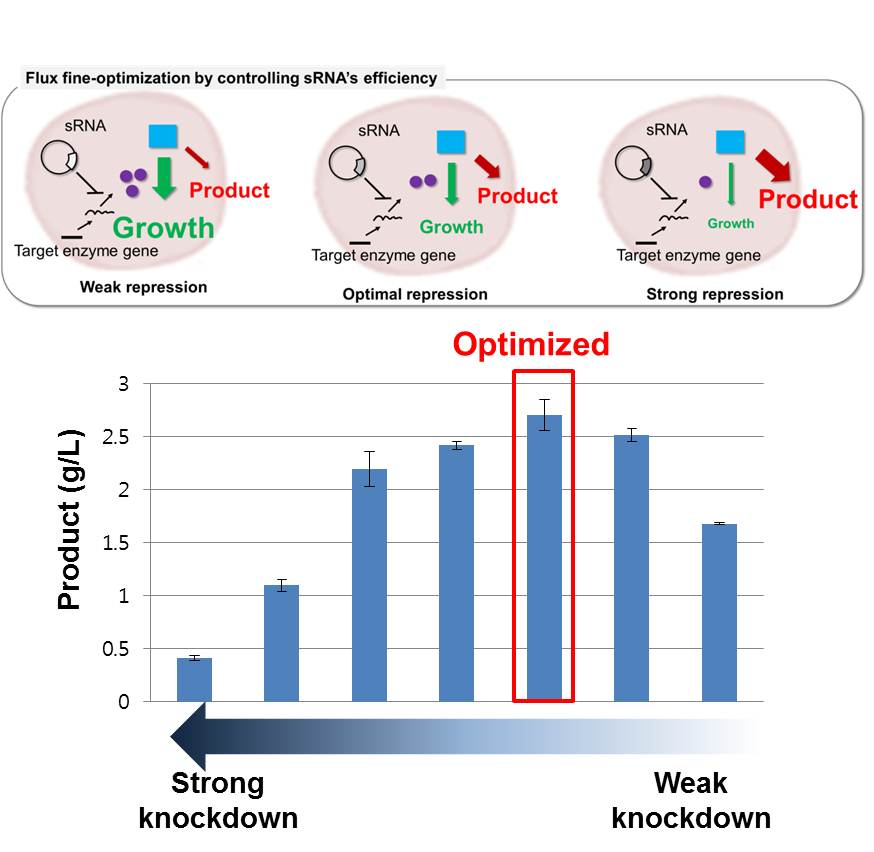
그림3. 세포 성장과 목표 화합물 생산 사이의 최적화를 위한 sRNA의 활용법
4. 고찰
sRNA는 30년 전에 이미 발견되었지만, 오랜 기간 동안 큰 관심을 받지 못 하였다. 하지만 분자생물학 기반 기술들이 급속도로 발전하고, 합성생물학과 대사공학이라는 연구 분야가 성장해나가면서 2010년 이후에 크게 관심을 받기 시작하였고, 유전자 전달체 기반으로 원하는 유전자의 발현을 억제할 수 있는 손쉽고 빠르게 사용할 수 있는 강력한 도구로 발전되었다. 그 잠재성은 아직 무궁무진하며 이 도구를 업그레이드하고 활용하고자 하는 연구가 활발히 이루어지고 있으며 대장균뿐만이 아니라 여러 미생물에서도 그 응용성이 기대되고 있다. 장차 White BT 기반의 친환경, 친생물적인 산업의 성장이 기대되는 만큼 sRNA는 더욱 중요한 연구 도구로 자리매김하게 될 것이라 전망된다.
참고문헌
1. Mizuno T, Chou MY, Inouye M (1984) A unique mechanism regulating gene expression: translational inhibition by a complementary RNA transcript (micRNA). Proc Natl Acad Sci U S A 81:1966–1970
2. Coleman J, Green PJ, Inouye M (1984) The use of RNAs complementary to specific mRNAs to regulate the expression of individual bacterial genes. Cell 37:429–436
3. Man SA, Cheng RB, Miao CC, Gong QH, Gu YC, Lu XZ, Han F, Yu WG (2011) Artificial trans-encoded small non-coding RNAs specifically silence the selected gene expression in bacteria. Nucleic Acids Res 39:e50
4. Sharma V, Yamamura A, Yokobayashi Y (2012) Engineering artificial small RNAs for conditional gene silencing in Escherichia coli. ACS Synth Biol 1:6–13
5. Rodrigo G, Landrain TE, Jaramillo A (2012) De novo automated design of small RNA circuits for engineering synthetic riboregulation in living cells. Proc Natl Acad Sci U S A 109:15271–15276
6. Maki, K., Morita, T., Otaka, H., & Aiba, H. (2010). A minimal base-pairing region of a bacterial small RNA SgrS required for translational repression of ptsG mRNA. Mol. Microbiol., 76(3), 782–92.
7. Otaka, H., Ishikawa, H., Morita, T., & Aiba, H. (2011). PolyU tail of rho-independent terminator of bacterial small RNAs is essential for Hfq action. Proc Natl Acad Sci U S A, 108(32), 13059–64.
8. Ishikawa, H., Otaka, H., Maki, K., Morita, T., & Aiba, H. (2012). The functional Hfq-binding module of bacterial sRNAs consists of a double or single hairpin preceded by a U-rich sequence and followed by a 3’ poly(U) tail. RNA (New York, N.Y.), 18(5), 1062–74.
9. Gottesman, S. (2004). The small RNA regulators of Escherichia coli: roles and mechanisms*. Annu. Rev. Microbiol., 58, 303-328.
10. Desai RP, Papoutsakis ET (1999) Antisense RNA strategies for metabolic engineering of Clostridium acetobutylicum. Appl Environ Microbiol 65:936–945
11. Kim JY, Cha HJ (2003) Down-regulation of acetate pathway through antisense strategy in Escherichia coli: improved foreign protein production. Biotechnol Bioeng 83:841–853
12. Kang Z, Wang XR, Li YK, Wang Q, Qi QS (2012) Small RNA RyhB as a potential tool used for metabolic engineering in Escherichia coli. Biotechnol Lett 34:527–531
13. Li FF, Wang Y, Gong K, Wang Q, Liang QF, Qi QS (2013) Constitutive expression of RyhB regulates the heme biosynthesis pathway and increases the 5-aminolevulinic acid accumulation in Escherichia coli. FEMS Microbiol Lett.
14. Negrete A, Majdalani N, Phue JN, Shiloach J (2013) Reducing acetate excretion from E. coli K-12 by over-expressing the small RNA SgrS. N Biotechnol 30:269–273
15. Na D, Yoo SM, Chung H, Park H, Park JH, Lee SY (2013) Metabolic engineering of Escherichia coli using synthetic small regulatory RNAs. Nat Biotechnol 31:170–174
16. Yoo SM, Na D, Lee SY (2013) Design and use of synthetic regulatory small RNAs to control gene expression in Escherichia coli. Nat Protoc 8:1694–1707
17. Kim, B., Park, H., Na, D., & Lee, S. Y. (2014). Metabolic engineering of Escherichia coli for the production of phenol from glucose. Biotechnology journal, 9(5), 621-629.
18. Libis, V. K., Bernheim, A. G., Basier, C., Jaramillo-Riveri, S., Deyell, M., Aghoghogbe, I., ... & Wintermute, E. H. (2014). Silencing of Antibiotic Resistance in E. coli with Engineered Phage Bearing Small Regulatory RNAs. ACS synthetic biology, 3(12), 1003-1006.
19. Liu, Y., Zhu, Y., Li, J., Shin, H. D., Chen, R. R., Du, G., ... & Chen, J. (2014). Modular pathway engineering of Bacillus subtilis for improved N-acetylglucosamine production. Metabolic engineering, 23, 42-52.





