코리네박테리움 기반 합성생물학적 연구 동향


KAIST 정기준 교수, 임성순 박사과정
1. 개요
분자생물학의 발전과 더불어 유전공학 기술들의 발전을 통해서 합성생물학 생물학적 시스템 엔지니어링 기술은 생명공학 시대에 새로운 산업 분야를 창출하며 생명과학 산업의 모습을 빠르게 변화시키고 있다. 특히 합성생물학은 바이오의약 뿐만 아니라 White BT, Green BT의 본격적 성장을 가능케 만들 것으로 기대되고 있다. 그 동안의 합성생물학 기반의 기술들은 대장균, 효모와 같이 다양한 genetic tool이 확립되어 있는 미생물에 기반해서 개발 및 응용이 되어오고 있었는데, 점차 Bacillus subtilis, Pichia pastoris, Streptomyces avermitilis, cyanobacteria까지 그 대상 미생물의 범위가 넓어지고 있다.
Corynebacterium glutamicum (코리네박테리움 글루타미쿰)은 포자를 형성하지 않는 그람 양성균이며, 병원성이 없는 GRAS (Generally recognized as safe) 균주로써 다양한 L-아미노산, 핵산 등을 생산하는 데에 산업적으로 널리 사용되고 있는 유용한 산업 균주이다.
또한 코리네박테리움은 아미노산 생산뿐만 아니라 그람 양성균의 특성으로 인한 용이한 단백질 분비 생산에서도 큰 장점을 가지고 있다. 그리고 현재의 아미노산이나 단백질 생산을 넘어 코리네박테리움을 통해 다양한 바이오 화합물을 생산하고자 하는 연구가 활발히 진행되고 있다. 본 보고서에서는 이렇게 유용한 산업균주 Corynebacterium glutamicum (코리네박테리움 글루타미쿰) 기반의 합생생물학적 연구 동향에 대해서 고찰해 보고자 한다.
2. 코리네박테리움 tunable gene expression system 연구 동향
코리네박테리움에서 다양한 목표 화합물을 고효율로 생산하려면 목표 화합물로의 대사 흐름을 최적으로 조절해주는 것뿐만 아니라 필요에 따라서는 새로운 합성 대사 경로를 구축해야 하는데 이를 위해서는 다양한 유전자의 발현을 정교하게 조절할 수 있는 기술이 필요하다. 기존 코리네박테리움의 유전자 발현 조절을 위해서는 보통 강한 발현 정도가 알려져 있는 몇몇 개의 코리네박테리움 유래의 프로모터(PsodA, PgapA, Peftu, PcspB)나 대장균 유래의 프로모터(Plac, Ptrc, Ptac) 등이 이용되어 왔다. 하지만 각각의 프로모터의 상대적인 세기는 직접적으로 비교된 바가 없으며, 강하다고 알려져 있는 프로모터를 사용하더라도 5’UTR (untranslated region)과 목표 유전자 서열과의 조합 등의 효과에 의해 달라질 수 있는 발현 효율 때문에 다양한 세기의 유전자 발현을 목표로 할 때, 목표 유전자에 대해서 최적의 세기를 보이는 각각의 프로모터를 선별하는 것이 매우 어려운 실정이었다.
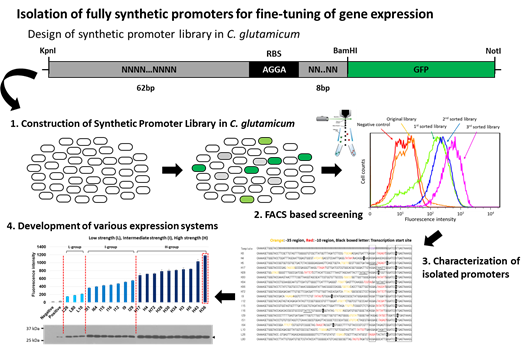
그림 1. 코리네박테리움 기반 합성 프로모터 라이브러리의 구축 및 FACS를 통한
다양한 세기의 프로모터 발굴
따라서 이러한 문제점을 해결하고자 2013년 본 연구진은 코리네박테리움에서 GFP를 보고 단백질로 활용한 합성 프로모터 라이브러리를 구축하였고, 이를 FACS (Fluorescence-Activated Cell Sorting) screening을 통해 다양한 세기를 보이는 constitutive 합성 프로모터들을 발굴할 수 있었다(그림 1). 여기서 발굴된 거의 대부분의 합성 프로모터들은 5’UTR을 포함하지 않으며 목표 유전자의 ATG 개시코돈부터 전사를 시작하는 leaderless transcription을 보이는 것으로 확인 되었는데, leaderless transcription system은 각각의 목표 유전자의 발현에 대해서 5’UTR의 효과를 배제하고 프로모터의 세기만을 가지고 예측 가능한 발현 시스템을 구축할 수 있기에 기존의 사용되어오던 유전자 발현 시스템에 비해서 매우 큰 장점을 가진다고 할 수 있겠다. 또한 최근에는 Rytter et al., 2014.에서 코리네박테리움 프로모터들에서 확인된 consensus sequence를 이용하여 다양한 종류의 합성 프로모터 라이브러리를 구축하여 다양한 세기의 합성 프로모터를 구축하였을 뿐만 아니라, 대장균 lac 프로모터에 기반한 프로모터 라이브러리를 통해 코리네박테리움에서 다양한 세기를 보이는 IPTG-inducible 프로모터들 또한 개발했음을 보고하였다.
Promoter 영역에서뿐만 아니라 코리네박테리움에서 점점 더 많은 DNA part들이 개발되면서 이를 조합해서 시험하고 사용할 수 있도록 해줄 합성생물학 기반의 plasmid platform이 필요하게 되었는데 이는 2012년 Ravasi et al., 2012.의 pTGR platform과 2014년 Kang et al., 2014.의 CoryneBrick platform에 의해서 구축되었다. 두 platform 모두 목표 유전자의 발현을 위한 프로모터, RBS, Terminator, Selection marker, E. coli origin, C. glutamicum origin 등을 특수한 제한 효소를 통해서 조합하고 시험할 수 있는 시스템이다. 특히 CoryneBrick platform은 목표 유전자들을 원하는 대로 조합하고 조립하기 쉬운 BglBrick 클로닝 방법을 도입하여 더 간편한 합성생물학 기반 plasmid platform이라 할 수 있겠다.
3. 코리네박테리움에서의 metabolite biosensor 연구 동향
코리네박테리움에서 목표 metabolite를 고효율로 대량생산 하기 위해 많은 대사공학적 접근이 이루어졌다. 그리고 대사공학의 여러가지 한계점을 극복하기 위해 합성생물학 개념을 도입하여 더 빠르고 정확하게 목표 화합물 생산 시스템을 구축하고자 하는 연구가 활발히 진행되고 있는데 대표적으로 세포 내 Transcription factor를 통해 세포 내의 대사산물의 양을 감지하는 biosensor 시스템이 있다. Biosensor 시스템은 세포 내에 존재하는 대사산물의 양을 감지하여 필요한 유전자의 발현을 조절할 수 있는데, 이를 이용하여 대사산물의 양을 형광 단백질의 발현으로 연결하여 FACS를 통하여 분석하고, 그를 통해 최적 유전자 발현 시스템 구축, 효소 engineering 또는 genome engineering을 high-throughput으로 수행하는 연구가 코리네박테리움에서도 보고되었다.
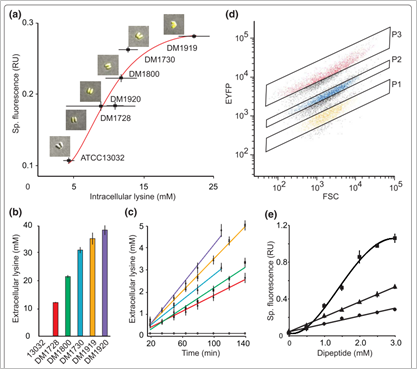
그림 2. 코리네박테리움의 세포 내 라이신 농도에 따라서 EYFP를 발현시키는
라이신 센서 시스템
독일의 Lothar eggeling group이 코리네박테리움의 metabolite sensor를 통해 많은 연구를 수행하고 있는데 대표적으로는 세포 내 라이신 농도가 높아질수록 라이신을 세포 밖으로 배출하기 위한 라이신 exporter LysE의 발현에 관련된 LysG transcriptional regulator와 lysE 유전자의 프로모터를 이용하여 라이신 농도에 따라 EYFP를 생산하는 라이신 센서를 구축한 예가 있다. 그리고 이러한 아미노산 센서를 이용하여 코리네박테리움 유전체 돌연변이에서 라이신 대량 생산에 도움이 되는 새로운 mutation 등을 식별하는 연구, 알지닌에 feedback inhibition을 덜 받는 ArgB 효소를 engineering 연구 등을 FACS를 통하여 High-throughput으로 수행하여 보고하였다 (그림 2). 이는 합성생물학에 기반한 유전자 발현 시스템 구축을 통해서 코리네박테리움에서 대사산물을 정확하게 감지하고 이를 통해 다양한 engineering을 신속하게 할 수 있다는 것을 보여주는 결과라 할 수 있겠다.
4. 코리네박테리움의 합성생물학 기반 genome engineering 연구 동향
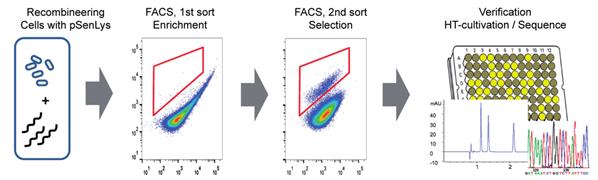
그림 3. Prophage의 RecT recombinase를 통한 genome engineering과
라이신 바이오 센서 시스템을 이용한 코리네박테리움 균주 개발
코리네박테리움의 genome engineering은 대체로 시간이 많이 들고 손이 많이 가는 전통적인 방법으로 homologous recombination에 의한 gene knock-out 혹은 integration을 통해서 이루어져왔다. 하지만 최근 Binder et al., 2013에 보고된 바에 따르면 single-strand DNA와 prophage recombinase RecT를 이용하여 genome의 특정한 위치에 정확하게 mutation을 일으키고, 목표로 하는 대사산물의 양을 감지하여 그 양에 따라 형광 단백질을 발현하는 바이오센서를 함께 이용하여 유익한 mutation을 가지는 돌연변이를 FACS를 통하여 발굴해내는 RecFACS 기술이 개발되었고, 이를 통해 코리네박테리움에서도 다중의 genome engineering이 가능하게 되었다. 또한 zinc-finger nucleases, transcription activator-like effector nucleases (TALENs) 그리고 CRISPR-Cas nuclease system과 같은 genome engineering tool 또한 코리네박테리움의 더 효과적인 균주 개발에 응용될 수 있을 것으로 보인다.
5. 고찰
이렇듯 다양한 합성생물학 기반 genetic tool이 코리네박테리움에서도 구축이 되고 있으며, 이를 통해서 더욱 더 쉽고 빠르고 효과적인 코리네박테리움의 engineering이 용이해지고 있다. 하지만 여전히 코리네박테리움에서 활용 가능한 engineering tool은 대장균 및 효모에 비해서 많이 부족한 실정이며 많은 연구가 더 필요하다. 대장균 및 효모에 비해서 산업용 균주로서 큰 장점을 지닌 코리네박테리움에서 더 다양한 합성생물학적인 genetic engineering tool이 개발된다면 White BT 분야에 미칠 수 있는 파급효과는 매우 클 것으로 예상한다.
참고문헌
1. 김태억, 김성우. 합성생물학 (Synthetic biology) 연구개발 및 산업화 동향 –
바이오산업 범용기술플랫폼. 생명공학정책연구센터.
2. Woo HM, Park JB. 2014. Recent progress in development of synthetic biology platforms
and metabolic engineering of Corynebacterium glutamicum. J Biotechnol 180:43-51.
3. Becker J, Wittmann C. 2011. Bio-based production of chemicals, materials and fuels –
Corynebacterium glutamicum as versatile cell factory. Curr Opin Biotechnol 23:1-10.
4. Yim SS, An SJ, Kang M, Lee J, Jeong KJ. 2013. Isolation of fully synthetic promoters for
high-level gene expression in Corynebacterium glutamicum. Biotechnol Bioeng
110:2959-2971.
5. Rytter JV, Helmark S, Chen J, Lezyk MJ, Solem C, Jensen PR. 2014. Synthetic promoter
libraries for Corynebacterium glutamicum. Appl Microbiol Biotechnol 98:2617-2623.
6. Ravasi P, Peiru S, Gramajo H, Menzella HG. 2012. Design and testing of a synthetic
biology framework for genetic engineering of Corynebacterium glutamicum. Microbial Cell
Fact 11:147.
7. Kang MK, Lee J, Um Y, Lee TS, Bott M, Park SJ, Woo HM. 2014. Synthetic biology
platform of CoryneBrick vectors for gene expression in Corynebacterium glutamicum and
its application to xylose utilization. Appl Microbiol Biotechnol 98:5991-6002.
8. Zhang F, Carothers JM, Keasling JD. 2012. Design of a dynamic sensor-regulator system
for production of chemicals and fuels derived from fatty acids.
Nat Biotechnol 30:354-359.
9. Binder S, Schendzielorz G, Stäbler N, Krumbach K, Hoffmann K, Bott M, Eggeling L. 2012.
A high-throughput approach to identify genomic variats of bacterial metabolite producers
at the single-cell level. Genome Biol 13:R40
10. Schendzielorz G, Dippong M, Grünberger A, Kohlheyer D, Yoshida A, Binder S,
Nishiyama C, Nishiyama M, Bott M, Eggeling L. 2014. Taking control over control: use of
product sensing in single cells to remove flux control at key enzymes in biosynthesis
pathways. ACS Synth Biol 3:21-29.
11. Binder S, Siedler S, Marienhagen J, Bott M, Eggeling L. 2013. Recombineering in
Corynebacterium glutamicum combined with optical nanosensors: a general strategy for
fast producer strain generation. Nuc Acids Res 41:6360-6369.





