합성 조절 sRNA를 이용한 유전자 발현 미세조절 시스템 및 대사공학적 응용 연구
한국과학기술원 이상엽 교수
Noh M, Yoo SM, Kim WJ, and Lee SY (2017). Gene expression knockdown by modulating synthetic small RNA expression in Escherichia coli. Cell Systems 2017 September DOI: 10.1016/j.cels.2017.08.016
1. 연구배경
미생물 유전자 조작 기술의 발달과 함께 미생물로부터 원하는 물질을 생산하거나 미생물에 새로운 기능을 부여하기 위한 연구가 대사공학 및 합성생물학 분야에서 활발히 이루어졌다. 이를 위하여 다양한 유전자 조작 도구들이 개발되어 왔는데, 그 중 대표적인 것이 유전자의 결실이다. 염색체에서 유전자를 삭제함으로 미생물의 기능을 원하는 대로 조작하는 기법이다. 하지만 이 기법은 많은 시간이 소요되며 세포 생장에 필수적인 유전자의 결실은 생장 저해 및 원치 않은 물질의 생산 등으로 많은 어려움을 야기한다. 또한 다양한 균주 또는 유전자에 적용하는 경우 전 과정을 반복적으로 수행해야 하는 문제점으로 인해 현대 기술의 발달과 함께 요구되는 복잡한 유전자 네트워크 조작 응용에 한계를 나타내고 있다. 따라서 본 연구진은 기존 연구를 통하여 합성 조절 sRNA를 이용한 유전자 발현 조절 기술을 개발하였으며, 이는 벡터 형태로 미생물 내 유전자의 발현을 쉽게 조절 가능케 하였을 뿐 아니라 유전체 수준의 sRNA 라이브러리의 제작을 통해 다양한 균주와 유전자에 대하여 동시 다발적으로 적용할 수 있는 지점에 이르렀다.
2. 연구내용
합성 조절 sRNA는 세포 내 mRNA와 일대일로 결합하여 해당 mRNA의 번역을 통한 단백질 생성을 막는다. 세포 내에는 각 유전자에 대하여 다수의 mRNA가 존재하며 이들의 생성과 분해가 섬세하게 조절되어 해당 유전자의 발현량을 생리적으로 조절하게 되어 있다. 따라서 본 연구진은 이 사실에 착안하여 sRNA의 발현량을 조절함으로써 목표 유전자의 발현량을 다양하게 조절할 수 있을 것이라 기대하여 아래 그림과 같이 합성 조절 sRNA를 이용한 유전자 발현 조절 전략을 수립하게 되었다 (그림 1).
그림 1. 합성 조절 sRNA의 발현량 조절을 이용한 유전자 발현 미세조절 전략 및
이를 응용한 화합물 생산과 세포 성장간의 조절 전략
다양한 강도를 지니는 합성 프로모터를 도입하여 합성 조절 sRNA의 발현량을 조절해 유전자 발현을 조작하는 이 전략은 합성 조절 sRNA 특유의 짧은 시간과 간단한 실험 과정을 거쳐 유전자의 발현을 미세조절 할 수 있다. 기존의 유전자 발현 미세조절 기법으로는 염색체에 존재하는 프로모터를 교체하거나 돌연변이를 도입하는 등의 방식이 있었다. 하지만, 이 방법은 매우 긴 시간이 요구되며, 특히 여러 유전자의 발현량을 동시에 조작하기 위해서는 순차적으로 유전자의 조작을 수행해야하기 때문에 유전자 수에 비례하여 긴 시간이 소요된다. 하지만 합성 조절 sRNA의 경우에는 동시에 여러 유전자들을 동시에 미세조절 할 수 있어 섬세한 조작이 필요한 대사회로의 최적화에 필수적인 도구가 될 것으로 전망하였다.
□ 미세 조절을 위한 합성 조절 sRNA 플랫폼의 목표 유전자 발현 억제능 테스트
본 연구진은 우선적으로 이러한 합성 조절 sRNA 미세조절 플랫폼의 효과를 확인하기 위하여 형광단백질을 도입하여 유전자 발현 테스트를 수행하였다 (그림 2).
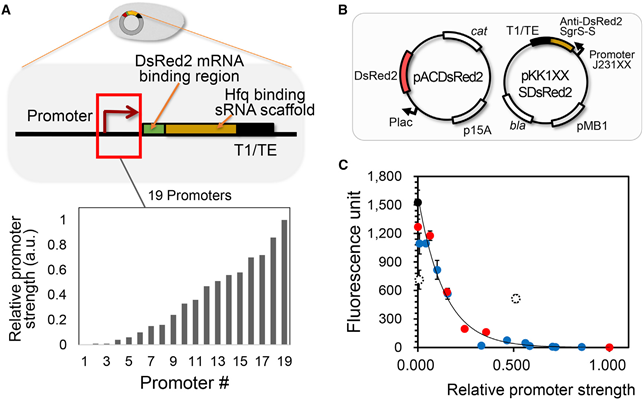
그림 2. (A) 본 연구에서 디자인된 미세 조절 sRNA의 구성. 서로 다른 세기를 가진 프로모터들의 조합을 통하여 target mRNA binding region, scaffold, T1/TE terminator을 포함한 sRNA의 발현량 조절을 시도하였다. (B) DsRed2 형광 단백질의 발현 및 미세 조절 sRNA의 발현을 위한 플라스미드에 대한 모식도. (C) 프로모터 세기에 따른 형광 단백질 발현 억제 정도.
하나의 플라스미드에서 DsRed2라는 형광단백질을 발현하고 다른 플라스미드에서 앤더슨 프로모터 콜렉션이라는 다양한 강도를 지니는 합성 프로모터 시리즈 (MIT Registry, BBa_J23100–BBa_J23118)를 이용하여 DsRed2 mRNA에 결합하는 sRNA의 발현량을 다양하게 변화시켰다. 그 결과 형광단백질 DsRed2의 발현 억제능력의 로그값이 합성 조절 sRNA의 발현량에 비례함을 확인할 수 있었다. 또한, 이는 플라스미드를 이용하여 도입된 유전자의 발현량을 미세조절한 것이므로 세포 내의 염색체에서 발현되는 단백질의 미세조절을 이용하여 합성 조절 sRNA의 발현량 조절을 통한 유전자 미세조절 기법의 성능을 한 번 더 확인하고자 하였다 (그림 3).
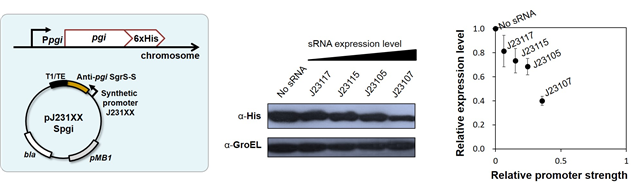
그림 3. 합성 조절 sRNA의 발현량 변화를 이용한 염색체 상의 유전자 pgi에 코딩된
glucose 6-phosphate isomerase의 발현량 변화 확인
대장균의 포도당의 해당작용 진입에 중요한 glucose 6-phosphate isomerase를 코딩하는 pgi 유전자에 6xHis tag을 결합시킨 유전자를 목표로 삼았다. 웨스턴 블랏을 이용하여 합성 조절 sRNA의 발현량 변화에 따른 유전자 발현량의 변화를 확인한 결과 비례관계가 성립함을 확인할 수 있었다. 이로써 합성 조절 sRNA의 발현량을 다양하게 변화시켜 목표 유전자의 발현량을 조절한다는 전략이 예상대로 작동함을 확인할 수 있었다.
□ 합성 조절 sRNA를 이용한 미생물 세포 공장 성능 향상 유전자 동정
위에서 개발된 sRNA의 발현량 변화를 이용한 유전자 발현 미세조절 시스템을 이용하여 대사공학에 적용하는 시도를 하였다. 이를 수행하기 위하여 기존에 본 연구진이 개발한 바 있는 푸트레신 (putrescine) 생산 균주를 이용하였다. 푸트레신 (putrescine)은 나일론 생산의 전구체로 이용되는 물질이며, 다양한 화학제품 및 의약품의 전구체로 활용될 수 있는 가능성이 높아 장차 산업적 사용량이 증가될 추세에 있는 중요한 화합물이다. 본 연구진은 기존 연구를 통하여 24.2 g/L의 농도와 0.75 g/L/h의 생산성의 푸트레신 생산 균주를 제작한 바 있다.
포도당으로부터 푸트레신에 이르는 대사회로와 이들의 경쟁 경로에서 총 13 가지 유전자들을 아래 그림 4와 같이 미세조절을 위한 목표로 선정하였다.
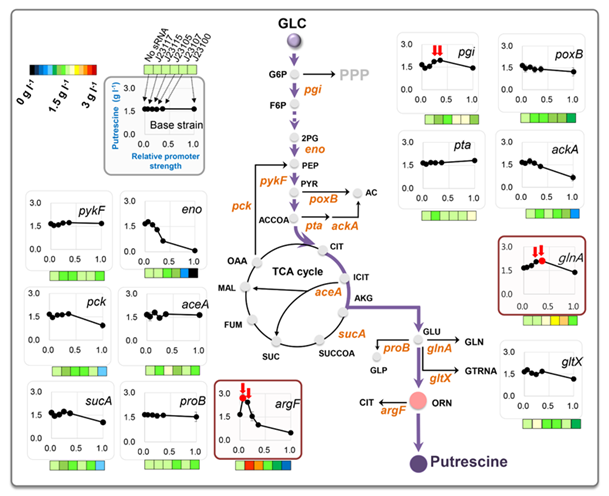
그림 4. 푸트레신 대사경로 관련 유전자들의 5단계 발현 억제를 통한 푸트레신 생산량 향상 억제 목표 유전자 스크리닝. 13 개의 유전자에 대한 sRNA를 5 가지 다른 강도를 지니는 프로모터를 이용하여 발현시켜 다양하게 발현 억제를 수행하였으며, 10% 이상 푸트레신 생산량 향상을 나타내는 균주들은 빨간 화살표로 표시하였다. pgi, glnA, argF의 발현 억제의 효과로 푸트레신 생산이 향상되었다.
총 65 가지 균주에 대하여 푸트레신 생산을 관찰하였으며 이들 중 10% 이상 푸트레신 생산이 향상된 억제 목표 유전자는 3개로 pgi, glnA, argF이다. 특이하게도 argF와 glnA의 너무 약하거나 너무 강한 발현 억제는 푸트레신의 생산량을 증가시키지 못하는 것을 확인할 수 있다. glnA 유전자는 최소배지에서 세포의 성장에 필수적인 필수 유전자로 알려져 있으며, argF는 아르지닌 생산에 관여하는 유전자이며 이와 같은 역할을 하는 유전자인 argI가 세포 내에 본래 존재하나 푸트레신 생산 균주에서는 해당 유전자가 결실되어 있기 때문에 최소배지에서의 세포생장을 위해서는 argF가 반드시 필요하다. 따라서 glnA나 argF의 너무 강력한 억제는 세포의 성장을 지나치게 저해하여 오히려 푸트레신의 생산성을 저해하게 된다. 따라서 이들 유전자의 발현 조절로 푸트레신의 생산성을 향상시키는 것은 반드시 최적화된 유전자 발현 조절을 통해서만 가능하며 이는 합성 조절 sRNA를 이용한 유전자 미세조절 기법의 중요성을 보여준다. sRNA가 발현되지 않는 푸트레신 생산 기본 균주에서는 푸트레신 생산량이 1.68 g/L이며 가장 높은 푸트레신 생산 향상을 보이는 J23117 프로모터로 anti-argF sRNA를 발현하는 균주는 2.71 g/L의 푸트레신을 생산하여 61.3%의 증가를 보였다. 또한 두 번째로 높은 푸트레신 생산 향상을 보이는 목표 유전자인 glnA의 sRNA를 J23107 프로모터로 발현하는 균주는 2.10 g/L의 푸트레신을 생산하여 sRNA가 발현되지 않은 균주 대비 25.0%의 푸트레신 생산 증가를 보였다.
□ 합성 조절 sRNA를 이용한 유가식 발효 기반의 푸트레신 생산
위에서 생산능이 향상된 푸트레신 생산 균주의 배양 스케일을 키웠을 때의 sRNA 효과를 확인하기 위하여 유가식 발효를 진행하였다. 6 L 발효기에 2 L 배지로 발효를 시작하였으며 플라스크 배양 기준으로 탁월한 푸트레신 생산을 보인 두 가지 균주, J23117 프로모터 이용 anti-argF sRNA 발현 균주와 J23107 프로모터 이용 anti-glnA sRNA 발현 균주의 유가식 발효를 수행하였다. 그 결과 두 가지 균주 모두 기존 균주 대비 약 40% 상승한 푸트레신 생산을 보여 약 34 g/L의 푸트레신 생산을 확인할 수 있었다 (그림 5 균주 1~3). 이에 따라서 포도당 사용 대비 푸트레신 수율도 20% 정도 상승하였다. 이들 sRNA들의 조합을 통해서 추가적인 균주의 성능향상을 시도하였다.
위의 두 가지 프로모터-sRNA 조합을 동시에 푸트레신 생산 균주에 적용하였을 때, 한 종류의 sRNA를 발현하였을 때보다 오히려 푸트레신 생산이 감소되는 것을 확인할 수 있었다. 수율과 시간당 생산율도 마찬가지로 감소하였다 (그림 5. 균주 4). 이는 두 가지 필수 유전자의 동시 억제에 따른 세포 성장의 저해로 인한 결과로 보여진다. 이러한 문제를 극복하여 두 가지 sRNA의 발현을 통한 증산효과를 얻어내기 위하여 각 sRNA의 발현을 조절하기로 하였다. 그 결과, 약한 J23105 프로모터로 anti-glnA sRNA를 발현시키고 카피 수가 더 낮은 플라스미드 상에서 앤더슨 프로모터 콜렉션 중 가장 약한 프로모터인 J23112 프로모터로 anti-argF sRNA를 발현시킨 균주의 유가식 발효를 통해 43.0 g/L의 푸트레신을 생산할 수 있었다. 또한 수율과 시간당 생산량도 0.256 g/g과 1.265 g/L/h로 sRNA 발현이 없는 기존 균주대비 대략 60~70%씩 향상된 결과를 보여주었다 (그림 5. 균주 9).
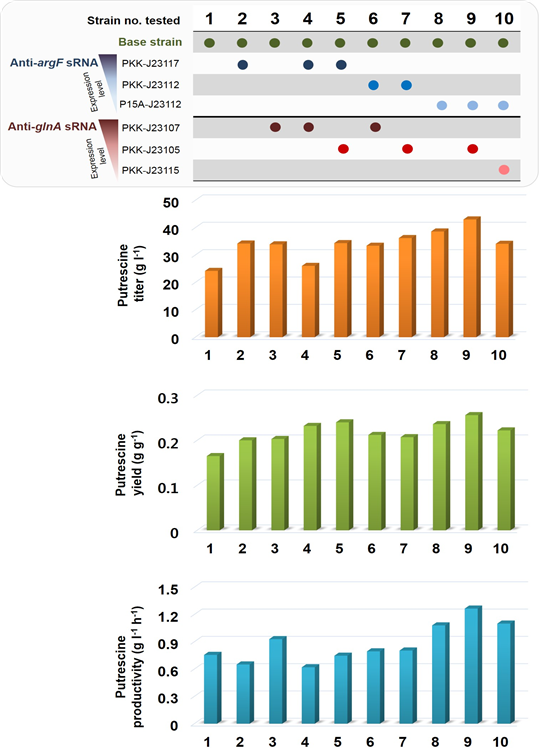
그림 5. 다양한 sRNA의 발현량을 지니는 푸트레신 생산 균주들의 유가식 발효
□ 합성 조절 sRNA를 이용한 프롤린 생산균주 제작
위에서 푸트레신 생산 균주의 성능을 향상시키는 두 가지 유전자를 발견하여 이 둘의 동시 조절을 통해 상승효과를 얻을 수 있음을 확인하였다. 이들 유전자의 미세조절을 이용하여 다른 화합물의 생산에서도 효과를 얻을 수 있는지 확인하기 위하여 푸트레신과 생산 대사 경로를 공유하는 화합물 생산 균주를 제작하고자 하였고, 목표 화합물을 프롤린으로 설정하였다. 이를 위하여 아래 그림 6. (A)와 같이 프롤린 생산 기본균주를 제작하였다.
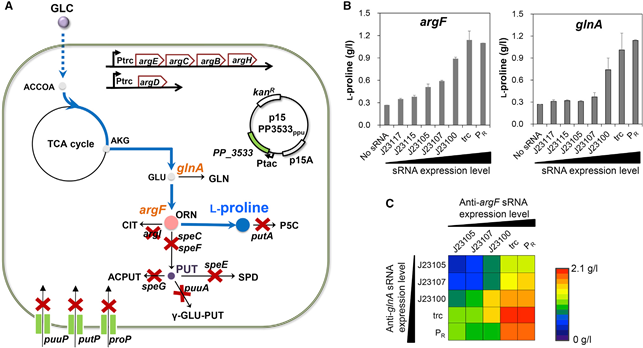
그림 6. (A) 프롤린 생산 균주 제작에 관한 모식도. (B) 서로 다른 세기의 프로모터를 통하여 argF 및 glnA 타겟 합성 조절 sRNA를 발현시킴으로 인한 프롤린의 생산량 추이. (C) glnA와 argF의 발현 억제를 각각 다른 세기의 프로모터의 조합으로 하였을 때 프롤린 생산량 추이.
이렇게 제작된 균주는 플라스크 상에서 0.27 g/L의 프롤린을 생산하였다. 이 균주에 위의 푸트레신 생산 균주에 사용하였던 argF와 glnA의 발현을 억제하는 sRNA를 도입하여 프롤린 생산의 변화를 관찰하였다. 우선적으로 푸트레신 생산 균주에서 사용한 5 가지 프로모터 (J23117, J23115, J23105, J23107, J23100)을 사용하여서 anti-argF sRNA를 발현시켜 프롤린 생산 변화를 확인하였다 (그림 6. (B)). 그 결과 sRNA의 발현량이 강해질수록 프롤린의 생산량이 향상되어 Ptrc 프로모터에 의하여 sRNA가 발현될 때에 1.14 g/L 까지 322% 프롤린 생산이 향상되었다. 위와 비슷한 실험을 anti-glnA sRNA를 이용하여 수행하였다. 이 또한 sRNA의 발현량이 강해질수록 프롤린 생산이 향상되는 결과를 보였으며 그 중에서도 Pr 프로모터로 sRNA를 발현시킬 때에 최대 1.14 g/L의 프롤린 생산을 확인할 수 있었다 (그림 6. (B)). 이 때 두 sRNA를 동시에 적용시킴에 있어서 더 효율적인 방법을 찾고자 in silico 시뮬레이션을 통해서 각 유전자의 발현억제가 어떻게 이루어졌을 때 최적의 상태가 되는지 알아보는 연구를 수행하였다. argF와 glnA는 모두 프롤린 생산경로에 경쟁적인 대사회로에 관여하는 유전자들이기 때문에 이들 유전자를 억제할수록 예측되는 프롤린 생산은 증가하는 추세를 보였다. 그리고 이들 유전자에 의해서 발현되는 효소의 활성이 전혀 없을 때에 프롤린 생산이 최대가 되었다. 하지만 이들 유전자는 세포 성장에 필수적인 유전자들이기 때문에 이러한 예측대로 세포 공장을 제작하는 것은 효율적이지 않다. 따라서 세포의 성장과 프롤린의 생산을 모두 변수에 포함시켜 시뮬레이션을 수행하였다. 그 결과 argF와 glnA의 발현량이 모두 너무 강력하게 억제되지는 않을 때에 생산이 최대가 됨을 알 수 있었다 (그림 7).
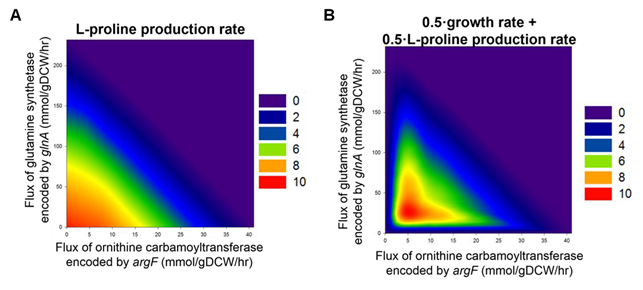
그림 7. argF와 glnA 두 가지 유전자의 발현 정도에 따른 프롤린의 생산과 세포 성장에 대한 in silico 시뮬레이션 결과.
이 두 가지 sRNA의 발현을 J23105, J23107, J23100, Ptrc, Pr 프로모터를 이용하여 다양화시키고, 이들을 조합하여 총 25 개의 균주를 제작하였다 (그림 6 (C)). 그 결과 최소 0.31 g/L에서 최대 1.95 g/L까지 프롤린 생산 농도가 향상되었다. 그 중 최고의 성능을 보인 균주는 위의 두 가지 sRNA를 각기 Ptrc 프로모터를 이용하여 발현시키는 균주로 이는 sRNA발현이 없는 균주 대비 600% 이상 향상된 성능이다. 이 균주 또한 유가식 발효에서도 탁월한 성능을 보일 것이라 기대되어 고농도 세포 배양시에 프롤린 생산을 관찰하였다. 그 결과 34.2 g/L의 프롤린을 생산할 수 있었다. 이 결과는 대장균을 통한 프롤린의 세계최대 생산량이다.
3. 기대효과
기존의 유전자 발현 조절 기술들은 기본적으로 염색체를 조작하여 유전자 발현을 감소시키거나 억제하는 기술들로, 많은 시간과 노력이 소요되었으며, 유전자에 따라서 염색체 상에서 조작함에 따라 심한 부작용을 일으키는 경우에는 염색체 조작이 불가능하였다. 이에 반하여, 합성 조절 sRNA는 플라스미드를 기반으로 하여 3-4일 만에 균주에 적용시킬 수 있다. 따라서 기존의 기법을 사용한다면 균주의 수와 유전자의 종류가 늘어날수록 기하급수적으로 늘어나는 시간과 노력의 소요를 합성 조절 sRNA 기술을 이용하여 수 백, 수 천분의 일로 줄일 수 있는 것이다. 또한, 본 유전자 발현 미세조절 기술을 이용한 맞춤형 sRNA는 학교, 연구소, 병원, 업체, 다국적 제약사 등으로 제품 공급이 가능하며, 유전자 합성 및 활용에 관한 추가 기술이전 사업화의 가능성이 매우 높다고 판단된다. 해당 기술을 통한 합성생물학 산업 확장 효과는 비단 바이오화학에만 한정되지 않으며 대장균을 이용한 화합물의 생산, 단백질 의약품의 생산에도 적용하여 그 가격 경쟁률을 높이고 산업 성장력을 높여 첨단 산업에 있어서의 일자리 창출에 기여할 것이라 예상된다.
참고문헌
1. Na D, Yoo SM, Chung H, Park H, Park JH and Lee SY (2013). Metabolic engineering of Escherichia coli using synthetic small regulatory RNAs. Nat. Biotechnol. 31:2, 170-174.
2. Yoo SM, Na D, and Lee SY (2013). Design and use of synthetic regulatory small RNAs to control gene expression in Escherichia coli. Nat. Protoc. 8: 1695;1707.
3. Qian ZG, Xia XX, and Lee SY (2009). Metabolic engineering of Escherichia coli for the production of putrescine: a four carbon diamine. Biotechnol. Bioeng. 104.4: 651-662





