항생제 생산 최적화를 위한 방선균 유전적 도구 개발
한국과학기술원 조병관 교수
Yi JS, Kim MW, Kim M, Jeong Y, Kim EJ, Cho BK, Kim BG (2016). A novel approach for gene expression optimization through native promoter and 5’ UTR combinations based on RNA-seq, Ribo-seq, and TSS-seq of Streptomyces coelicolor. ACS Synth. Biol. 2016 December DOI: 10.1021/acssynbio.6b00263
1. 연구배경
방선균은 다양한 항생제를 생산하는 미생물 균주로, 지금까지 알려진 자연 발생 항생물질의 70% 이상을 생산한다. 이 중 의학·농업적으로 유용한 항생제 생산을 증가시키기 위해 방선균에서 항생제 생합성 유전자와 조절 네트워크를 조작하려는 시도가 되어 왔는데, 이 과정에서 방선균 유전체를 조작할 때 사용할 수 있는 유전적 도구가 부족하다는 점이 한계로 작용하고 있다. 이를 해결하기 위해 최근에는 마이크로어레이나 RNA 염기서열분석 결과 등 전사물 발현양을 기준으로 몇 개의 프로모터가 개발되었다. 그러나 실제로 생산물의 양은 전사물의 발현량 뿐만 아니라 전사물이 번역되는 효율에 더 직접적으로 영향을 받기 때문에 유전적 도구 개발 시 번역 효율 또한 고려해야 할 필요가 있다. 최근 모델 방선균인 Streptomyces coelicolor에서 여러 차세대 염기서열분석기술(Next-generation sequencing)을 이용하여 유전적 구조 정보와 전사 및 번역 수준에서의 유전자 발현을 관찰함으로써, 전사시작지점의 정확한 위치와 전사물 발현양, 그리고 번역 효율을 전체 유전체 수준에서 밝힌 연구 결과가 있다.
2. 연구내용
본 연구에서는 세 종류의 차세대 염기서열분석 응용기술(전사시작지점 염기서열분석, RNA 염기서열분석, 리보솜 결합 RNA 염기서열분석)을 S. coelicolor에 적용해서 얻은 전체 유전체 수준의 정보를 이용하여, 항생제 생산 증대를 위한 유전자 조작 시 이용할 수 있는 유전적 도구(프로모터, 리보솜결합부위)를 합리적인 디자인을 통해 개발하였다.
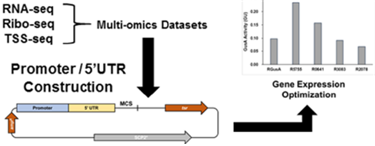
그림 1. 합리적인 디자인을 통한 유전적 도구 개발 과정
전사물 발현양은 전사시작지점의 앞에 존재하는 프로모터 서열에 의해 조절되고, 이 전사물이 번역되는 효율은 5‘비번역부위(untranslated region)에 있는 리보솜결합부위 서열에 영향을 받기 때문에, 다양한 조건에서 지속적으로 유전자 발현을 특정 수준으로 유지할 수 있는 유전적 도구를 선정하기 위해 RNA 염기서열분석 결과와 리보솜 결합 RNA 염기서열분석 결과로부터 프로모터와 리보솜결합부위 후보를 선정하였다.
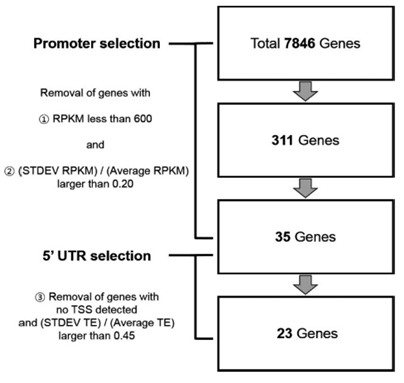
그림 2. 프로모터 및 리보솜 결합 위치 선정 과정
선정된 프로모터와 리보솜결합부위로부터 최종적으로 다양한 유전자 발현 수준을 가진 8개의 조합을 만들었고, 전사시작지점 염기서열분석을 통해 얻은 정확한 전사시작지점 정보를 이용함으로써 프로모터와 5‘비번역부위 사이의 간섭이 최소화 되게 하였다. 이와 같이 디자인 했을 때, RNA 염기서열분석 결과와 리보솜 결합 RNA 염기서열분석 결과로부터 계산된 발현 수준과 리포터 시스템을 이용하여 얻은 실제 실험 결과 간의 상관관계가 높았기 때문에, 실험 결과가 실험 디자인을 잘 반영하는 것을 확인할 수 있었다.
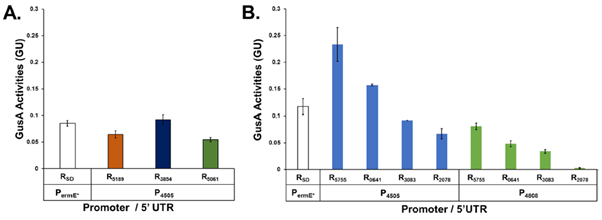
그림 3. 무작위적으로 선정된 리보솜결합부위(A) 또는 합리적으로 선정된 리보솜결합부위(B)와 프로모터 조합 작동 확인
또한 기존에 여러 방선균 종에서 사용되는 프로모터와 본 연구에서 선정한 강력한 리보솜결합부위를 조합했을 때 유전자 발현 수준이 4배에서 11배까지 증가하는 것을 확인함으로써, S. coelicolor 뿐만 아니라 다른 방선균에서도 이 시스템을 적용할 수 있는 가능성을 제시하였다.
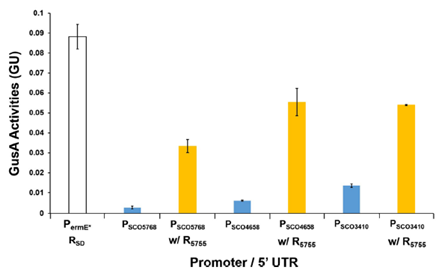
그림 4. 기존에 방선균에서 사용되는 프로모터와 합리적으로 선정된 리보솜결합부위 조합 작동 확인
선정한 프로모터/리보솜결합부위 조합을 항생제 생합성 조절 유전자 actII-orf4에 적용한 결과, 예상한 대로 강력한 프로모터와 강력한 리보솜결합부위 조합이 사용된 균주에서 항생제 생산이 2.6배 증가한 것을 확인하였다. 또한 적정량이 발현될 때 항생제 생산을 증가시킨다고 알려진 metK 유전자에 같은 시스템을 적용했을 때, 오히려 약한 프로모터와 약한 리보솜결합부위 조합에서 가장 많은 항생제가 생산되어서 기존의 정보와 실험 결과가 일치하는 것을 확인하였고, 이 결과는 본 연구에서 개발한 다양한 프로모터/리보솜결합부위 조합이 타겟 유전자를 특정 수준으로 발현시킬 때 유용하게 사용될 수 있음을 보여준다.
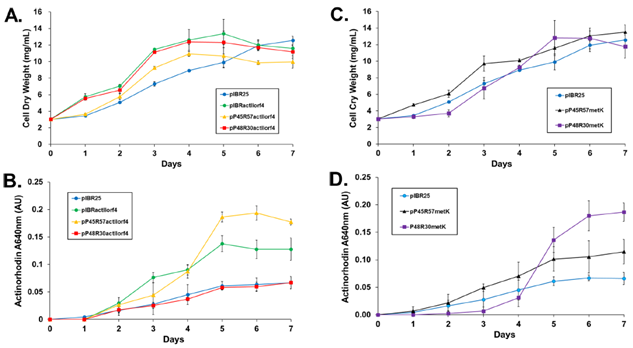
그림 5. 항생제 생합성 유전자 actII-orf4 조작 균주의 생장곡선(A)과 항생제 생산량(B),
ActII-orf4 조절 유전자 metK 조작 균주의 생장곡선(C)과 항생제 생산량(D)
3. 기대효과
본 연구에서 개발한 발현 시스템은 엔지니어링 시 유용한 도구를 제공함으로써 유전적 디자인과 실험적 구현 사이의 갭을 줄일 것으로 기대된다. 또한 본 연구는 전사체 정보에 의존하던 기존의 프로모터 검색 방법을 여러 수준의 차세대 염기서열분석 정보를 이용한 합리적인 디자인 방법으로 발전시켰고, 이는 다른 종에서 다양한 단백질 발현 시스템 구축을 위한 유전자 조작 디자인 시 통찰력을 제공할 것이다.
참고문헌
Jeong Y, Kim JN, Kim MW, Bucca G, Cho S, Yoon YJ, Kim BG, Roe JH, Kim SC, Smith CP, Cho BK (2016). The dynamic transcriptional and translational landscape of the model antibiotic producer Streptomyces coelicolor A3(2). Nat Commun. 2016 Jun 2;7:11605.





