항생제 생합성 방선균 유전자 발현 조절 규명

한국과학기술원 조병관 교수
Y. Jeong, J.N. Kim, M.W. Kim, G. Bucca, S. Cho, Y.J. Yoon, B.K. Kim, J.H. Roe, S.C. Kim, C.P. Smith, and B.K. Cho. (2016) The Dynamic Transcriptional and Translational Landscape of the Model Antibiotic Producer Streptomyces coelicolor A3(2), Nature communications., 7:11605
1. 연구배경
방선균은 전 세계의 항생제 중 70% 이상을 생산하는 미생물로, 의학적으로나 농업적으로 그 중요성이 널리 알려져 있다. 이 중 모델 미생물인 Streptomyces coelicolor는 다양한 항생제 생산에 관여하는 이백여 개의 이차대사 유전자를 가지고 있다 (그림 1). 이러한 대사물의 생합성은 보통 일차대사에서 이차대사로의 생리적인 변화와 형태적인 분화를 동반하고, 이것을 조절하는 것은 전사, 번역, 그리고 번역 후 수준으로 연결되는 조절 메커니즘이다. 그동안 방선균 유전체를 이해하기 위한 유전체 수준의 분석이 이루어져 왔으나, 유전자 발현 및 조절과 관련된 정보가 규명되지 않아 항생제 생산성을 높이는데 걸림돌이 되어 왔다. 또한 항생제 유전자 조절에 대해 분자 수준에서 이해하려는 많은 노력을 통해 유전자 발현에 전사 수준 조절이 중요하다는 것은 알려져 있으나, 전사 후 번역 등 이후 유전자 발현 조절의 중요성은 상대적으로 많이 알려져 있지 않다.
그림 1. Streptomyces coelicolor가 생산하는 이차대사물 구조와 전사 및 번역 수준 발현 패턴
2. 연구결과
본 연구에서는 최근 급속도로 발전하고 있는 차세대 염기서열분석기술(next-generation sequencing)을 기반으로 한 세 종류의 차세대 염기서열분석 응용기술 (TSS-seq, RNA-seq, Ribo-seq)을 S. coelicolor의 성장단계에 따라 적용하고 이를 통합 분석함으로써 일차전사체부터 전사체, 그리고 번역체에 이르는 유전자 발현의 전체 흐름을 밝혀내었다.
단일염기수준의 일차전사체 데이터 분석을 통해 유전자 전사과정의 핵심정보인 전사시작지점 3,570개를 발굴하였고, 이 정보로부터 보존된 프로모터 서열을 찾아낼 수 있었다 (그림 2a). 전체 전사시작지점 중 80%에서 -10 위치의 TANNNT motif가 검색되었고, 59%에서 -35 위치의 NTGACC motif가 검색되었다. 또한 -10 motif는 비교적 일정한 위치에 존재하는 데 비해, -35 motif는 다양한 위치에 존재해서 두 motif 사이의 spacer가 다양한 길이로 나타났다. 기존에 몇몇 시그마 인자가 서로 다른 spacer 길이를 인식하는 것으로 알려져 있었는데, 이 결과 또한 방선균에 존재하는 다양한 시그마 인자를 반영하는 것으로 보인다.
전사시작지점 위치로부터 프로모터뿐만 아니라 번역과정의 핵심정보인 5’비번역부위 (untranslated region) 정보 또한 얻을 수 있었다 (그림 2b). 전체 5’비번역부위 길이를 계산해 봤을 때 중앙값은 44 nt였고, 30에서 39 nt가 가장 높은 비율로 나타났다. 긴 길이의 5’비번역부위는 전사 후 조절을 받을 가능성이 높은데, 413개의 5’비번역부위가 150 nt 이상의 긴 길이를 가지고 있었다. 특이하게도 5’비번역부위가 9 nt 이하인 리더리스 유전자가 전체 중 21%를 차지하고 있었고, 이는 bacteria 중에서 매우 높은 비율에 속한다.
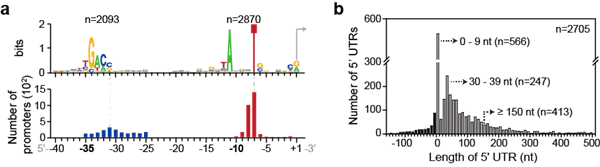
그림 2. 프로모터 서열 및 위치(a)와 5’비번역부위 길이 분포(b)
성장단계에 따른 전사체와 번역체 발현 차이를 시스템 수준에서 비교해 봤을 때, 전체적으로는 전사체와 번역체 모두 성장단계에 상관없이 발현 변화량이 0에 가까운 중앙값을 가졌다 (그림 3). 이차대사 유전자를 따로 확인할 경우 성장이 진행될수록 전사와 번역 수준에서 모두 발현이 증가하는 경향을 보였는데, 전사체 발현이 폭발적으로 증가하는 데 비해 번역체 발현 증가 폭은 일정한 현상을 관찰하였다. 이는 mRNA에서 단백질을 만들어내는 번역속도가 DNA에서 mRNA을 만들어내는 전사속도보다 현저히 느린 번역 버퍼링 (translational buffering) 현상으로 알려져 있으며, 이러한 결과에 미루어 봤을 때 방선균 세포공장 구축 시 전사 수준뿐만 아니라 번역 수준도 반드시 고려해야 할 필요성이 대두된다.
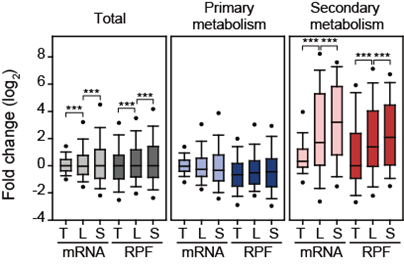
그림 3. 성장단계에 따른 전체 유전자, 일차대사 유전자, 이차대사 유전자의 전사체 및 번역체 발현 변화
전사와 번역 수준 발현양을 통해 번역 효율(translation efficiency)을 계산함으로써 번역 효율이 높은 상위 20% 유전자와 번역 효율이 낮은 하위 20% 유전자를 선정하고, 이것과 일차전사체 정보로부터 발굴한 5’비번역부위와의 관계를 비교해 보았다 (그림 4). 그 결과, 번역 효율이 낮은 유전자들의 5’비번역부위가 더 높은 GC content를 가지고 있었고, 자유에너지는 더 낮은 현상이 관찰되었다. 즉, 5’비번역부위에 더 복잡한 구조를 가질 가능성이 높은 유전자는 mRNA로부터 번역되는 효율이 낮았다. 또한 높은 번역 효율을 가진 유전자는 더 보존된 리보솜결합부위 서열을 가지는 것이 관찰되었고, 결과적으로 5’비번역부위의 서열이 번역 효율에 영향을 미치는 것을 확인하였다.
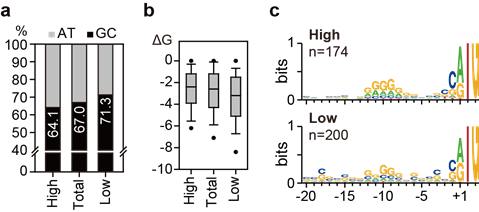
그림 4. 성장단계에 따른 전체 유전자, 일차대사 유전자, 이차대사 유전자의 전사체 및 번역체 발현 변화
전체 이차대사 유전자들의 발현 패턴을 관찰해 본 결과, 28개의 유전자 군집들 중 S. coelicolor의 대표적인 4개 항생제 (CDA, actinorhodin, prodiginine, coelimycin) 유전자 군집이 특히 성장단계에 따라 큰 차이를 보이는 것을 발견하였다 (그림 5). 또한 이 항생제 유전자 군집을 특이적으로 조절하는 유전자들의 발현 패턴을 살펴 보면, 전사물의 양은 성장단계에 따라 꾸준히 증가하는 반면, 번역 수준 발현은 특정 성장단계에서만 급격히 증가하는 현상이 관찰된다 (그림 6). 이는 특정 성장단계에서 항생제 생합성 조절 유전자들이 번역 수준 조절을 받을 가능성을 의미한다.
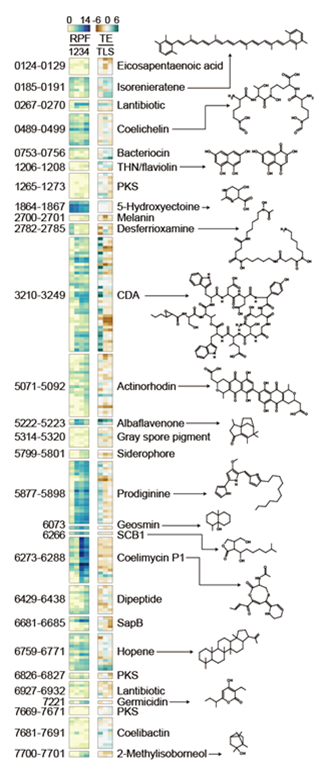
그림 5. 이차대사물 관련 유전자의 번역 수준 발현과 번역효율 분석
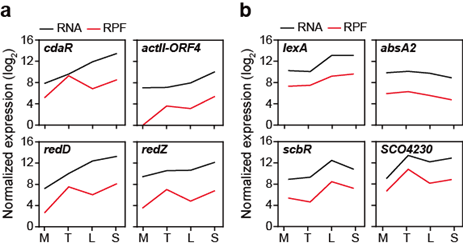
그림 6. 성장단계에 따른 항생제 특이적 조절 유전자(a)와
그 외 이차대사물 관련 조절 유전자(b)의 전사 및 번역 수준 발현 패턴
3. 연구의 성과 및 의의
본 연구에서 다양한 차세대 염기서열분석 기술을 통해 얻은 전체 유전체 수준 정보는 향후 분자 유전학적, 시스템적 연구에 중요한 참고자원으로 사용될 것이다. 다양한 DNA 구조 정보는 방선균 세포공장 구축에 사용 가능하고, 대용량의 전사체 및 번역체 정보는 엔지니어링 타겟 선정에 유용하게 사용될 것으로 기대된다. 또한 본 연구결과는 합성생물학 기술을 이용하여 번역 수준 조절을 받는 항생제 생합성 조절 유전자들을 엔지니어링함으로써 항생제 생산량을 증대시킬 수 있는 가능성을 제시하였다.
참고문헌
1. Nett, M., Ikeda, H. & Moore, B. S. Genomic basis for natural product biosynthetic diversity in the actinomycetes. Nat. Prod. Rep. 26, 1362-1384 (2009).
2. Challis, G. L. Exploitation of the Streptomyces coelicolor A3(2) genome sequence for discovery of new natural products and biosynthetic pathways. J. Ind. Microbiol. Biotechnol. 41, 219-232 (2014).
3. Liu, G., Chater, K. F., Chandra, G., Niu, G. & Tan, H. Molecular regulation of antibiotic biosynthesis in streptomyces. Microbiol. Mol. Biol. Rev. 77, 112-143 (2013).
4. van Keulen, G. & Dyson, P. J. Production of specialized metabolites by Streptomyces coelicolor A3(2). Adv. Appl. Microbiol. 89, 217-266 (2014).
5. Sorek, R. & Cossart, P. Prokaryotic transcriptomics: a new view on regulation, physiology and pathogenicity. Nat. Rev. Genet. 11, 9-16 (2010).
6. Cho, B. K. et al. The transcription unit architecture of the Escherichia coli genome. Nat. Biotechnol. 27, 1043-1049 (2009).
7. Sharma, C. M. et al. The primary transcriptome of the major human pathogen Helicobacter pylori. Nature 464, 250-255 (2010).
8. Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nat. Rev. Genet. 15, 205-213 (2014).
9. Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M. & Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nat. Protoc. 7, 1534-1550 (2012).
10. Schrader, J. M. et al. The coding and noncoding architecture of the Caulobacter crescentus genome. PLoS Genet. 10, e1004463 (2014).
11. McManus, C. J., May, G. E., Spealman, P. & Shteyman, A. Ribosome profiling reveals post-transcriptional buffering of divergent gene expression in yeast. Genome Res. 24, 422-430 (2014).
12. Bentley, S. D. et al. Complete genome sequence of the model actinomycete Streptomyces coelicolor A3(2). Nature 417, 141-147 (2002).
13. Touzain, F. et al. SIGffRid: a tool to search for sigma factor binding sites in bacterial genomes using comparative approach and biologically driven statistics. BMC bioinformatics 9, 73 (2008).
14. Zheng, X., Hu, G. Q., She, Z. S. & Zhu, H. Leaderless genes in bacteria: clue to the evolution of translation initiation mechanisms in prokaryotes. BMC genomics 12, 361 (2011).





