Cross linker를 통해 안정화되는 융합 알파헬릭스 시스템과 이를 통한 두 단백질의 연결

한국과학기술원 이지오 교수
Woo Hyeon Jeong, Haerim Lee, Dong Hyun Song, Jae-Hoon Eom, Sun Chang Kim, Hee-Seung Lee, Hayyoung Lee and Jie-Oh Lee (2016) Connecting two proteins using a fusion alpha helix stabilized by a chemical cross linker. Nature Communications, 7:11031
1. 연구배경
두 개의 단백질을 예측 가능한 단단한 구조로 연결하는 것은 복잡한 단백질 복합체를 만드는데 필수적이다 [1]. 대부분의 화학적 크로스링커들은 표적으로 하는 단백질의 반응 부위에 쉽게 접근할 수 있도록 하기 위해서 길이가 길고 구조적으로 불안정한 링커를 가지고 있다. 따라서 이렇게 만들어진 하이브리드 단백질은 구조적으로 유동적인 경우가 많고, 융합 단백질의 구조를 예측하거나 디자인 하는 것이 불가능하다. 이러한 문제들을 해결하기 위해서 단백질 복합체를 원하는 모양으로 만들 수 있는 여러 가지 새로운 방법들이 시도되고 있다 [2-4].
알파헬릭스는 80% 이상의 단백질이 가지고 있는 이차구조이다. 알파헬릭스는 정확하게 예측이 가능한 구조를 가지고 있으며, 베타 스트랜드에 비하여 독립적인 구조를 가지고 있기 때문에, 비교적 쉽게 디자인이 가능하다 [5]. 따라서, 만약 두 개의 단백질을 알파헬릭스 구조를 가지는 링커로 연결할 수 있으면 융합 후 두 단백질의 상대적인 구조를 정확하게 예측할 수 있을 것이다 (그림 1).
본 연구에서는 두 개의 단백질 내에 존재하는 알파헬릭스를 서로 연결하여 하나의 확장된 알파헬릭스를 만드는 새로운 방법을 제안하였다. 모델 시스템으로 C-말단에 알파헬릭스를 가지고 있는 ankyrin 단백질과 N-말단에 알파헬릭스를 가지고 있는 protein A 단백질을 이용하였고, 이 두 말단에 있는 알파헬릭스를 연결하여 하나의 확장된 알파헬릭스를 만들어서 두 단백질을 연결하였다. 일반적으로 단순한 유전자 클로닝 기법으로는 구조적으로 안정하고 예측 가능한 융합 알파헬릭스를 만들 수 없기 때문에, 우리는 알파헬릭스와 선택적으로 반응하여 안정화 시켜주는 합성 화학물질 EY-CBS (3,3’-ethyne-1,2-diylbis-{6-[(chloroacetyl)amino]benzenesulfonic acid})를 이용하였다. EY-CBS는 알파헬릭스의 i 위치와 i+11 위치에 시스테인이 있을 때 이 두 시스테인과 반응할 수 있다. EY-CBS가 융합 단백질의 형성을 잘 감지할 수 있는지 확인하기 위해, 알파헬릭스의 융합된 부위 내에 서로 11개 아미노산만큼 떨어져 있는 아미노산 두 개를 선택하여 시스테인으로 바꾸었다.
실험결과 성공적으로 융합된 알파헬릭스의 경우에는 EY-CBS와의 반응성이 매우 좋았고, EY-CBS를 통해 두 단백질이 연결될 때 융합 알파헬릭스가 실제 알파헬릭스 구조를 가지도록 도와준다는 것을 발견하였다. 우리가 알파헬릭스를 디자인하는 방법은 복잡한 컴퓨터 시뮬레이션 등을 필요로 하지 않아서 두 단백질을 연결하여 구조적으로 예측 가능한 융합단백질을 디자인하는 데에 손쉽게 이용될 수 있을 것으로 기대된다.
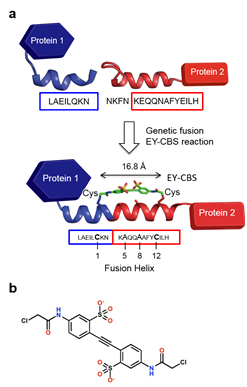
그림 1. 본 연구에서 제안한 cross linker를 통해 안정화되는 융합 알파헬릭스 시스템 디자인 모식도. 그림 a는 한 단백질의 C-말단 헬릭스와 다른 단백질의 N-말단 헬릭스를 유전적 융합하는 방법과 EY-CBS와의 반응을 위하여 시스테인으로 치환하는 방법을 보여주며, 그림 b는 EY-CBS의 화학 구조를 보여준다. EY-CBS는 시스테인의 thiol 그룹과 반응할 수 있다.
2. 연구결과
융합 헬릭스 방법을 테스트하기 위한 모델 시스템으로 말토스-바인딩 단백질 (MBP)에 바인딩하는 ankyrin 단백질과 protein A 단백질의 B4 도메인을 이용하였다. 이 두 단백질은 알파헬릭스로 이루어져 있고 시스테인이 없기 때문에 모델 시스템으로 적당한 단백질로서 선택되었다. ankyrin 의 C-말단 알파헬릭스와 protein A 의 N-말단 알파헬릭스를 융합하는 방법으로 총 17개의 융합 단백질을 만들었다. ankyrin과 protein A의 알파헬릭스에서 각각 한 개씩의 아미노산을 선택하되 이 두 아미노산은 11개 아미노산만큼 떨어져 있는 i, i+11 자리의 아미노산으로 선택해서 각각을 시스테인으로 치환하여 EY-CBS와 반응할 수 있도록 하였다.
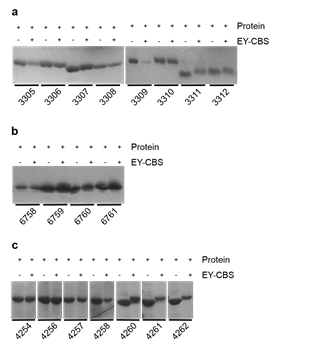
그림 2. 퓨전 단백질의 EY-CBS 반응성 비교. 그림 a, c는 protein A와 ankyrin 융합 단백질의 반응성을 보여주며, 그림 b는 protein A와 calmodulin 융합 단백질의 반응성을 보여준다.
또한 EY-CBS 반응시 긴 아미노산 잔기에 의한 공간적 충돌을 막기 위해서 i+4, i+7 자리의 아미노산은 길이가 짧은 알라닌으로 치환하였다. 같은 방법으로 protein A 와 calmodulin N-말단 도메인을 이용하여 4개의 융합 단백질을 만들었다.
정제한 융합 단백질은 1mM EY-CBS와 반응하였다. SDS-PAGE 분석 결과 총 21개의 융합 단백질 중 5개의 융합 단백질의 밴드가 위로 이동하는 것을 확인하였다. 이러한 밴드 이동은 결합한 EY-CBS에 따른 사이즈 변화에 의한 것뿐만 아니라 시스테인을 통해 융합 단백질이 형성되면서 융합 알파헬릭스 구조로의 변화로 인한 것임을 확인하였다.
우리는 EY-CBS와의 반응성이 가장 좋았던 3311 융합 단백질을 선택하여 세부 실험을 진행하였다. 3,311 융합 단백질이 EY-CBS와 반응하였음을 정확히 확인하기 위하여 흡수 스펙트럼을 측정하였다. EY-CBS는 340 nm에서 흡수 피크를 보이는데, EY-CBS와 반응한 3,311 융합 단백질 또한 340 nm에서 강한 흡수 피크를 보였다. 이로 통해 3,311 융합 단백질은 EY-CBS와 결합하였음을 확인할 수 있었다. 또한 pH에 따른 EY-CBS의 반응성을 확인해본 결과, pH 8에서 거의 대부분 EY-CBS 반응이 가는 것을 확인하였다.
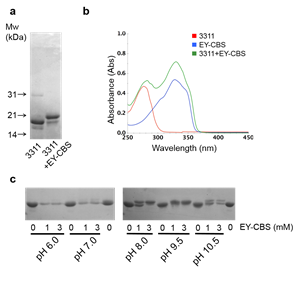
그림 3. 융합 단백질 3,311의 EY-CBS 반응성. 그림 a는 융합 단백질 3,311의 EY-CBS 반응성을 보여주며, 그림 b는 EY-CBS와 반응한 3,311의 흡수 스펙트럼을 보여주고, 그림 c는 pH에 따른 EY-CBS 반응성을 보여준다.
융합 알파헬릭스가 잘 합성되었는지 확인하기 위해서 EY-CBS와 반응하기 전과 후의 3311 융합 단백질을 각각 결정화하여 그 구조를 확인하였다. 전자 밀도를 통해 예상한 대로 단백질의 융합 알파헬릭스 부위에 하나의 EY-CBS가 반응하였음을 확인할 수 있었다. 융합 알파헬릭스가 거의 완전한 알파헬릭스 모양을 갖추고 있음도 확인하였다. ankyrin과 protein A 단백질의 구조에는 변화가 없었는데 이로써 융합 알파헬릭스가 융합한 헬릭스 위치 이외에는 구조적으로 영향을 주지 않음을 알 수 있다.
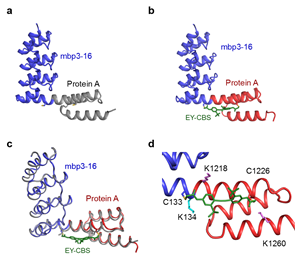
그림 4. 융합 단백질 3311의 EY-CBS 반응 후의 결정 구조. 그림 a는 EY-CBS와 반응한 3311이 융합 알파헬릭스를 형성시에 예상하는 결정 구조이며, 그림 b는 EY-CBS와 반응한 3,311 융합 단백질의 실제 구조이다. 그림 c는 EY-CBS와 반응한 3,311 융합 단백질의 예상하는 구조와 실제 구조가 거의 비슷함을 보여주고, 그림 d는 융합 알파헬릭스 부위의 자세한 모습이다.
반응하지 않은 3311 융합 단백질의 결정 구조 또한 확인해 보았는데 이를 통해 3,311 단백질이 EY-CBS와 왜 강한 반응성을 보이는지를 이해할 수 있었다. 반응하지 않은 3,311 융합 단백질은 ankyrin 단백질과 protein A 단백질이 20도 정도로 살짝 휘어진 알파헬릭스로 연결되어 있음을 확인하였다.
이를 통해 왜 3311이 강하게 EY-CBS와 반응하는지를 알 수 있었다. 3,311의 융합 알파헬릭스는 EY-CBS와의 반응 이전에 이미 형성되어 있을 것으로 생각된다. 융합 알파헬릭스는 EY-CBS와의 반응 후 더욱 실제 알파헬릭스의 구조를 갖추게 되는 것으로 보인다. 이는 EY-CBS 반응이 알파헬릭스에 치환되어 존재하는 두 시스테인 간의 거리가 16.8 옹스트롱이 되도록 도와주기 때문이다.
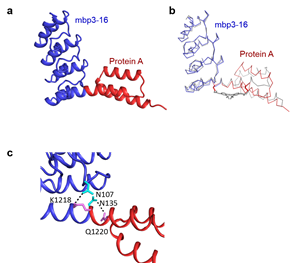
그림 5. 반응하지 않은 융합 단백질 3311의 결정 구조. 그림 a는 3,311의 융합 알파헬릭스가 20도 정도 휘어져 있음을 보여주고, 그림 b는 EY-CBS와 반응한 3311과 반응하지 않은 3311의 구조를 겹쳐서 비교한 그림이다. 그림 c는 반응하지 않은 융합 단백질 3311의 융합 알파헬릭스 부위의 자세한 모습이다.
21개의 융합 단백질 중 EY-CBS에 반응하지 않은 16개의 융합 단백질들은 대부분이 구조적인 불안정성으로 인해 결정화할 수 없었고 6761 융합 단백질만 유일하게 결정화에 성공하여 그 구조를 확인할 수 있었다. 6761 융합 단백질은 protein A 단백질과 calmodulin 단백질이 융합된 형태이다. 6761 은 두 개의 알파헬릭스가 하나의 연결된 알파헬릭스를 형성하지 못하고 있음을 확인하였다. EY-CBS와의 반응을 위해 치환한 두 개의 시스테인이 가까이 존재하면서 이황화 결합으로 연결되어 있는 예상치 못한 구조를 확인하였다. 두 알파헬릭스가 이황화 결합으로 연결된 예상 외의 구조로 인해 6761 융합 단백질은 단단한 구조를 가질 수 있었고 결정화될 수 있었던 것으로 생각되어진다.
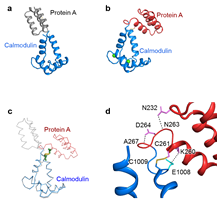
그림 6. 6761 융합 단백질의 결정 구조. 그림 a는 원래 예상하던 6761 융합 단백질의 구조를 보여준다. protein A 와 calmodulin이 하나의 확장된 알파헬릭스로 연결되어 있다. 그림 b는 6761의 실제 결정 구조이다. 치환된 시스테인을 통해 예상하지 못했던 이황화 결합이 형성되어 있음을 보여준다. 그림 C는 예상하던 6761의 구조와 실제 구조를 겹쳐놓은 모습이며 그림 d는 6761의 융합 알파헬릭스 부위의 자세한 모습이다.
이를 통해 두 개의 알파헬릭스를 단순히 클로닝 기법으로 연결하는 것은 하나의 확장된 알파헬릭스를 형성할 수 없음을 알 수 있었다. 그러나 EY-CBS에 높은 반응성을 보이는 융합 알파헬릭스의 경우에는 하나의 확장된 알파헬릭스가 형성되었을 것으로 생각할 수 있고 이는 EY-CBS와의 반응성이 융합 알파헬릭스 형성의 지표가 됨을 말해준다.
3. 연구의 성과 및 의의
본 연구에서는 두 개의 알파헬릭스를 이어서 하나의 긴 확장된 융합 알파헬릭스를 만드는 새로운 방법을 개발하였고 크리스탈 구조분석을 통하여 융합 알파헬릭스 형성이 가능함을 보여주었다. 반응 전에는 휘어져 있었던 융합 알파헬릭스가 EY-CBS와의 반응 후에는 거의 이상적인 구조의 알파헬릭스로 성공적으로 변형되었다. 알파헬릭스는 구조적으로 단단하기 때문에 알파헬릭스로 연결된 융합 단백질은 구조적으로 예측 가능하였다. 이 EY-CBS를 이용한 융합 알파헬릭스 방법은 두 단백질을 원하는 구조를 가지도록 서로 연결하는 데 폭넓게 활용될 수 있을 것이다. 또한 융합하는 두 단백질의 기하학적인 배열에 따라 다양한 인공적인 구조 형성이 가능해지기 때문에 복잡하고 안정된 구조의 융합단백질을 디자인 할 수 있을 것이다.
참고문헌
1. Lai, Y. T., King, N. P. & Yeates, T. O. Principles for designing ordered protein assemblies. Trends Cell Biol. 22, 653-661 (2012).
2. Radford, R. J., Nguyen, P.C., Ditri, T. B., Figueroa, J. S. & Tezcan, F. A. Controlled protein dimerization through hybrid coordination motif. Inorg. Chem. 49, 4362-4369 (2010).
3. Brodin, J. D. et al. Metal-directed, chemically tunable assembly of one-, two- and three-dimentional crystalline protein arrays. Nat. Chem. 4, 375-382 (2012).
4. King, N. P. et al. Computational design of self-assembling protein nanomaterials with atomic level accuracy. Science. 336, 1171-1174 (2012).
5. Padilla, J. E., Colovos, C. & Yeates, T. O. Nanohedra: using symmetry to design self assembling protein cages, layers, crystals, and filaments. Proc. Natl Acad. Sci. USA 98, 2217-2221 (2001).





