Antisense RNA 기반 정족수인식 차단효소 고속다중선별 시스템

전남대학교 김근중 교수
Sang-Soo Han, Won-Ji Park, Hak-Sung Kim and Geun-Joong Kim (2015) Antisense RNA-based High-Throughput Screen System for Directed Evolution of Quorum Quenching Enzymes. ACS Chemical Biology, 10, 2598-2605.
1. 연구배경
많은 미생물들이 세포농도 의존적으로 특정한 유전자의 발현을 조절하기 위한 정족수 인식 (QS, quorum sensing) 기작을 지니고 있다. 알려진 바에 의하면, 독소, 생물발광, 생체막 (biofilm) 생성조절은 물론, 운동성이나 포자형성, 플라스미드의 이동에까지 정족수 인식 기작이 관여하고 있다 [1]. 이러한 과정에 신호를 전달하는 물질로는 acyl-homoserine lactone (AHL), 4,5-dihydroxy-2-cyclopenten-1-one (DHCP), 4-hydroxy-5-methyl- furan-3-one (HMF), 그리고 다양한 peptide 가 알려져 있다. 특히 AHL 을 신호물질로 이용한 정족수 인식시스템은 많은 병원균에서 발견되어지며, 이들 신호를 통해 독소를 생성하거나 생체막을 만들어 약물에 대한 저항성을 보이기도 한다. 이러한 기작은 인간을 비롯한 다양한 동식물에 매우 위험한 감염병 (infection disease) 의 치료를 어렵게 하고 있다. 따라서 QS 기작을 지닌 병원균이 지닌 다재약물 저항성을 극복할 수 있는 새로운 항생제의 개발이 시급한 실정이다.
병원균이 지닌 정족수 인식 기작이 규명된 이후, 신호물질을 분해해 인식기작을 차단하는 효소 (QQ, quorum quenching enzyme) 의 활성으로 독소의 생산이나 병원성을 제어할 수 있는 새로운 개념의 항생제로 이용하기 위한 노력이 진행중이다 [2,3]. 이는 QQ 효소에 의해 생성이 제어되는 생체막이 지닌 약물저항성이 감소된 결과에 기인한다. 현재까지 다양한 미생물로부터 관련 활성을 지닌 효소들이 발굴되었고, 활성을 증진시키기 위한 개량이 진행중이다 [4]. 하지만 신호전달 물질에 대한 낮은 특이성과 활성으로 인해 실응용이 가능한 효소의 개발은 아직 이루어지지 않고 있다.
QQ 효소의 개량과정에서는 정밀하고 재현성있게 활성을 측정할 수 있는 선별방법이 매우 중요하다. 현재까지 LuxI 에 의해 합성된 QS 신호전달 물질인 AHL 과 결합하여 전사를 유도하는 LuxR 을 이용하는 선별법이 주로 보고되었고, 선별법을 보다 선택적으로 구분하기 위해 항생제 마커활성과 접목시키는 방법도 제안되었다 [5]. 하지만 leaking에 의한 기저 수준 (basal level) 이상의 발현과 높은 빈도로 확인되는 false-positive clone 의 비율은 좀 더 정확하고 재현성있는 선별방법이 필요함을 제시하고 있다.
본 연구에서는 QQ 효소 선별과정에 효율적으로 활용할 수 있는 antisense RNA 기반의 고속다중선별 시스템을 제안하였다 (그림 1). 잘 알려진 antisense RNA 인 RyhB 와 이에 의해 발현이 조절되는 sense RNA, 즉 sodB를 리포터 유전자와 융합해 AHL 농도 의존적으로 발현이 유도되도록 설계함으로써 리포터 활성(형광)에 따라 재현성있고 정확하게 QQ 효소활성을 측정하는 방법이다.
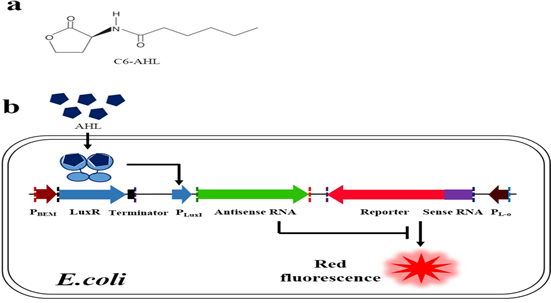
그림 1. 본 연구에서 제안한 antisense/sene RNA 기반의 QQ 효소 활성 측정시스템 구동 모식도. 그림 a는 전형적인 AHL 구조를 보여주며, b 는 AHL 과 결합한 luxR 에 의해 전사가 유도된 antisense RNA 에 의해 번역이 조절되는 sense RNA-reporter 융합 유전자의 발현기작이다.
설계된 QQ 효소 선별방법의 경우, 번역수준에서 리포터 발현을 조절하기 때문에 반응시간이 빠르고 false-positive 비율이 상대적으로 낮은 결과를 보여주었다. 따라서 비교적 간단한 선별과정에 의해 기존효소에 비해 catalytic efficiency 가 증가한 효소를 쉽게 선별하였다.
2. 연구결과
우선 AHL 농도 의존적으로 리포터의 발현을 번역수준에서 조절할 수 있는 antisense/sense RNA 쌍을 발굴하였다. 이를 위해 다양한 문헌과 선행연구결과에서 보고된 관련 유전정보들을 분석 한 후, 비교적 잘 알려진 기작과 정보를 지닌 RyhB 와 sodB 를 후보로서 선정하였다. 선정된 antisense RNA 인 RyhB 의 경우, 대장균에서 발견되어지고 생리적으로는 다양한 target 의 발현양을 조절하는 것으로 알려져 있다. 이중 상호작용이 상대적으로 강하고 관련된 유전자의 생리기능이 잘 알려진 sodB 를 sense RNA 로 결정하였다. Antisense/sense RNA 쌍의 상호작용은 숙주내의 다른 유전자들과 간섭과 교란이 적어야 하고 결합에 필요한 충분한 구조를 확보해야 한다. 이를 위해 특정한 유전자의 발현이 제한적인 숙주를 선정한 후, 이차구조 예측 프로그램를 활용하여 적절한 위치의 상호작용 영역을 설계하였다. 준비된 antisense/sense RNA 쌍의 주형을 대장균 염색체로부터 PCR로 증폭하여 설계요소와 접목한 후, 각각을 과발현시켜 대장균생리에 미치는 영향을 분석하였으나 성장속도나 표현형질에는 큰 문제를 유발하지 않았다.
상기 과정에서 준비된 antisense/sense RNA 쌍의 조절능력을 확인하기 위해, antisense RNA 인 RyhB 를 pBAD 벡터에 클로닝하고, sense RNA 인 sodB를 GFPuv 리포터와 융합하여 lac promter 를 지닌 pMAL 벡터에 클로닝하였다. 그림 2에서와 같이, 충분한 정도로 발현이 유도된 sense RNA-리포터 유전자의 발현양이 arabinose 에 의해 조절된 발현양을 지닌 antisense RNA 에 의해 농도 의존적으로 조절되어짐을 확인하였다.
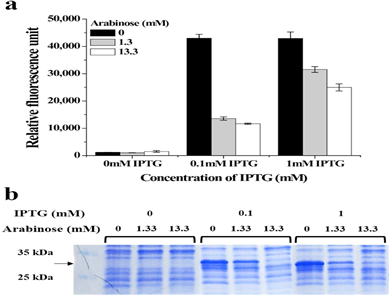
그림 2. Antisense/sene RNA module 에 의한 리포터 유전자의 발현조절. 그림 a는 antisense RNA 의 발현양에 의해 농도 의존적으로 조절되는 리포터 형광을 나타내며, b는 SDS-PAGE를 이용해 실제 리포터 단백질의 발현양을 모니터링한 결과이다.
선정된 antisense/sense RNA 쌍의 기능성과 조절능력을 이용하여, 본 연구의 목적인 QQ 효소활성을 정확하고 재현성 있게 검출할 수 있는 선별시스템을 설계하였다. 그림 1에 도식화된 선별시스템을 포함한 pASRR 벡터의 경우, 항시발현되는 LuxR 단백질이 AHL 과 결합하여 농도 의존적으로 antisense RNA 의 발현을 유도하면, 충분한 양으로 항시발현되는 sense-reporter 전사체의 번역(translation)양을 조절하는 원리에 의해 작동한다. 이때 선별을 위해 조사하는 excitation 파장에 의한 숙주의 damage 를 최소화하기 위해 리포터 단백질을 mCherry 로 변경하였다. 제작된 선별시스템의 AHL 농도 의존적인 리포터 발현양을 확인한 결과, 그림 3a 에서와 같이 pASRR 벡터를 지닌 숙주는 배지에 첨가된 AHL 농도에 의존적인 형광을 보여주었다. 세포양이나 배양배지, 온도, 측정기기에 따른 편차를 줄이기 위한 다양한 선별조건을 최적화하면 특정한 AHL 농도에서의 배양과정에서는 물론, 전배양한 세포를 센서로 이용하는 과정에서도 동일한 감도와 재현성을 보여주었다.
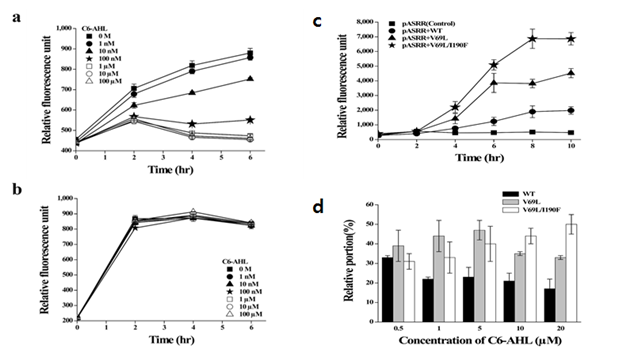
그림 3. AHL 농도 의존적으로 작동하는 선별시스템의 반응성과 QQ 효소활성에 따라 정확하고 재현성 있게 발현되는 리포터 형광 측정결과. 그림 a 는 배지에 첨가된 AHL 농도에 의존적인 리포터 형광을 보여주며, b 는 LuxR을 지니지 않은 벡터 시스템 (pControl)을 지닌 세포 배양배지에 AHL을 첨가한 결과이다. 그림 c 에서는 실제 QQ 활성을 지닌 유전자를 동시에 형질전환한 숙주들이 보여 주는 활성에 따른 형광을 보여주며, d 에서는 알려진 활성을 지닌 QQ 효소 선별과정에서의 false-positive ratio 을 확인한 결과이다. False-positive 비율은 선별된 클론이 지닌 실제 활성을 유전자와 단백질 수준에서 재검사하는 방법으로 결정하였다.
구동성과 반응성을 확인한 선별시스템 pASRR을 지닌 숙주를 이용하여 QQ 효소활성에 따른 선별능력을 확인하였다. 이를 위해 이미 알려진 활성을 지닌 QQ 효소 야생형과 상대적으로 높은 활성을 지닌 변이(mutant) 유전자를 지닌 벡터를 동시 형질전환한 후, 리포터 발현양를 확인하였다. 그림 3c 에서와 같이, 선별시스템을 지닌 숙주의 경우 QQ 효소활성에 따라 뚜렷한 차이를 보이는 형광을 보여주었다. 활성 측정에 필요한 세포배양은 96 well plate에서 가능할 정도로 정확했으며, 물리적인 배양환경에 따른 선별능의 차이는 크게 나타나지 않았다. 반복실험을 통해 리포터의 형광에 따라 선별된 클론을 유전자와 단백질 수준에서 재검토한 결과, 기존 시스템에 비해 월등히 향상된 신뢰도 (낮은 false-positive 비율) 를 보여 주었다.
제작된 QQ 효소 선별시스템과 확립된 대용량선별법을 이용하여, 본 연구의 최종 목표인 활성이 증대된 변이효소를 라이브러리보부터 신속하게 선별하는 연구를 진행하였다. 선별의 대상이 되는 QQ 변이효소 라이브러리는 야생형 AHL lactonase (AiiA)를 주형으로 무작위 돌연변이법과 DNA shuffling 법을 접목해 제작하였다. 제조된 변이유전자를 클로닝한 벡터를 pASRR 벡터를 지닌 숙주로 형질전환해 라이브러리를 제작한 후, 확립된 조건에 따라 선별을 진행하였다. 총 3회의 변이제작과 선별과정을 거쳐 야생형 단백질에 비해 31배의 향상된 catalytic efficiency (Kcat/Km)를 보이는 변이효소를 발굴하였다 (표 1).
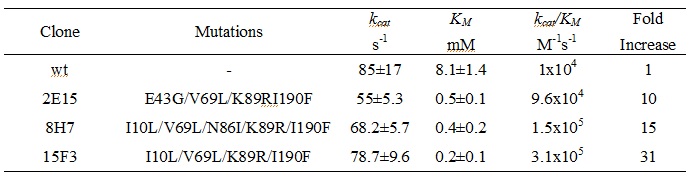
표1. 새로운 선별시스템을 이용하여 선별된 QQ 변이 효소의 반응특성
결과적으로, 기존의 알려진 선별법에 비해 높은 신뢰도와 감응성, 낮은 기저수준의 발현양과 false-positive ratio 을 지닌 선별시스템을 실응용이 가능한 수준으로 구축하였음을 확인하였다. 따라서 기존의 QQ 효소 선별시스템이 지닌 단점의 대부분을 해결하였다.
3. 연구의 성과 및 의의
본 연구에서는 정확하고 재현성있게 AHL 농도를 측정할 수 있는 antisense/sense RNA 기반의 발현조절논리를 정족수 인식시스템과 접목시켜, 정족수인식 차단효소(Quorum Quenching enzyme)를 대용량으로 쉽게 선별할 수 있는 시스템을 개발하였다. 제작된 시스템 자체는 다양한 생태계로부터 AHL (유도체도 가능)를 생산하거나 분해하는 효소를 지닌 미생물을 선별하는데 직접 이용할 수 있다. 또한 본 연구 예에서와 같이, 관련된 효소원의 인공개량과정에서도 효율적으로 활용할 수 있다. 부수적으로, 합성생물학적 연구 부품이 지닌 확장성이나 접목수월성을 고려하면, antisense/sense RNA 쌍이나 전사유도인자들을 다른 목적으로 활용하는 것도 가능하다. 따라서 효소활성이나 이를 저해하는 물질을 발굴하는데 폭 넓게 활용할 수 있을 것이다.
참고문헌
1. Miller, M. B., and Bassler, B. L. (2001) Quorum sensing in bacteria, Annu Rev Microbiol 55, 165-199.
2. Van Gennip, M., Christensen, L. D., Alhede, M., Phipps, R., Jensen, P. O., Christophersen, L., Pamp, S. J., Moser, C., Mikkelsen, P. J., Koh, A. Y., Tolker-Nielsen, T., Pier, G. B., Hoiby, N., Givskov, M., and Bjarnsholt, T. (2009) Inactivation of the rhlA gene in Pseudomonas aeruginosa prevents rhamnolipid production, disabling the protection against polymorphonuclear leukocytes, APMIS 117, 537-546.
3. Skindersoe, M. E., Alhede, M., Phipps, R., Yang, L., Jensen, P. O., Rasmussen, T. B., Bjarnsholt, T., Tolker-Nielsen, T., Hoiby, N., and Givskov, M. (2008) Effects of antibiotics on quorum sensing in Pseudomonas aeruginosa, Antimicrob. Agents. Chemother 52, 3648-3663.
4. Cegelski, L., Marshall, G. R., Eldridge, G. R., and Hultgren, S. J. (2008) The biology and future prospects of antivirulence therapies, Nat. Rev. Microbiol.6, 17-27.
5. Kim, J. H., Lee, S. C., Kyeong, H. H., and Kim, H. S. (2010) A genetic circuit system based on quorum sensing signaling for directed evolution of quorum-quenching enzymes, Chembiochem 11, 1748-1753.





