대장균 총유전체내의 ArgR 전사인자와 DNA 결합체의 다양한 결합 구조의 규명

한국과학기술원 조병관 교수
The architecture of ArgR-DNA complexes at the genome-scale in Escherichia coli. Nucleic Acids Res. 2015. 43(6):3079–3088
1. 연구배경
전사인자는 일반적으로 프로모터 부근의 특수한 서열의 DNA를 인지함으로써, 프로모터를 인지하는 RNA 중합효소를 다양한 외부의 조건변화에 따라 활성 혹은 억제함으로 총체적인 유전체의 발현을 조절하는 중요한 인자이다. 또한, 전사인자는 합성생물학 gene circuit 구축 등 다양한 분야에 이용되고 있어서 전사인자에 의해 인지되는 DNA 의 서열 및 위치를 파악하는 것은 매우 기본적이며 선행되어야 할 중요한 연구이다. 일반적으로 대장균내의 전사인자는 12bp~30bp의 길이의 DNA에 결합하며, 종종 반복되거나 역보상성 염기서열 (palindrome)을 인지하는 특이성을 보이기도 한다. 특이하게 박테리아 내에서 cAMP receptor protein (CRP)이나 arginine repressor (ArgR) 과 같은 전사인자는 DNA 와 결합 시 DNA 구조를 휘게 하는 역할을 함으로써 전사를 조절할 뿐만 아니라 진핵세포에서 DNA 의 packing을 담당하는 chromatin 과 같은 역할 또한 하고 있다.
L-Arg 과의 결합을 통해 전사를 조절하는 ArgR 단백질은 6개 단백질의 복합체로 이루어져 있으며, 역보상성 염기서열을 인지하며, 결합 부위를 중심으로 약 70˚~90˚ 가량 휘는 현상을 보인다. ArgR 의 결합 특이성을 알기 위해 생화학적 방법이나 NMR 과 같은 구조분석학적 접근이 많이 시도되었으나, 이 방법은 in vitro 에서의 특수한 조건에서, 정해진 서열의 DNA와의 분석만이 가능했기 때문에 전체 유전체에서의 메커니즘을 이해하는 데에는 충분하지 않다. 이를 해결하고자, in vivo 내의 총 유전체내의 ArgR 의 결합부위를 규명하기 위해 크로마틴 침전법에 기반한 ChIP-chip 방법이 시도되었고, 실제 거시적인 관점에서의 결합 위치가 파악되었다. 그러나 약 1kb 의 분해능의 한계 때문에 정확한 결합부위만을 파악하는데 한계가 있다.
최근 DNA footprinting 과 같은 생화학적 방법 이상의 고분해능을 보이면서 총유전체의 결합을 한번에 분석할 수 있는 ChIP-exo 방법이 개발되었다. 이는 기존의 크로마틴 침전법 (ChIP) 방법에 기반을 두면서, 추가로 exonuclease를 처리함으로 결합하는 위치의 고분해능 분석이 가능하다. 연이어 이를 차세대 고속 대량 시퀀싱 기술을 이용해 결합하는 부위의 DNA 의 서열을 정확하게 시퀀싱함으로써 실제 총 유전체의 결합위치와 결합 하는 서열을 단일 염기 분해능으로 분석할 수 있도록 한다.
본 연구에서는 이러한 ChIP-exo 방법을 사용하여, ArgR 의 총유전체에서의 결합 부위를 규명하였고, 각각의 유전자에 대한 차별적인 결합 구조를 제시하였으며, 마지막으로 RNA polymerase 와의 연계성을 밝혔다.
2. 연구결과
ArgR 의 유전체에서의 인지 위치를 파악하기 위해 ChIP-exo를 수행하였다. 우선 대장균 게놈의 argR 의 c’ 말단에 8개의 myc을 삽입하였고, 세포내에서 crosslinking을 시킨후 myc 항체를 사용하여, ArgR을 크로마틴 침전시킨후, exonuclease 로 결합하지 않는 부위를 모두 제거 시키고, 이후 결합부위의 말단에 아답터를 결합시키고, 마지막으로 차세대 시퀀싱을 진행하였다 (그림 1a). 그 결과 ChIP-exo를 통하여, 4개의 새로운 결합부위와 함께, 총 62개의 결합부위를 찾아냈다 (그림 1b). Arginine 의 첨가 여부에 따라 총 유전체에 대한 결합 영역은 그림 1c 에 나타내었고, 특히 잘 알려진 aroP와 hisJ 그리고 argD 에 대한 결합을 살펴보았다 (그림1c-d). 특이적으로 이 3가지의 유전자들에 대한 결합 타입이 각각 1, 2, 3개의 고유의 peak-pair (sense-antisense)를 가지고 있음을 관찰하였다.
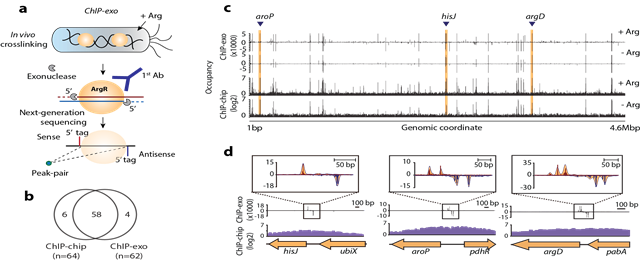
그림 1. ChIP-exo 방법을 사용한 대장균내 ArgR 의 결합 부위의 분석 및 다양한 결합방법의 발견.
이러한 다양한 peak-pair의 분포도를 총유전체에 대하여 살펴본 결과, 1개의 peak-pair (I)는 21개, 2개의 경우 (II)에는 25개, 3개의 경우(III)에는 17개로 다양하게 존재함을 알 수 있었다(그림 2a). 이들의 각각의 peak-pair 들끼리의 peak을 합산 한 결과 peak 간의 거리, peak-pair 간의 거리등에서 동일한 패턴을 보였고 (그림 2b-c), 특이적으로 각각의 peak-pair는 모두 ~91bp 의 동일한 거리를 나타냄을 알 수 있었다 (그림 2d).
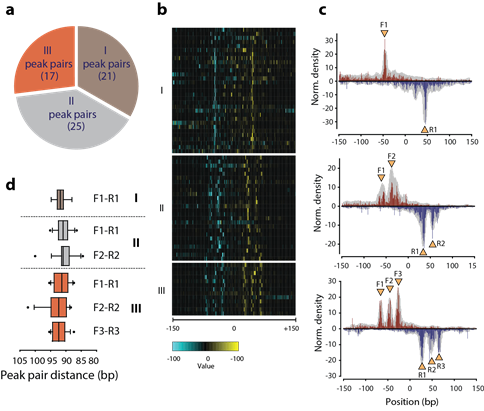
그림 2. 다양한 peak-pair 패턴의 분포도와 특성연구. a) peak pairs 개수에 땨른 결합 유전자의 분류 (I, II, III). b) Heat map 을 사용한 I, II, III peak 위치의 비교. c) I, II, III 각각 peak-pair 의 평준화된 평균값의 분포도. F1-R1, F2-R2, and F3-R3 는 서로 pair를 이룸. d) 이들 peak-pair 거리의 분포도
이와 같이 감지된 peak-pair 사이는 ArgR 이 결합하는 부위를 의미한다. 이들 사이에서의 공통 되는 결합 모티프(motif) 를 찾기 위하여, I 에서는 F1-R1, II 에서는 F2-R2, III에서는 F3-R3 사이의 시퀀스를 추출한 후 MEME 프로그램으로부터 그림 3a 와 같은 결과를 얻었따. 18bp 의 두 개의 역보상성 서열을 가지는 모티프를 찾았고 이들 사이에는 3bp 의 연결하는 서열이 관찰되었다. 그러나 이들의 motif 의 전체 길이가 39 bp 인것을 감안했을 때 본 실험에서 관찰된 거리는 다소 긴 91bp 였다. 이로부터 hexamer 인 ArgR 이 DNA 에 결합하여 루프를 형성할 때 특이적인 결합과 함께 비특이적 결합이 존재함을 알 수 있었다. 얻어진 모든 peak-pair 로부터 각각의 모티프의 위치를 살펴본 결과 91bp 의 위치중에 5’(α), 중간(β), 3‘(γ) 말단에 각각 존재함을 관찰 하였다 (그림 3b). 이 결과로부터 각각 α, β, γ 형태의 결합 형태를 모식도로 나타내었다 (그림 3c). 6개 단백질의 DNA 결합 부위들은 각각의 motif와 결합하게 되고, 나머지 2개는 비특이적으로 결합하게 된다. 앞에서 언급한 다양한 peak-pair 패턴 (I, II, III) 의 경우, 이들은 다양한 형태의 α, β, γ 타입의 구조를 같게 되는데, I 의 경우 α, β, γ 중 하나의 구조를 같게 되며, II의 경우 α 와 β 타입의 결합이 모두 함께 나타나게 되고, III의 경우에는 α, β, γ 형태 모두가 나타나는 경우를 갖게 된다 (그림 3d). 이와 같이 실제 세포내의 유전체에서는 ArgR 은 주위 생리학적 조건이나 혹은 DNA 구조에 따라서, 다양한 형태로 DNA 에 결합하며 RNA polymerase를 조절한다는 것을 알 수 있다.
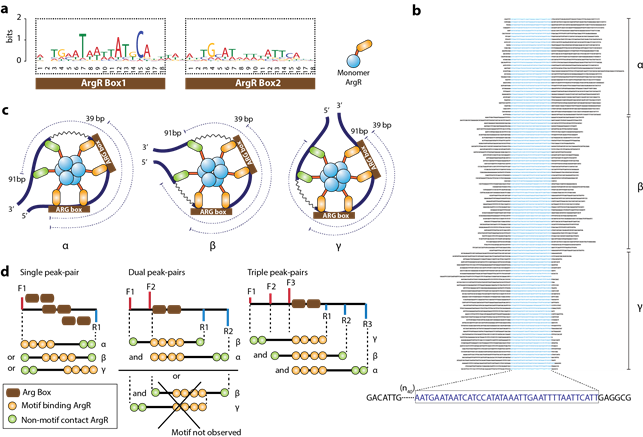
그림 3. ArgR 결합 모티프의 발견 및 다양한 형태의 결합 구조 분석
다음으로 ArgR 의 결합 부위위치가 RNA polymerase 의 프로모터 인지 위치와 어떤 상관관계가 있는지 살펴보았다. 그림 4에서 보이는 것 과 같이 결합하는 위치는 프로모터와 교차하는 경우, upstream, 혹은 downstream 에 위치하는 경우로 다양하였다(그림 4a-c).
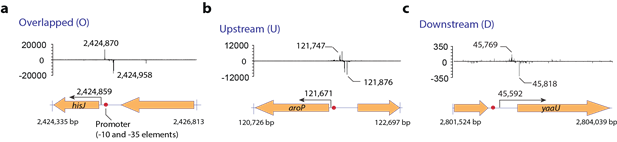
그림 4. ArgR 결합 부위와 프로모터와의 위치적 관계
3. 연구의 성과 및 의의
본 연구에서는 고분해능으로 유전자 전체에 분포하고 있는 전사인자 ArgR 의 결합부위를 규명하였고, 또한 in vivo 에서 실제로 존재하는 이들의 다양한 결합 구조형태를 설명하였다. ArgR 가 모티프를 인지하는 특이적 결합 외에 비특이적 결합을 통해 70˚-90˚ 정도의 DNA bending 에 중요한 역할을 하고 있음을 밝혔다. 프로모터를 중심으로 ArgR 은 upstream, downstream 혹은 프로모터와 같은 위치에 결합함으로써 RNA polymerase 의 활성을 조절하였지만, 이들 위치에 따른 공통적인 조절능력의 차이는 보이지 않았다. 이는 ArgR이 매우 다양한 메커니즘으로 RNA polymerase 와 상호관계를 맺고 있음을 간접적으로 시사해준다.
본 연구와 같은 단염기 수준의 고분해능의 분석 방법을 통해 ArgR 뿐 만 아니라, 수많은 전사인자의 결합위치를 규명할 수 있을 뿐 만 아니라, in vivo에서 다양한 생리학적 조건에 따라 변화하는 이들의 활성 및 구조까지 파악할 수 있는 다양한 가능성을 제시한다. 또한, 합성생물학에 이용되는 다양한 전사인자의 정확한 결합부위를 단시간 내에 규명하여 보다 정밀한 유전자회로를 설계할 수 있다.
참고문헌
1. Rodionov, D.A. (2007) Comparative genomic reconstruction of transcriptional regulatory networks in bacteria. Chem Rev, 107, 3467-3497.
2. van Helden, J., Rios, A.F. and Collado-Vides, J. (2000) Discovering regulatory elements in non-coding sequences by analysis of spaced dyads. Nucleic acids research, 28, 1808-1818.
3. Semsey, S., Virnik, K. and Adhya, S. (2005) A gamut of loops: meandering DNA. Trends in biochemical sciences, 30, 334-341.
4. Cho, B.K., Knight, E.M., Barrett, C.L. and Palsson, B.O. (2008) Genome-wide analysis of Fis binding in Escherichia coli indicates a causative role for A-/AT-tracts. Genome research, 18, 900-910.
5. Lu, C.D., Houghton, J.E. and Abdelal, A.T. (1992) Characterization of the arginine repressor from Salmonella typhimurium and its interactions with the carAB operator. Journal of molecular biology, 225, 11-24.
6. Cho, B.K., Federowicz, S., Park, Y.S., Zengler, K. and Palsson, B.O. (2012) Deciphering the transcriptional regulatory logic of amino acid metabolism. Nat Chem Biol, 8, 65-71.





