전사체 분석과 DNA-free CRISPR 시스템을 이용한 CHO 세포주 부유배양 적응속도 가속화
한국과학기술원 이남일 석박사통합 과정
Namil Lee, JongOh Shin, Jin Hyoung Park, Gyun Min Lee, Suhyung Cho, and Byung-Kwan Cho (2016) Targeted Gene Deletion Using DNA-Free RNA-guided Cas9 Nuclease Accelerates Adaptation of CHO cells to Suspension Culture. ACS Synthetic Biology, DOI: 10.1021/acssynbio.5b00249
1. 연구배경
고령 인구의 증가로 인해 암과 면역 관련 질환이 급속도로 증가되고 있어 항체의약품 시장규모는 지속적으로 확대되고 있다. CHO 세포주는 치료용 재조합 단백질의 생산을 위하여 산업적으로 가장 많이 사용되는 세포주로 현재까지 생산되는 1/3 이상의 치료용 단백질이 CHO 세포주를 통하여 생산되고 있다. 하지만 기존의 CHO 세포주 관련 연구는 배양 조건 최적화에 초점을 맞추어 진행되어 왔을 뿐, 근본적으로 개량된 세포주를 개발하기 위한 연구는 매우 부족한 현실이다.
치료용 단백질 의약품의 대량 생산을 위해서는 부착배양 세포인 CHO 세포주를 무혈청배지에서 부유배양이 가능토록 적응시키는 것이 일반적이다. 한 달이 넘는 기간이 소요되지만 세포가 같은 면적당 많은 수로 자랄 수 있고, 생산된 의약품의 정제가 유리하며, 혈청으로 인해 발생하는 오염문제들로부터 자유롭기 때문에 필수적인 과정이라고 할 수 있다. 하지만 아직까지 적응 기작에 대해 알려진 바가 없어 개량한 세포주에 대해 최적화된 무혈청배지를 개발하는 연구들만 진행이 되어왔다.
최근 DNA sequencing 기술의 비약적인 발전으로 CHO 세포주의 genome sequence, 전사체, 단백체 등의 다양한 omics 데이터가 누적되고 있으며, 방대한 데이터를 바탕으로 유전자와 표현형의 상관관계를 밝히려는 시도가 증가되고 있다. 특히, CHO 세포주의 genome sequence 정보는 유전자 편집 기술로 각광 받고 있는 기술인 Clustered, Regularly Interspaced Short Palindromic Repeats (CRISPR) 시스템을 CHO 세포주에 적용 가능하게 하면서 유전자 엔지니어링을 통한 개량 세포주 개발이 가능해졌다.
CRISPR 시스템은 Cas9 단백질과 sgRNA로 구성되어 있으며 sgRNA가 genome 상의 특이적인 위치로 endonuclease 기능을 가진 Cas9 단백질을 유도하여 Double Strand Break (DSB)을 일으킨다. 잘려진 두 genome은 생명체가 가지고 있는 DNA 복구 기작인 Non Homologous End Joining (NHEJ) 또는 Homologous Recombination (HR)에 의해 복구 되나, NHEJ 같은 경우 정확히 복구하지 못하고 짧은 염기배열을 삽입 또는 결실하며 복구하기 때문에 해당 위치에 존재하는 유전자가 정상적으로 작동할 수 없게 만든다. 이와 같은 특성 때문에 CRISPR 시스템은 유전자를 knockout 시켜 기능을 연구하는데 활발히 사용되고 있다. 일반적으로 CRISPR 시스템을 사용할 때는 Cas9 단백질과 sgRNA를 DNA 형태로 세포 내에 전달한 뒤 발현시키는 방법을 이용한다. 하지만 이와 같은 DNA 전달 방법은 genome상의 무작위적 위치에 DNA 삽입을 유발할 수 있기 때문에 치료용 단백질 생산용 세포주에는 안정성의 측면에서 적합하지 않은 방법이다. 이를 해결할 수 있는 방법으로는 Cas9 단백질은 단백질 형태로, sgRNA는 RNA 형태로 세포에 전달하는 DNA-free CRISPR 시스템이 있다. 단백질과 RNA는 세포내에 전달된 뒤 짧은 시간 내에 분해되기 때문에 genome상의 다른 부분을 바꾸지 않고 원하는 위치만을 엔지니어링 하는 확률이 높다. 또한 외래 단백질인 Cas9을 발현시키기 위해 사용하는 프로모터와 코돈의 최적화 과정이 필요치 않아 손쉽고 경제적인 방법이다.
본 연구에서는 전사체 분석기술과 DNA-free CRISPR 시스템을 이용하여 빠르고 합리적인 CHO 세포주 엔지니어링 기술을 제안하였다. CHO 세포주 부유배양 적응 과정 중의 전사체 변화를 추적하여, 발현양이 변화하는 유전자들을 스크리닝 해내었고 DNA-free CRISPR 시스템을 CHO 세포주에 최초로 도입하여, 밝혀낸 유전자와 표현형의 상관관계를 증명하였다.
그림 1. 본 연구에서 제안한 전사체 분석기술과 DNA-free CRISPR 시스템을 이용한
CHO 세포주의 합리적인 엔지니어링 과정.
2. 연구결과
부유배양 적응 과정에서의 전사체 변화양상을 살펴보기 위하여 적응 과정 중 총 5개의 시점에서 세포를 샘플링하고 Strand-specific RNA sequencing을 진행하였다 (그림2A). 얻어진 1억 8천만개의 sequencing read들을 CHO 세포주의 genome sequence에 mapping시킨 결과 93% 이상의 read들이 성공적으로 mapping되는 것을 확인하였으며 biological duplicate끼리의 재현성도 매우 높음을 확인하였다 (그림2B). 각 유전자의 발현양을 수치화 한 뒤에 5개의 시점에 대하여 발현양이 증가 또는 감소하는 유전자들을 K-mean clustering 기법으로 그룹화 하였다 (그림2C). 각 그룹에 대하여 KEGG 대사경로 분석을 진행한 결과 세포 생장, 복제, 핵산 대사와 관련된 그룹들은 유전자 발현양이 증가하는 것을 확인하였으며, 세포 부착과 직접적으로 관련된 유전자들은 발현양이 감소하는 것을 확인할 수 있었다 (그림2D). 결과적으로 전사체 분석을 통하여 기작이 밝혀지지 않은 부유배양 적응 과정에서 어떠한 기능을 가지는 유전자들의 발현양이 변화하였는지를 분석해낼 수 있었다.
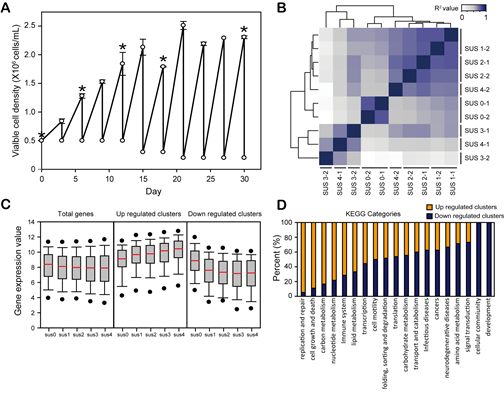
그림 2. CHO 세포주의 부유 배양 적응과정 중의 전사체 변화 분석. (A) CHO 세포주의 부유 배양 적응 과정의 생존 곡선 (B) RNA sequencing 결과의 재현성 분석. (C) K-mean clustering으로 그룹화한 발현양이 증가 또는 감소하는 유전자들의 발현양 분포도. (D) 발현양이 증가 또는 감소하는 유전자들이 속해 있는 KEGG 대사경로.
부유배양 적응을 가속화 시킬 수 있는 유전자 후보를 선별하기 위하여 부유배양 적응이 진행되는 동안 발현양이 가장 큰 폭으로 감소한 유전자 5개를 선별하였다. Igfbp4, Sulf2, Fos, Aqp1, Nr4a1이 타겟으로 선정 되었으며 이 유전자들은 암의 발생과 관련이 있는 유전자들이다. CHO 세포주의 부유배양 적응과정의 표현형 변화가 암의 전이과정과 매우 유사하기 때문에 이 유전자들이 핵심적인 역할을 할 것이라 예상할 수 있었다. 이 유전자들의 역할을 증명하기 위하여 DNA-free CRISPR 시스템을 CHO 세포주에 적용하여 각 유전자 knockout을 진행하였다. Cas9 단백질을 E.coli에서 발현시킨 뒤 Ni-NTA bead를 이용하여 정제하였고, 타겟으로 선정한 다섯 개의 유전자에 대하여 각자 sgRNA를 디자인하고 In vitro transcription 시스템을 이용하여 RNA 형태로 정제하였다. 정제된 Cas9 단백질과 sgRNA를 세포에 처리해준 결과, 39~54%의 효율로 타겟하는 유전자를 knockout하는데 성공하였다.
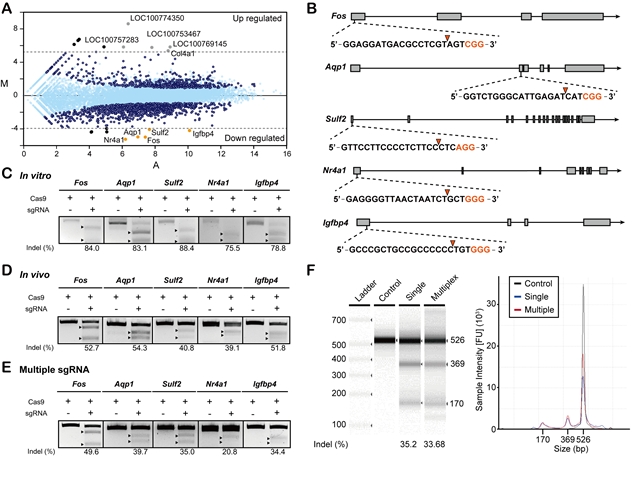
그림 3. DNA-free CRISPR 시스템을 이용한 CHO 세포주 부유배양 적응 관련 유전자 엔지니어링. (A) MA-plot 분석기술을 이용한 부유배양 적응 관련 엔지니어링 타겟 유전자 선정. (B) 5개의 선정된 타겟 유전자에 대한 sgRNA 디자인 (C) In vitro에서 DNA-free CRISPR 시스템 작동 여부 확인. (D) in vivo에서 DNA-free CRISPR 시스템을 이용한 5개의 유전자에 대한 genome 엔지니어링 결과 (E) 5개의 유전자에 대한 동시다발적 genome 엔지니어링 결과 (F) Bio analyzer를 이용한 genome 엔지니어링 결과 추가 확인.
타겟 유전자가 knockout된 CHO 세포주 라이브러리를 무혈청배지에서 부유배양 적응시키며 각 knockout이 부유배양 적응 속도에 어떤 영향을 끼쳤는지 확인하였다. 그 결과 다섯 개의 유전자 중 Igfbp4, Aqp1을 knockout시킨 CHO 세포주 라이브러리가 ~54%의 감소된 부유배양 적응 시간을 보이는 것을 확인하였다.
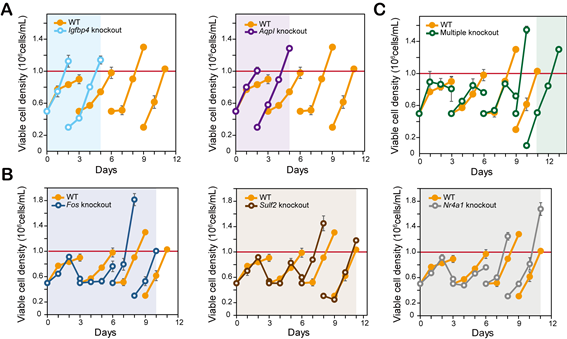
그림 4. 유전자 엔지니어링을 완료한 CHO 세포주 라이브러리를 무혈청배지에서 부유배양 적응 시킨 결과. (A) 부유배양 적응 시간이 감소된 Igfbp4와 AqpI knockout CHO 세포주 라이브러리의 부유배양 적응 생존 곡선. (B) 부유배양 적응 시간에 영향을 끼치지 못한 Fos, Sulf2, Nr4a1 knockout CHO 세포주 라이브러리의 부유배양 적응 생존 곡선. (C) 다섯 개의 타겟 유전자를 동시다발적으로 엔지니어링한 CHO 세포주 라이브러리의 부유배양 적응 생존 곡선.
추가 확인을 위하여 다섯 개의 유전자에 대한 sgRNA를 동시에 처리하여 다양한 knockout 조합을 포함하고 있는 CHO 세포주 라이브러리를 제작하였다. 이 CHO 세포주 라이브러리의 부유배양 적응을 시도한 뒤에 부유배양 적응 시작 시점과 완료 시점의 세포주 구성을 차세대 시퀀싱 기반의 Targeted deep sequencing 기술로 분석하였다. 그 결과 다른 세 개의 유전자 knockout을 포함하고 있는 세포주는 부유배양 적응이 완료됨에 따라 그 비율이 감소하거나 유지되었지만 Igfbp4와 Aqp1이 kncokout된 CHO 세포주의 비율은 증가한 것을 확인할 수 있었다. 이 결과는 Igfbp4와 Aqp1의 knockout이 실제로 CHO 세포주의 부유배양 적응에 효과를 가진다는 것을 다시 한번 보여주었다.
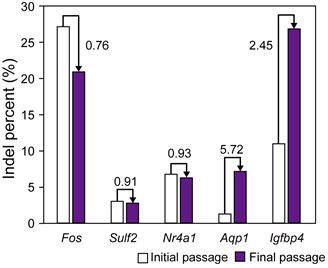
그림 5. 5개의 유전자를 동시다발적으로 엔지니어링한
CHO 세포주 라이브러리의 부유배양 적응 과정에서의 세포주 구성 변화.
3. 연구의 성과 및 의의
부유배양 적응은 치료용 단백질 의약품의 대량 생산에 있어서 필수적인 단계이다. 본 논문에서는 차세대 시퀀싱 기반의 RNA-seq 기술을 이용하여 부유배양 적응 동안의 전사체 변화를 밝혀내었다. 그 결과, 부유배양 적응과정에서 생장과 관련된 유전자들의 발현양은 증가하는 것으로 확인되었으며, 세포부착에 관련된 유전자들은 발현양이 감소하는 것을 시스템적인 레벨에서 확인할 수 있었다. 발현양이 변화하는 유전자들 중 발현양이 가장 많이 감소하는 5개의 유전자를 엔지니어링 타겟으로 선정하였으며 DNA-free CRISPR 시스템을 적용시켜 각 유전자에 대한 knockout을 진행하였다. 39%-54%의 높은 효율로 엔지니어링이 됨을 확인하였으며, 엔지니어링된 CHO 세포주 라이브러리를 부유배양 적응시켜 본 결과, Igfbp4와 Aqp1유전자가 knockout 된 경우 부유배양 적응이 가속화 된다는 것을 최종 확인할 수 있었다. 이 연구를 통하여 원하는 표현형을 가지는 CHO 세포주를 제작하는 합리적인 엔지니어링 플랫폼 기술을 구축할 수 있었다. 알려지지 않은 기작으로부터 엔지니어링 후보 유전자들을 발굴 해내고 실제 엔지니어링 효과까지 검증한 이 플랫폼 기술은 CHO 세포주 뿐만 아니라 다양한 세포주에도 쉽게 적용 가능하며, 향후 고품질의 단백질 의약품을 고생산하는 CHO 세포주를 개발하는데 크게 기여할 것이라고 생각한다.
참고문헌
1. Xu, X., Nagarajan, H., Lewis, N. E., Pan, S., Cai, Z., Liu, X., Chen,W., Xie, M., Wang, W., Hammond, S., Andersen, M. R., Neff, N.,Passarelli, B., Koh, W., Fan, H. C., Wang, J., Gui, Y., Lee, K. H.,Betenbaugh, M. J., Quake, S. R., Famili, I., Palsson, B. O., and Wang, J.(2011) The genomic sequence of the Chinese hamster ovary (CHO)-K1 cell line. Nat. Biotechnol. 29, 735?741.
2. Lewis, N. E., Liu, X., Li, Y., Nagarajan, H., Yerganian, G., O’Brien,E., Bordbar, A., Roth, A. M., Rosenbloom, J., Bian, C., Xie, M., Chen,W., Li, N., Baycin-Hizal, D., Latif, H., Forster, J., Betenbaugh, M. J.,Famili, I., Xu, X., Wang, J., and Palsson, B. O. (2013) Genomiclandscapes of Chinese hamster ovary cell lines as revealed by theCricetulus griseus draft genome. Nat. Biotechnol. 31, 759?765.
3. Baik, J., Ha, T., Kim, Y., and Lee, G. (2011) Proteomicunderstanding of intracellular responses of recombinant chinesehamster ovary cells adapted to grow in serum-free suspension culture.Biotechnol. Prog. 27, 1680?1688.
4. Ronda, C., Pedersen, L. E., Hansen, H. G., Kallehauge, T. B.,Betenbaugh, M. J., Nielsen, A. T., and Kildegaard, H. F. (2014)Accelerating genome editing in CHO cells using CRISPR Cas9 andCRISPy, a web-based target finding tool. Biotechnol. Bioeng. 111,1604?1616.
5. Sander, J. D., and Joung, J. K. (2014) CRISPR-Cas systems forediting, regulating and targeting genomes. Nat. Biotechnol. 32, 347?355.





