포유세포에서 CRISPR 전사 억제 장치와 유전자 회로 개발

KAIST 바이오융합연구소 허진행 연구원
Kiani, S., Beal, J., Ebrahimkhani, M. R., Huh, J., Hall, R. N., Xie, Z., Li, Y., Weiss, R. CRISPR transcriptional repression devices and layered circuits in mammalian cells. Nature Methods, 11, 723-726 (2014).
1. 연구배경
유전자 회로 개발은 살아있는 세포의 근본적인 생물학에 대한 통찰력과 의료에서부터 산업까지 이르는 많은 문제에 대한 잠재적인 해결 방안을 제공한다1,2. 이러한 정교한 유전자 회로를 효율적으로 개발하기 위해서는 새롭고 예측가능한 행동을 구현할 수 있고 여러 조건에서 연결 할 수 있는 제어장치 라이브러리가 필요로 하다. 합성 생물학에서 제어 장치란input의 정보를 프로세스하여 output과 연결 시키는 생화학적인 상호작용으로 크게 정의 내린다1,3. 우리는 input을 어느 인자의 발현으로 이용하고, 그 인자가 output의 전사를 조절하는 전사 장치를 연구하였다. 특히 전사 활성인자들과는 달리 전사 억제 단백질들은 이론상 어느 연산 회로나 만들 수 있기 때문에 전사 억제 장치에 초점을 맞추어 왔다4,5. 현재까지 미생물에서든 동물세포에서든 크고 복잡한 회로를 만들 수 없었던데에는 끼고 맞추어 작동하는 유전자 회로를 만들 만큼의 충분한 숫자의 orthogonal한 전사인자와 그의 프로모터 짝이 없다는 것이었다3.
이 문제를 해결하기 위해 zinc finger 단백질이나 transcription activator-like effector (TALE) 단백질을 이용한 노력이 있지만, 복잡한 DNA 조립 방법과6,7,8 이들 단백질에 의해 타겟 프로모터의 epigenetic 변형에 의해 응답 속도가 늦어지는 문제를 가지고 있다9. 이 밖에 최근 Streptococcus pyogenes CRISPR-Cas immune systme의 Cas9 단백질을 RNA-guided 게놈 편집과 유전자 조절에 적용시킨 연구들이 발표되었는데8,10,11,12,13, 이 메커니즘이 유전자 조절 장치의 라이브러리를 만들 때 유용한 이유는 어느 DNA 서열이든지 그에 맞게 small guide RNA (gRNA)의 유전자 서열을 바꾸어주면 쉽게 Cas9으로 그 DNA 서열을 잘라 낼 수 있기 때문이다6. 또한 catalytically inactive Cas9 (Cas9m)을 이용해 전사를 억제할 수 있다8,9,10,11. 그러므로 우리는 이 메케니즘을 이용해 전사 억제 장치와 유전자 회로를 만들어 보았다. 구체적으로RNA polymerase type II (RNA Pol II)와 RNA Pol III를 사용하여 인간 세포에서 gRNAs의 통제된 전사 방법을 고안하였고, CRISPR 전사 억제 장치는 높은 on/off 비율을 가진 유전자 회로를 조립할 수 있다는 것을 보여 주었다.
2. 연구결과
전사 억제 장치는 Cas9m과 gRNA로 구성되어 있는데, gRNA가 특정한 프로모터를 타겟팅하게 함으로써 Cas9m이 그 프로모터의 전사 기능을 억제 시키는 메케니즘이다. gRNA를 전사하기 위해 일반적으로 RNA Pol III를 사용하는데 U6 프로모터가 자주 쓰인다. 이 부품들을 이용해 layered된 유전자 회로를 만들고 cascade를 실험하였다 (그림 1). 그런 다음, intron을 지닌 단백질이 발현될 때 intron이 splice되는 것을 이용하여 intron자리에 gRNA를 넣음으로써 유전자를 전사할때 쓰이는 Pol II를 이용하여서도 gRNA를 전사할 수 있게 하였다. 이 방법의 장점은 기존의 유전자 회로에 많이 사용되는 GAL4VP16이나 TRE 시스템과 같은 유전자 조절 부품들을 이용하여 gRNA의 전사를 조절할 수 있기 때문이다. Doxycycline으로 전사되는 정도를 조절할 수 있는 TRE 프로모터를 더하여 CRISPR 전사 억제 장치와 유전자 회로를 만들고 cascade를 실험하였다 (그림 2).
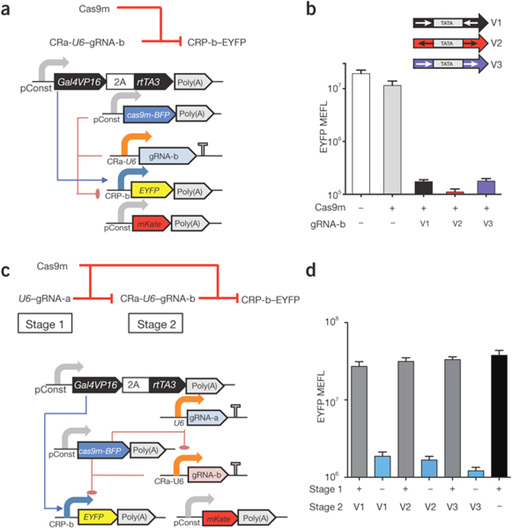
그림 1: RNA Pol III U6 프로모터를 이용한 CRISPR 전사 억제 장치와 유전자 회로의 설계 및 실험 분석. (a) CRP 억제 장치의 회로도. CRa-U6는 gRNA-b의 전사를 조절하고, gRNA-b는 EYFP output의 전사를 조절한다. (b) HEK293세포에서 세가지 버전의CRa-U6 프로모터로 조절되는 gRNA-b. (C) CRISPR 전사 억제 장치 cascade의 회로도. U6에 의해 조절되는 gRNA-a는 CRa-U6에 의해 조절되는 gRNA-b를 조절하고, 이 gRNA-b는 EYFP ouput의 전사를 조절한다. (D) CRISPR 전사 억제 장치 cascade의 시험.
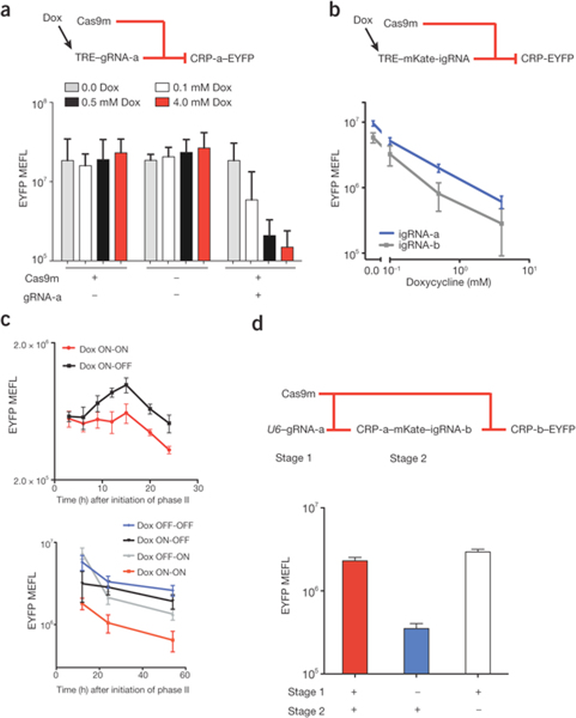
그림 2: RNA Pol II를 이용한 CRISPR 전사 억제 장치와 유전자 회로의 설계 및 실험 분석. (a) TRE 프로모터와 doxycycline (Dox)에 의해 작동되는 gRNA-a 전사 억제 장치. (b) TRE 프로모터에 의해 조절되는 mKate과 igRNA를 발현하는 igRNA 전사 억제 장치. (C) Dox를 이용한 reversible한 전사 억제 실험 분석. (D) igRNA를 이용한 cascades 실험.
3. 연구의 성과 및 의의
Zinc finger 단백질이나 TALE 단백질을 이용한 전사 조절 장치에 비해 CRISPR를 이용한 전사 조절 장치는 유전자 회로에 layer를 하나 하나 더할 때 마다 유전자 회로 자체에 더해지는 총 DNA크기가 작기 때문에 크고 복잡한 유전자 회로를 만들때 유용하다. 이는 같은 topology를 가진 유전자 회로를 훨씬 작은 크기의 DNA에 인코딩 할 수 있으며 매우 복잡한 회로를 size limitation이 있는 운반체에 담을수 있음을 의미한다. 또한 CRISPR 전사 억제 장치는 RNA interference에 의한 유전자 조절 메케니즘과 잘 통합 될것으로 예상되어 세포의 상태나 프로파일을 구별할 수 있는 유전자 회로를 만들 때 중요하게 쓰일것이다. 또한 orthogonal Cas9 단백질들을 이용하면14 한 층 더 복잡한 유전자 회로를 만들 수 있을것이다. 마지막으로, gRNA와 그에 맞는 프로모터를빠르고 쉽게 추가로 만들 수 있기 때문에 빠른 시간안에 큰 제어장치 라이브러리를 만들고 실험 및 최적화가 가능하다. 따라서 우리가 개발한 전사 억제 장치는 이러한 장점들을 바탕으로 인간 세포에서 많은 어플리케이션에 적용될 수 있을것이다.
참고문헌
1. E. Andrianantoandro, S. Basu, D. K. Karig & Weiss, R. Mol. Systems Biol. 2, 0028 (2006).
2. Ruder, W.C., Lu, T. & Collins, J.J. Science 333, 1248–1252(2011).
3. Slusarczyk, A.L., Lin, A. & Weiss, R. Nat. Rev. Genet. 13,406–420 (2012).
4. Bird, J. Engineering Mathematics 532 (Elsevier Science,2007).
5. Peirce, C.S. Collected Papers of Charles Sanders Peirce vol.4, 12–20 (Harvard University Press, 1933).
6. Farzadfard, F., Perli, S.D. & Lu, T.K. ACS Synth. Biol. 2,604–613 (2013).
7. Khalil, A.S. et al. Cell 150, 647–658 (2012).
8. Garg, A. et al. Nucleic Acids Res. 40, 7584–7595 (2012).
9. Kramer, B.P., Fischer, C. & Fussenegger, M. Biotechnol. Bioeng. 87, 478–484 (2004).
10. Stanton, B.C. et al. Nat. Chem. Biol. 10, 99–105 (2014).
11. Fu, Y. et al. Nat. Biotechnol. 31, 822–826 (2013).
12. Mali, P. et al. Science 339, 823–826 (2013).
13. Qi, L.S. et al. Cell 152, 1173–1183 (2013).
14. Esvelt, K.M. et al. Nat. Methods 10, 1116–1121 (2013).





