합성유전자의 발현을 위한 이종숙주의 genome engineering
이강록, 노정혜
서울대학교 생명과학부
1. 개요
합성생물학은 대부분의 경우 새로운 물질을 합성하거나 특정 물질을 분해하도록 설계된 생명체를 만들어내는 것을 목적으로 한다. 이를 위해서는 기존에 존재하지 않던 새로운 유전 형질을 생명체 내로 도입하는 것이 필요하다. 이를 위해 플라스미드에 원하는 유전자들을 도입해서 발현시키는 방법이 있지만, 기존에 존재하는 유전자들의 산물과의 상호작용으로 인해서 독성을 나타내는 등의 부작용이 발생할 수 있다. 이러한 문제 해결하기 위해 대장균에서 유전체 내에 존재하는 유전자에 돌연변이를 일으키고, 이를 대신할 수 있는 다른 유전자를 도입하는 방법들이 개발되고 있다.
유전체로의 유전자 도입 방법은 대장균뿐만 아니라, High GC 그람 양성인 Streptomyces에서도 적극적으로 활용되고 있다. Streptomyces는 미생물로부터 만들어지는 항생제와 유용한 이차대사산물 중 약 절반을 만들어 내고 있다. 그러나 실제 발견되는 Streptomyce 중 많은 경우 배양하기 부적합해서 아직 다양한 종의 Streptomyces가 연구되지 못하고 있다. 게다가 항생제 내성균의 증가로 인해 이러한 배양하기 힘든 균주들이 가진 많은 항생이나 항균에 관여하는 물질의 중요성이 커지고 있다. 이러한 유전체들을 도입해서 발현시킬 균주로 비슷한 유전정보를 가진 이종숙주 Streptomyces들이 이용되고 있다. 또한 Bacteria의 효과적인 genome editing을 위해 RNA를 활용한 editing system도 점차 많이 활용되고 있다.
2. 대장균의 recombineering
특정 위치에서 상이한 유전자를 발현하는 돌연변이를 만들기 위해서, phage-based homologous recombination을 이용하는 것을 Recombineering이라고 한다. 대표적으로 대장균에서 linear DNA를 이용한 간편한 돌연변이 제조를 위해서 λ phage DNA를 이용한 site-directed recombination 방법이 있다. Red system이라고 하는 5’→3’ exonuclease, recombinase와 RecBCD의 inhibitor이 활성화된 균주에, homologous region과 maker 유전자를 가진 linear DNA를 transformation하는 방법을 통해서 돌연변이를 제조하게 된다. 새로운 형질 도입 과정에서 marker들은 모두 제거되는데, marker의 종류에 따라 다양한 제거 방법이 존재한다. Flp-FRT, Cre-lox system 혹은 I-SceI는 marker의 종류와 상관없이 site-specific recombination을 이용해서 원하지 않는 DNA를 잘라내는 방법으로 유전체에 scar라고 하는 DNA sequence를 남기게 된다. scar를 만들지 않기 위해서 사용되는 counter-selection은 homologous recombination을 이용해서, marker로 사용된 sacB, tetAR, rpsL, 혹은 galK가 도입하고자 하는 유전자로 대체된 것을 골라내는 방법이다.
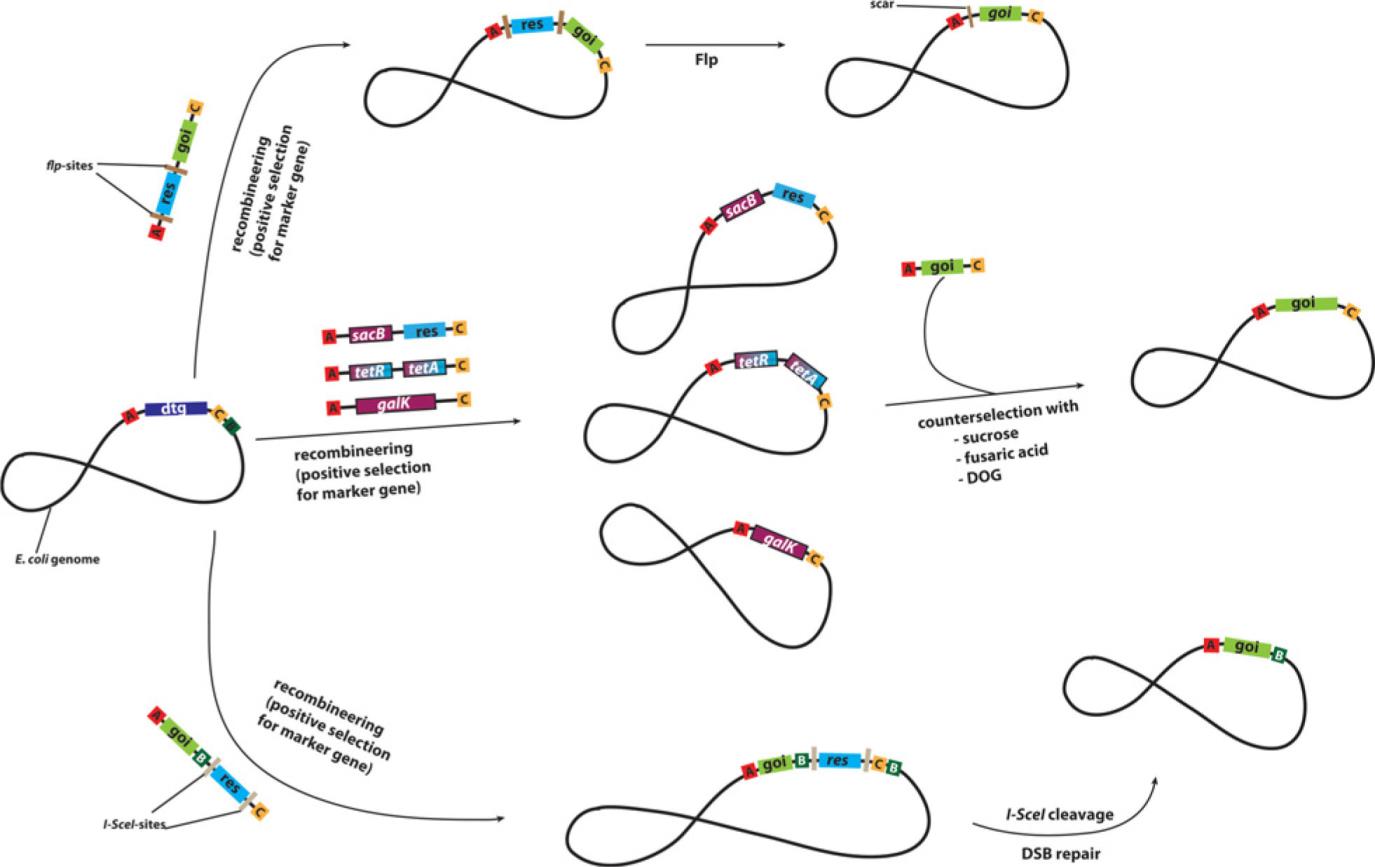
그림 1. 대장균 유전체 상에 유전자들을 directed integration하는 방법들. 상동 지역은 적색 (A), 녹색 (B), 노란색 (C) 상자로
표시된다. DTG, 삭제 대상 유전자, res, 항생제 저항성 유전자, GOI, 그 유전자 FLP와 SceI 인식 사이트들도 표시된다.
(Richter and Gescher, 2012)
Recombineering이 원하는 위치에 유전자를 도입할 수 있어서 대부분 사용하는 방법임에도 불구하고, Phage- and transposon-based site-specific integration systems는 많은 장점으로 인해서 여전히 유용하게 이용되고 있다. 그 장점들로는 유전체에 integration 효율이 매우 높고, 특정 위치에만 일어나기에 예측이 가능하며, integration이 일어나더라도 가역적이라는 것 등이 있다. CRIM (conditional-replication, integration and modular) plasmids는 다섯가지의 다른 attP site와 서로 다른 항생제 저항성을 이용해서 모두를 사용할 수 있다. 또다른 방법으로 Tn7이 매우 높은 효율로 glmS gene의 3’ end에 끼어 들어가는 원리를 이용해서 marker를 이용하지 않고 형질을 도입하기도 한다.
도입된 유전자들의 발현을 위해서는, 발현을 통해 나타날 수 있는 잠재적 독성을 방지하거나 합성과정의 다양한 단백질 활성을 조절하는 것이 매우 중요하다. 발현량의 조절에서 promoter의 선택은 통제가능한 전사 조절을 위해서 필수적이다. 이를 위해서 lac, ara and tet promoters와 변형체들 외에도 propionate promoter, alkS와 alkB promoter, ugp promoter, tac promoter, rhaPBAD promoter 등이 사용되고 있다.
3. Streptomyces를 이용한 이차대사산물 합성 유전자의 이종숙주 발현
Streptomyces는 1) 이차 대사산물 생합성 유전자의 clustering 확인, 2) 천연물의 분석, 3) 유전자 engineering을 통한 천연물의 변이체 생산, 4) 유전자 library screening을 통한 새로운 화합물의 발견에 이용되어 왔다. 구체적인 예로, 일본 Kitasato 대학의 Ikeda 교수 연구진은 avermectin (Polyketide 계열의 항 기생충제)의 생산 균주인 Streptomyces avermitilis의 genome minimize를 통해 다양한 이차대사산물의 생산이 가능한 방선균 이종숙주를 개발하였다. 해당 연구진은 Spectinomycin, chephamycin, pladienolide, sesquiterpene 등 다양한 이차대사산물의 이종숙주 생산에 성공하여 방선균 세포공장의 개발 가능성을 보여주었다. John Innes Centre의 Mervyn J. Bibb 교수 연구진은 이차대사산물의 이종숙주발현을 최대화 하고, 최종산물의 순수분리과정을 단순화기 위하여, 기존의 2차대사 gene cluster들을 차례로 deletion시킨 S. coelicolor균주를 개발하였다. Gene cluster의 deletion을 위해서는 cluster의 5’ side와 3’ side에서 각각 2 kb정도의 유전자조각을 합성하고 이것을 ligation한 recombinant DNA를 suicide vector를 이용하여 숙주에 도입한후 double cross-over가 일어난 균주를 찾는 방식으로 진행한다. 여기에 전사와 번역의 특이성을 변화시킨 RNA 중합효소 rpoB와 ribosome단백질 rpsL의 point mutation을 첨가하여 heterologous gene cluster로부터 이차대사산물의 생산성을 크게 증가시킬 수 있는 방식이 성공할 수 있음을 보여주었다. 긴길이의 생합성 유전자 cluster를 이종숙주에 안정적으로 도입하는 artificial chromosome vector들은 대장균의 F factor나 P1 phage에 기반한 BAC과 PAC으로부터 개발되어 100 kb이상의 DNA를 도입하는 시스템들이 쓰이고 있다 (Alduina and Gallo, 2012).
4. CRISPR/Cas system의 활용
최근에는 CRISPR(clustered, regularly interspersed, short palindromic repeats)과 연계된 Cas9 endonuclease를 활용한 genome editing방법이 많이 사용되고 있다. 대장균과 Streptococcus를 대상으로 한 실험에서, 두개의 short CRISPR RNA (crRNA)를 사용한 multiplex mutagenesis가 가능하고, mutation 되지 않은 genome은 dual-RNA-Cas complex를 이용하여 잘라냄으로써 selectable marker나 counter-selection을 우회해가는 방법이 개발되었다. Rockefeller 대학 Marraffini group의 보고에 따르면, S. pnemoniae는 골라낸 균주의 100%가 모두 mutant였고, E. coli는 65%에 해당한다고 한다. 이 방법의 적용이 다른 균주들의 genome editing에도 유용하게 사용될 것으로 전망된다. Genome editing 뿐아니라 engineered CRISPR/Cas system을 이용하여 유전자의 발현을 억제하거나 활성화시키는 전략도 사용될 수 있다.
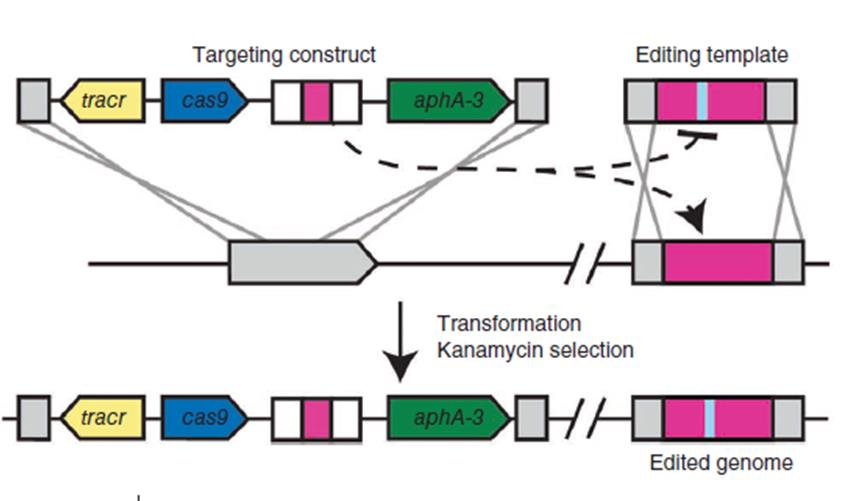
그림 2. CRIPR-Cas 시스템을 이용한 genome editing의 개념. Chromosome상의 target 유전자에 대한 Dual-RNA:Cas9의
nuclease활성을 이용. 원하는 형태로 editing이 된 template로 치환된 target은 잘리지 않고, 치환되지 않은 원래
유전자는 잘림으로써, edited genome을 얻을 확률을 극대화 할 수 있음. (Jiang et al., 2013)
5. 참고문헌
- Richter, K. and Gescher, J., (2012) The molecular toolbox for chromosomal heterologous multiprotein expression in Escherichia coli. Biochem. Soc. Trans. 40:1222-1226.
-Komatsu M, Uchiyama T, Omura S, Cane DE, Ikeda H. (2010) Genome-minimized Streptomyces host for the heterologous expression of secondary metabolism. Proc Natl Acad Sci U S A. 2010 Feb 9;107(6):2646-51.
-Gomez-Escribano, J.P., Bibb, M.J. (2011) Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microb Biotechnol. 4(2):207-15
-Gomez-Escribano, J.P., Bibb, M.J. (2012) Streptomyces coelicolor as an expression host for heterologous gene clusters. Methods Enzymol. 517:279-300
-Alduina, R. and Gallo, G. (2012) Artificial chromosomes to explore and to exploit biosynthetic capabilities of actinomycetes. J Biomed Biotechnol. 2012:462049
-Cong, L., Ran, F.A., Cox, D., Lin, S., Barretto, R., Habib, N., Hsu, P.D., Wu, X., Jiang, W., Marraffini, L.A., Zhang, F. (2013) Multiplex Genome Engineering Using CRISPR/Cas Systems. Science 339:819
-Bikard, D., Jiang, W., Samai, P., Hochschild, A., Zhang, F., Marraffini, L.A. (2013) Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system. Nucleic Acids Res. 41(15):7429-7437
-Jiang, W., Bikard, D., Cox, D., Zhang, F., Marraffini, L.A. (2013) RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat Biotechnol. 31(3):233-9.





