합성생물학 부품 표준화 및 설계 정형화에 관한 연구 동향
이승구/김하성 (한국생명공학연구원)
1. 개요
합성생물학의 목표 중 하나는 예측 가능한 새로운 기능을 수행하는 생물학적 시스템을 구현하는 것으로 다른 공학 분야의 설계에서와 같이 시스템을 이루는 기본 구성 요소들을 조합하고 연동하여 구현될 수 있다. 합성생물학에서는 이러한 시스템의 기본 구성 요소들을 부품이라고 정의하고 있는데 이들은 짧은 DNA 서열로서 프로모터, 리보좀 결합 사이트, ORF, 종결자 등을 포함한다. 이러한 부품들은 토글 스위치나 신호 발생기와 같은 간단한 기능을 하는 모듈로서 조립될 수 있으며 모듈들의 조합으로 보다 큰 규모의 시스템으로 확장될 수 있다.
그러나 생물 시스템의 복잡성과 부품간의 간섭현상 등으로 시스템이 원하는 기능을 수행하면서 정확한 조절과 예측을 기대하기는 어려운 현실이다. 이러한 특성을 갖추기 위해서는 해당 시스템을 구성하는 부품의 특성을 정확히 파악하고 주어진 조건의 입력과 출력이 정량적으로 표준화된 부품을 사용하는 것이 필수적이라고 할 수 있다. 특히 합성생물학은 생물, 화학, 물리 등 기초과학 학문뿐만 아니라 전자나 컴퓨터 등 공학 관련 학문들의 연계 연구가 필수적인 영역으로 사용되는 부품의 표준화가 더욱 절실히 필요한 상황이다. 본 보고서에서는 이러한 합성생물학에서의 부품 및 표준화에 관한 연구동향을 살펴보고 이들을 이용하여 조절과 예측이 가능한 생물 시스템 설계를 위한 정형화된 설계의 발전 방안에 대한 고찰해본다.
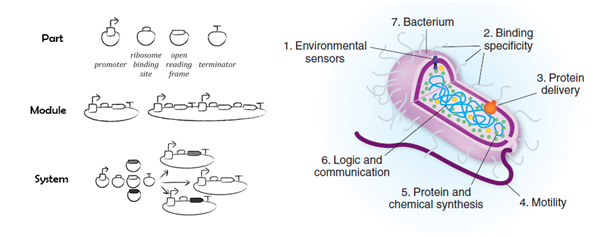
그림 1. (좌) 부품 (Part), 모듈(Module), 시스템(System) 개념도[4].
(우) 합성 박테리아 시스템에서 기능별 모듈 구분도 [3].
2. 부품 표준화 연구
전통적인 유전공학 연구는 하나 또는 두 개의 유전자를 조절하면서 복잡한 전체 시스템의 표현형을 분석하는 반면 합성생물학에서는 시스템에서 기능을 하는 각 구성 요소들의 상호작용을 고려하여 시스템의 기능을 발현을 유도하도록 각 요소들을 조합하는 상향식 연구라고 할 수 있다. 이러한 합성생물학은 초기 간단한 스위치 제작으로부터 시작하여 [1] 계단식 회로, 시간 지연 회로, 오실레이터 등 여러 가지 모듈 연구가 활발히 진행되었다 [2]. 결국 이러한 간단한 모듈 연구의 발달은 궁극적으로 보다 복잡하고 정교한 생물 시스템의 구축을 목표로 하는 연구 흐름을 형성하게 되었는데 자연스럽게 부품의 표준화와 [3] 시스템 설계의 효율성에 대한 연구에 대한 필요성이 주목 받게 되었다 [2].
유럽지역의 대표적인 부품 표준화 및 직교화 연구로 EU Seventh Framework Programme 에서 지원하는 “Standarization and orthogonalization of the gene expression flow for robust engineering of NTN (new-to-nature) biological properties” 연구를 들 수 있다 [5]. 이 프로젝트는 유럽에 위치한 14개의 주요 합성생물학 그룹들이 모여서 진핵 생물 시스템의 설계를 위한 재료와 컴퓨팅의 기준을 확립하기 위해서 조직되었다. 4년간 약 90억 원의 지원을 받는 이 프로젝트는 구성물의 물리적 원리를 포함한 완전히 예측 가능한 기능을 가진 생물학적 장치나 모듈 더 나아가 전체 시스템의 기본적인 원리들을 발견하고 구현 가능하도록 하는 것이다. 원하는 시그널을 생산해내는 DNA시퀀스를 합성하는 것에서부터 시작하여 세포가 최종 생산물을 만들어내는 경로 전체에 대한 모든 과정에 대한 연구가 진행되는데 각각의 유전자 발현 정보를 탐색하여 구성물질의 물리적 성질과 생물학적 기능의 연관관계를 연구하는 상향식 방법과 최종 결과물의 예측값과 결과값의 차이를 보정하는 하향식 방법의 두 가지 방법을 상호보완적으로 사용하고 있으며 윤리 관련 이슈도 다루고 있는 프로젝트이다. 그러나 합성생물학에서 가장 눈에 띄는 표준화 노력은 Biobricks Foundation의 iGEM 경연대회와 Registry of Standard Biological Parts라고 할 수 있다 [6]. 그들은 BioBricks라는 표준화된 유전적 부품을 개발하여 참가자들에게 제공하고 이들을 조합하여 좀 더 복잡한 시스템을 만드는 작업을 한다. 이러한 노력에는 여러 재미있는 특징들이 있지만 그 중 가장 중요한 부분은 참가자들의 노력으로 표준화할 수 있는 데이터를 쌓는 것이다. 몇 년간의 데이터를 축적한 후 BioBricks는 IETF RFC 포맷을 따르는 RFC를 발간했다. 이러한 표준화 문서는 초기단계 이지만 머지않아 중요한 표준화 문서로 자리매김 할 것이 분명해 보인다.
전통적인 과학에 있어서 특히 생물학 분야의 표준화는 다음과 같은 다섯 가지 사항이 필수적이라고 할 수 있다. 1) 샘플들간 결과가 비슷해야 한다 2) 실험실간 결과가 비슷해야 한다 3) 여러 실험실에서 실험이 재현 가능해야 한다 4) 연구자들이 같은 명명법을 써야 한다 5) 실험 데이터는 전산처리 가능해야 한다. 이러한 요구사항은 합성생물학의 부품 표준화 연구에 있어서도 동일하게 적용 될 수 있는데 이를 위해서는 DNA 부품의 특성 파악이 선행되어야 한다. DNA 부품들 중에서 가장 중요한 것 중 하나가 조절 프로모터인데 이 부품의 DNA 개량에 의한 다양한 특성 연구들이 진행되어 왔다. 그 중 대표적으로 Collins랩과 임페리얼의 CSynBI에서 프로모터들의 강하고 약한 세기에 따른 라이브러리를 구축하였는데 (그림 2 A) [7] 이들에 의해서 나온 출력 커브는 모든 입력값에 대한 범위를 포함하는 완전한 데이터가 아니라는 단점을 가지고 있다 [8]. 실제로 부품의 특성을 파악하기 위한 조건으로 다음 네 가지를 들 수 있는데 1) 대규모 독립세포 측정기술 2) 부품의 전체 입력 수준에 대한 출력 신호 수준 측정 3) 시스템의 카피당 효과 계산 4) 행동의 변화를 추정하기 위한 각 입력 신호에 대한 출력 신호의 분포 가 그것이다. 이러한 조건을 만족하는 기술 개발의 연구로 TASBE (A Tool-Chain to Accelerate Synthetic Biological Engineering) 프로젝트를 들 수 있다 [9]. TASBE는 회로의 설계를 세포 내에서 구현될 수 있는 고수준의 언어로 바꾸어주어 실험실의 로봇 및 FACS (Fluorescence activated cell sorting) 장비와 연계하여 빠르게 부품의 특성 파악해주는 방법이라고 할 수 있다.
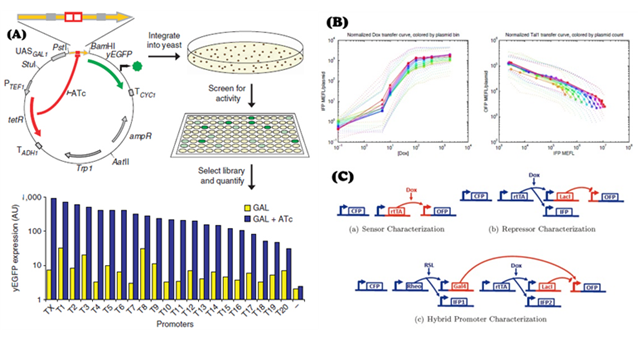
그림 2. (A) 프로코터 특성 파악 라이브러리 제작 [7]
(B) 각종 부품의 입력 값에 따른 출력 값 정량적 특성 파악 [9]
(C) 각종 부품의 특성파악을 위한 테스터 회뢰 [9]
TASBE는 기존의 방법들과 비슷하지만 다른 방법들과는 달리 고수준의 설계 기법으로 유기적으로 가깝게 묶여있지 않아서 해당 부품의 독립적 특성을 예측하기에 충분한 데이터를 모을 수 있다. 이 방법은 또한 단일세포 형광 측정 분류기인 FACS를 기반으로 하고 있어서 세포 내 부품의 변동을 정밀하게 측정할 수 있다. 그림 2 C의 회로들은 다음 세가지 구성물을 포함하고 있어서 정확한 수준의 플라스미드당 형광 데이터를 만들어내는데 1) 세포내 시스템의 카피수를 측정할 수 있는 필수 구성 형광 단백질, 2) 형광 단백질/리드 체널 스크리닝하는 방법, 3) 잡음 대비 신호를 증가시키는 다차원 데이터 조각 등이 그 구성물이다. 이러한 방법으로 각 부품들의 특성을 파악하고 그 데이터들을 수집해 놓으면 원하는 기능을 수행하는 시스템을 구축 할 때 좀 더 쉽고 정확하게 타깃 부품들을 고르고 적용시킬 수 있으며 시스템의 확장이나 부품 교체에 있어서도 용이한 장점을 가질 수 있다.
마지막으로 표준화된 바이오 부품의 빠른 생산으로 용이한 이용과 신속한 디자인 및 제품화를 가능하게 하는 바이오팹에 대한 연구 동향에 대해서 알아보자. 바이오팹 (International Open Facility Advancing Biotechnology) 프로젝트는 BioBrick의 Registry of Standard Biological Part를 통해 수집된 바이오 부품을 표준화하여 실제로 쉽게 사용할 수 있게 만들어서 부품들의 효율을 높이고 균일한 기능을 하도록 만드는 프로젝트이다 (http://biofab.org/). 즉, BIOFAB 프로젝트의 미션은 표준화된 바이오 부품의 광범위한 수집을 통해서 대학 및 산업계에서 용이하게 이용할 수 있는 맞춤형 유전적 구조물(genetic construct)의 신속한 설계/생산 및 시제품화(prototyping)를 가능하게 하는 것이다. 이 프로젝트는 다음 세대 생명공학을 뒷받침할 수 있는 오픈소스 기반의 최초의 집중적인 투자라는데 의의가 있고 현재는 C-Dogma 프로젝트라 불리는 대장균의 핵심 유전자 발현을 조절하는데 필요한 부품의 특성을 밝히고 표준화시키는 작업을 진행 중이다. C-Dogma프로젝트에서는 원핵세포에서 전사와 번역 초기화를 담당하는 부품들의 경계와 연결점을 정의하여 정확한 유전자 발현을 조절할 수 있는 유전자 발현 카세트 구조를 개발했다. 전사 부품은 공통적인 mRNA 시작위치의 인코딩을 담당하고 번역 초기화 부품은 많은 자연계 시스템에서 발견되는 유전 서열 모티프를 사용하였다. 이러한 번역 초기화 부품들과 함께 항시 발현되거나 또는 억제자에 의해서 조절되는 프로모터들의 라이브러리를 구축하여 이들 라이브러리의 활성 분포를 측정하였고 약 1000배 정도의 발현 범위를 보이는 부품들을 선정했다. 이렇게 만들어진 유전자 발현 키트는 93% 정도의 신뢰 수준으로 임의의 유전자 발현을 2배 이내의 예측 범위 내에서 발현시킬 수 있었다. 이러한 연구는 앞으로 일반적인 부품의 개량과 표준화에도 적용될 수 있으며 신뢰성 높은 지놈스케일의 유전자 발현 시스템을 구축하는데 기여할 수 있을 것이다. 또한 바이오팹에서는 SynBERC의 파트너로써 긴밀하게 SynBERC의 요청에 의한 시제품(prototyping)의 생산을 진행 중에 있다.
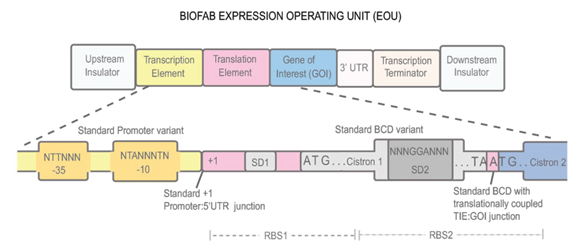
그림 3. 전사담당 부품 (노랑)부터 번역 (빨강), 삽입한 유전자 (파랑) 부품까지 정의된 서열 모티프들의
라이브러리를 구축하여 삽입된 유전자의 발현을 조절할 수 있는 바이오팹의 표준화된 발현 유닛
3. 정형화된 시스템 설계를 위한 연구
위에서 언급한 부품들의 표준화 연구가 진행될수록 합성생물학 연구자들은 좀 더 복잡하고 정교한 시스템을 설계할 수 있게 된다. 그러나 복잡한 시스템이 되어갈수록 원하는 기능과 활용 가능한 분자 물질 그리고 반응 경로 등의 설계 과정이 구조적으로 정립될 필요가 있는데 정형화된 설계 과정을 통한 다른 공학분야에서의 성공적인 사례들과는 달리 생물학적 시스템은 아직 알려지지 않은 불확실성과 확률적이고 비선형적인 상호작용 등을 고려해야 하는 어려움이 있다. 그림 4는 시스템이 복잡해질수록 우리가 원하는 표현형의 출력에 노이즈가 확률적으로 증가하고 있음을 보여준다 [10].
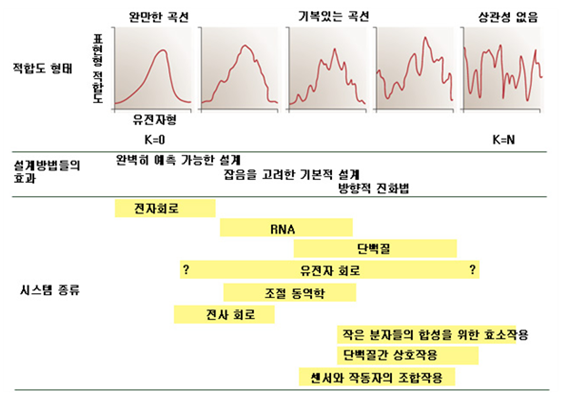
그림 4. 표현형의 적합도 형태로 보는 회로 설계법과 특징. 합성 유전자 회로가 예측한대로 행동하지 않는 이유 중 하나는 이 회로가 독립적이 아닌 살아있는 세포 내 구성원의 일부로 작동하기 때문이다. NK 모형에 의하면 (N=노드의 수, K=한 노드당 연결된 평균 노드의 수) 만약 한 노드의 효과가 시스템에 단독적으로 영향을 준다면 적합도는 완만한 곡선형이 될 것이다 (K=0). 반대로 그 효과가 시스템의 다른 요소에도 제한 없이 영향을 준다면 해당 시스템의 적합도는 최대한의 비선형성을 갖는다 (K=N). 실제 생물학적 시스템은 중간 정도의 적합도 형태를 가지고 있는데 완만한 곡선의 형태를 가질수록 더 효과적인 설계를 수행 할 수 있으므로 합성생물학 연구의 목표는 이러한 적합도 형태가 완만한 곡선(직교화)이 되도록 부품과 모듈을 설계하는 것에 있다.[10]
현재 합성생물학은 상향식과 하향식 방법을 조합할 수 있는 수준에 이르렀는데 이를 위해서 사용 가능한 잘 정의된 부품과 모듈을 확보하고 최소한의 전사 조절 요소들이 그들의 조건에 만족하도록 하는 것이 필요하다. 컴퓨터를 이용해서 최적의 반응 경로를 선택하는 것도 중요한데 합성생물학 분야에서도 이미 많은 프로그램이 소개되었고 여전히 활발히 개발 중에 있어서 여러 생물학적 부품들의 동역학이나 한계를 고려하여 회로를 설계하는데 도움을 주고 있다 [11]. 비록 이렇게 고수준으로 정형화시킨 도구들을 합성생물학의 모든 설계에 적용시킬 수는 없지만 적어도 센서와 작동자로 이루어진 전사 조절 회로에 대해서는 이미 적용이 잘 되어있는 상태이다.
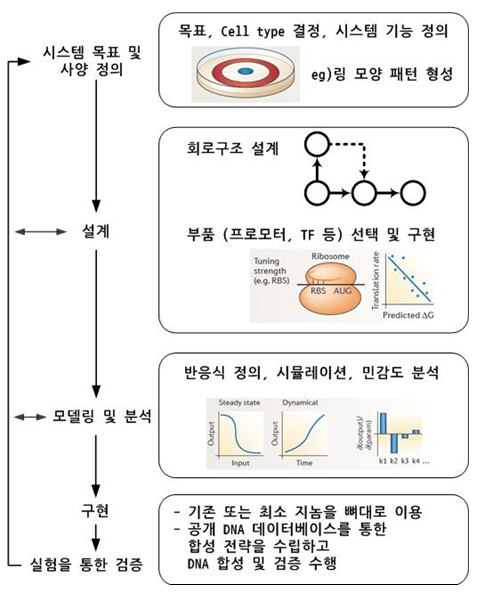
그림 5. 이상적인 설계법의 다섯 단계로 a) 사양 정의, b) 회로와 부품 설계, c) 모델링 및 분석,d) 구현,
e) 실험적 검증이 있고 원하는 시스템을 얻을 때까지 이 작업들을 반복 수행할 수 있다 [10].
정형화된 설계에서 중요한 요소 중 하나는 두 세 단계의 추상화 과정을 갖는 복잡한 표현형의 시스템을 하향적 분해와 상향적 조립 방법의 조합으로 설계하는 것이다. 예를 들어 Tabor와 그 동료들에 의해 제안된 경계검출 회로는 하향식 분해법에 의한 설계가 어떻게 기능적 복잡성을 유지하면서 쉽게 구현할 수 있는지를 보여준다 [12]. 또한 다세포 환경에서 복잡한 시스템을 개발하는 예로 Tamsir 팀은 NOR 게이트만을 이용해서 두 개의 입력값을 갖는 16개의 가능한 논리 기능을 구현했다. 그들은 복잡한 표현형을 간단한 NOR 논리 회로들의 조합으로 분해하고 미리 정의된 부품들을 상향식으로 조립하였다. 그 NOR 논리 회로를 분리된 박테리아 그룹들에 적용시키고 쿼럼센싱에의한 세포간 통신으로 논리 회로를 완성하였다 [13]. 이 외에도 모듈들을 시스템으로 조합했을 때 정의된 기능 외에 다른 상호작용을 없애는 직교적 모듈간의 연결 법이 있고 모듈의 배치를 다르게 했을 때 기대하지 않는 의존성이 발생하는 것을 (역작용을) 막는 연구도 진행되었다 [14]. 결과적으로 [10] 에서는 이상적인 설계법을 그림 5와 같은 다섯 단계로 구분하여 제안하고 있다.
4. 고찰
합성생물학 연구가 발전됨에 따라서 부품의 표준화와 정형화된 설계 기법의 도입은 정밀하고 예측 가능한 시스템을 구현하는데 필수적이라고 할 수 있다. 특히 기반 기술이 되는 부품의 표준화는 적절한 특성화 관측 기법에 다양한 부품을 적용함으로써 정량화된 데이터를 수집하는 것이 그 핵심 기반이 된다. 이렇게 잘 정의되고 정량화된 부품을 정형화된 설계 기법과 소프트웨어를 사용하여 활용할 수 있다면 합성생물학을 기반으로 한 실용적인 기술 개발과 상업화에 있어서 중요한 기여를 할 수 있을 것이다.
5. 참고서적
1. Gardner, T. S., Cantor, C. R. & Collins, J. J., Construction of a genetic toggle switch in Escherichia coli. Nature 403,
339-342 (2000)
2. Purnick, P., Weiss R., The second wave of synthetic biology: from modules to systems, Nature reviews. Molecular
cell biology 10, 410-432 (2009)
3. Arkin A., Setting the standard in synthetic biology, Nature biotechnology 26, 771-775 (2008)
4. T. Ellis, T. Adie and G. S. Baldwin, DNA assembly for synthetic biology: from parts to pathways and beyond, Integr.
Biol. 3, 109-118 (2011)
5. http://cordis.europa.eu/search/index.cfm?
fuseaction=proj.document&PJ_LANG=EN&PJ_RCN=12428453&pid=0&q=5A9E14B8E45BEB7F73FB2996A3BE10F8&type=sim
6. C. Smolke, Building outside of the box: iGEM and the BioBricks Foundation, Nature biotechnology 27, 1099-1201 (2009)
7. T. Ellis, X. Wang, and J. Collins. Diversity-based, model-guided construction of synthetic gene networks with
predicted functions. Nat. Biotech, 27 465–471, (2009)
8. Centre for Synthetic Biology and Innovation (CSynBI). Part characterisation.
http://www3.imperial.ac.uk/syntheticbiology/parts, (2012)
9. J. Beal, R. Weiss, F. Yaman et al., Computer Science and Artificial Intelligence Laboratory Technical Report A Method
for Fast , High-Precision Characterization of Synthetic Biology Devices A Method for Fast , High-Precision
Characterization of Synthetic Biology Devices, Technical Report, MIT (2012)
10. Slusarczyk, A.L. and Lin, A. and Weiss, R., Foundations for the design and implementation of synthetic genetic
circuits. Nature Reviews Genetics. 13, 406-420 (2012)
11. Chandran, D., Bergmann, F. T., Sauro, H. M. & Densmore, D. Design and Analysis of Biomolecular, Circuits:
Engineering Approaches to Systems and Synthetic Biology 203–224 (Springer, 2011)
12. Tabor, J. J. et al. A synthetic genetic edge detection program. Cell 137, 1272–1281 (2009)
13. Tamsir, A. Tabor, J. J. & Voigt, C. A. Robust multicellular computing using genetically encoded NOR gates and
chemical “wires”. Nature 469, 212–215 (2011)
14. Del Vecchio, D., Ninfa, A. J. & Sontag, E. D. Modular cell biology: retroactivity and insulation. Mol. Systems Biol. 4,
161 (2008)
※ 파일 다운로드는 로그인 후 이용하여 주시기 바랍니다.





