게놈 스케일 DNA 합성 기술 동향 조사
방두희, 연세대학교 화학과, duheebang@yonsei.ac.kr
유전자 또는 게놈 합성 기술은 데이터베이스에 저장되어 있는 유전 정보를 가공하여, 유전자나 더 나아가 게놈 크기에 해당하는 DNA를 화학적으로 합성하는 기술이다. 1Kb 이상의 유전자 수준의 DNA를 신속하고 정확하게 그리고 저비용으로 합성한다는 점에서 주로 20~100bp 길이의 DNA를 주로 합성하던, 기존의 올리고 합성 기술과는 구분된다.
유전자 합성 기술을 통해 만들어진 합성 유전자가 직접적으로 활용될 수 있는 분야를 요약하면 다음과 같다. 합성 유전자 및 게놈을 형질 변환에 이용하거나, 인공적으로 만들어진 세포를 통해 셀룰로스 공급 원료를 (cellulosic feedstock) 효율적으로 바이오 연료로 변환하는 기술 개발, 바이러스 게놈의 합성을 통한 백신의 개발(2008, Wimmer), 유전자 클러스터의 디자인 및 합성을 통한 새로운 약 분자 생산 기술 개발 (2005, Santi), 유전자 디자인 알고리즘과 유전자 합성 기술을 이용한 최적화된 항체(antibody) 합성 등으로 매우 다양하다. [그림 1]
[그림 ] 유전자 합성 기술의 적용 분야
기존의 유전자 합성법은 주로 고체상 합성법으로 합성된 조각 올리고를 이용, 그것들을 어셈블 하는 과정을 거쳐 원하는 유전자를 합성한다. 조각 올리고들을 어셈블 하는 방법으로는, 증폭물의(amplicon) 단계적 유도를 이용한 Assembly PCR(1995, Stemmer), 단일가닥 올리고가 서로 빈 공간 없이 겹쳐지게 디자인 한 후 thermostable ligase를 이용해서 어셈블 시켜주는 LCR(Ligase chain reaction), exonuclease와 thermostable ligase, polymerase를 이용해서 이중가닥 올리고를 연결하는 Gibson assembly의 크게 세 가지로 나눌 수 있다.(2009, Gibson)[그림 2]
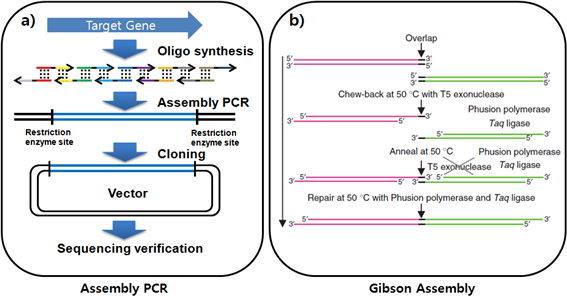
[그림 ] Assembly PCR 과 Gibson Assembly의 모식도
이러한 유전자 합성기술의 발달에 기반하여, 현재 국내외 여러 회사에서 유전자 합성 서비스를 제공할 수 있을 정도로 유전자 합성기술이 보편화되었고, 이는 게놈 스케일 DNA합성의 발판이 되었다.
게놈 스케일 DNA 합성은 2008년 Craig Venter 그룹에서 580kbp에 해당하는 Mycoplasma genitalium 게놈을 합성하면서 시작되었다. 이들은 합성하고자 하는 게놈 서열에 water mark를 위치시켰고 5~7 kbp의 101개의 DNA cassette로 나누어서 합성하였다. 이렇게 합성된 DNA조각들은 대장균과 Yeast 숙주세포의 재조합 시스템을 이용하여 순차적으로 assemble해 나가는 방식을 이용, 최종적으로 580kbp의 인공 게놈으로 어셈블 되었다. (2008, Gibson) [그림 3-(a)]
그 후 Mycoplasma genitalium 게놈 합성 성공을 토대로, 이 연구진은 2010년에 1080bp 크기의 DNA조각 1078개를 합성한 뒤 순차적으로 어셈블하여 1.08M(mega-)bp 크기의 Mycoplasma mycoides 게놈을 인공적으로 합성하는데 성공을 거두었다. 나아가 이렇게 합성된 인공 게놈이 다른 종인 Mycoplasma capricolum recipient 세포에 transplant 시킴으로서 인공 게놈의 작동여부를 확인하고자 하였다. 합성된 인공 게놈은 정상적으로 작동 시 푸른색의 콜로니를 형성하도록 디자인 되었는데, transplant된 세포 중 푸른색을 띄는 것을 확인함으로서 인공 게놈이 정상적으로 작동하는 것을 증명하였다.(2010, Gibson) [그림3-(b)]
또한 2011년 Boeke 그룹에서는 진핵세포인 Yeast의 게놈 중 일부인 90 Kbp크기의 DNA를 합성하는데 성공하였다. 이들은 조각 올리고를 이용해 게놈 스케일로 합성한 것이 아니라 기존의 효모 게놈을 추출 한 후 미리 디자인 해둔 게놈 서열을 세포 외부에서 PCR을 이용해 조직적으로 배열하여 게놈의 일부를 서열을 완성하였다. 또한 합성된 게놈은 훗날 genetic code를 확장하는데 사용하기 위해서 TAG코돈을 TAA코돈으로 치환하는 등 여러 인공적인 요소의 디자인이 가미되었다. 이렇게 합성된 효모의 게놈 일부분을 다시 효모에 이식하여 다양한 유전형과 그에 수반되는 다양한 표현형을 확인하였다.(2011, Dymond) [그림 3-(c)]
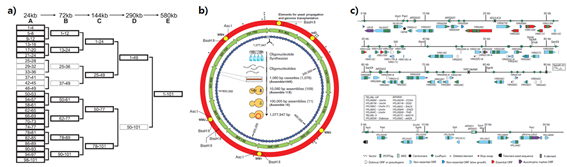
[그림 ] 580kbp M.genitalium 게놈, 1.08Mbp M.mycoides 게놈, Yeast chromosome 합성 모식도
이렇듯 최근 미국의 연구진들에 의해 수 만 bp ~ 메가 bp크기의 DNA를 합성하는데 성공하였지만, 아직 극복해야할 한계가 여럿 존재한다. 기존의 보편화된 합성생물학의 방법은 고체상의 올리고를 이용하여 어셈블하는 기술이기에 이와 함께 동반되는 높은 올리고의 가격, 합성 과정의 노동적, 시간적 한계 그리고 오류 유무를 확인하기 위해서 반드시 거쳐야 하는 염기서열 분석 등이 합성생물학이 극복해야 하는 큰 숙제로 꼽히고 있다.
현재 유전자 및 게놈 스케일 합성의 가장 큰 장애물은 합성에 사용되는 조각 올리고의 높은 가격이다. 최근 조각 올리고의 합성 기술의 발달로 1nt당 합성비용이 $0.10~0.20로 많이 낮아졌지만, 이 가격으로 1 Kbp크기의 유전자 하나를 합성하는데 올리고 비용만 대략 $200~$400가 필요하게 된다. 실제로 Venter 그룹이나 Boeke 그룹에서 인조 게놈을 합성하기 위해 올리고 비용만으로 수 억 원에서 수 십 억 원이 사용되었다.
두 번째 장애물로는 높은 시퀀싱 비용을 꼽을 수 있다. 합성하는 유전자 또는 게놈은 1 bp의 오류만으로도 우리가 확인하고자 하는 성능을 발휘하지 못할 수 있기 때문에 오류가 없는 형태로 합성 되어야 한다. 실제로 벤터 그룹에서 합성한 미생물 게놈은 1 bp의 합성 오류가 전체 미생물 시스템의 re-booting을 막는 결과를 초래하였다. 따라서 오류 없는 게놈 합성을 위해서는, 합성된 유전자 조각들을 vector에 클로닝한 뒤 염기서열을 확인하는 시퀀싱 과정을 거친다. 전통적인 클로닝 과정은 매우 노동집약적이고 많은 시간을 소요하고 오류 없는 합성 유전자를 찾기 까지 많은 양의 시퀀싱 비용을 지출하게 된다.
앞서 언급된 문제들을 해결하기 위한 연구들이 최근 수행 되었는데 그 중 한 가지가 마이크로칩 올리고를 이용한 유전자 합성법이다. 마이크로칩 올리고의 특징은 저렴한 가격에 수천~수만 가지의 올리고를 한 번에 합성할 수 있다는 것이다. Church 그룹에서는 2011년에 마이크로 칩 올리고를 이용해서 42개의 합성하기 어려운 치료용 항체 서열을 포함하는 47개의 유전자를 합성하는데 성공하였다. (2011, Kosuri) [그림 4-(a)]
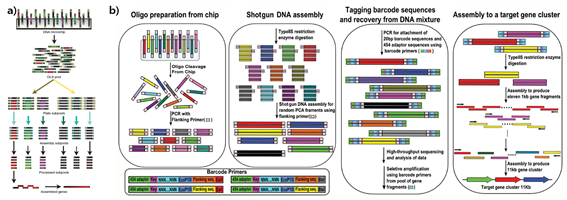
[그림 ] 마이크로 칩 올리고 및 차세대 시퀀싱을 이용한 유전자합성 모식도
올리고 가격을 낮추려는 노력 외에 시퀀싱 비용을 낮추려는 노력도 시도 되었다. 2012년 방두희 교수 연구진에서는 Shotgun DNA synthesis 논문을 통해 마이크로칩 DNA로 어셈블된 DNA중 효율적으로 오류 없는 DNA조각들만을 회수할 수 있는 방법을 개발하였다. 이 기술은 유전자 합성에 마이크로 칩 올리고를 이용하고, 염기서열 분석은 차세대 시퀀싱 기술을 이용함으로서 유전자 합성비용의 절감을 시도한 첫 번째 논문이다. 어셈블이 진행된 DNA분자마다 서로 다른 바코드 서열을 달아준 뒤 차세대 시퀀싱 기술을 이용해서 한 번의 반응으로 합성된 DNA 분자 하나하나의 오류 여부와 오류 없는 DNA조각이 갖는 바코드 서열을 알아내었다. 이렇게 알아낸 정보를 바탕으로 오류 없는 DNA조각들만을 선별하여 증폭 및 회수한 뒤 더 큰 DNA를 합성하는데 이용함으로써 기존의 노동 집약적이고 많은 비용이 소모되던 클로닝 및 시퀀싱 분석과정을 대체할 수 있는 대안을 제시 하였다. (2012, Kim) [그림 4-(b)]
마지막으로 합성된 DNA조각이 갖는 오류율 자체를 낮추려는 노력도 진행되었다. 합성이 완료된 뒤, 만들어진 유전자 조각에 대하여 열을 가해주고 다시 결합시켜 주는 (heat denatured and reannealed) 방법을 이용하여 mismatch를 형성시킨 뒤 수정하는 방법이 제시 되었다. Assembly PCR을 이용하여 만든 유전자 결과물에 대한 오류는 불규칙적으로 분포하기 때문에 열을 가해주고 다시 결합시켜 줌으로써 최종적으로 어닐링 된 DNA 중에는 mismatch를 포함하는 부분을 가지는 DNA가 생성된다. 이러한 mismatch 오류에 대해 T7 endonuclease라는 효소를 적용하면 dsDNA 상에 오류를 갖는 염기서열이 존재할 때, 이를 인식하고 분해하게 된다.(2008 Bang; 2012, Ma) 또한 endonuclease를 이용하는 방법과 비슷한 방법으로, 오류로 인해서 어닐링 시 발생하는 mismatch 오류에 대해, mismatch를 인식하는 MutS 단백질을 붙임으로써 오류를 찾는 방법이 있다. 그리고 mismatch가 존재하는 DNA에 단백질이 붙으면, 젤 전기연동을 이용하여 분리해 낼 수 있다. 이러한 방법으로 오류가 없는 유전자를 얻을 수 있다.(2004, Carr) [그림 5] 실제로 앞서 언급한 Church 그룹의 2011년 유전자 합성 연구에서는 이미 합성된 유전자에 추가적인 오류 수정작업을 진행했을 때 합성된 유전자의 오류율이 현저하게 낮아지는 것을 확인하였다.
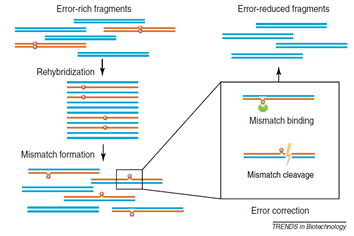
[그림 5] 오류 수정 과정 모식도
이렇듯 합성생물학 분야에서 유전자/유전체 합성의 계속되는 연구 ․ 발전으로 인해 유전체 크기의 DNA 합성이 바이오 에너지 산업, 의약품 개발 산업 등에 활용되면서 그 중요성이 나날이 증대되고 있다. 또한, DNA 합성 기술은 향후 생물학, 화학, 나노 과학, 대체 에너지와 의학 산업에 커다란 파급 효과를 줄 것으로 기대된다. 현재 많은 합성생물학 연구진들은 유전체 크기의 DNA를 합성하기 위해 들어가는 천문학적 비용과 많은 시간 그리고 인력이 소요되는 것을 극복하기 위해 힘쓰고 있으며 이와 같은 연구는 기존의 유전체 합성 방식과는 근본적으로 다른 접근법의 유전체 합성 원천 기술이 될 것으로 생각된다.
메가 유전체 수준으로 합성할 수 있는 기술 개발은 합성에 대한 숙주 세포 의존성을 낮추고, 합성 시간을 기존의 최소 1년에서, 한 달, 또는 심지어는 수 일 이내로도 줄일 수 있을 것으로 기대된다. 더불어 메가 유전체 기반 바이오 시스템뿐만 아니라, 화학 의약품, 2차 대사물 관련 유전자 클러스터 합성에 적용하여, 수 백 개의 유전자 클러스터 합성과, 수 천 개의 항체 단백질 유전자 합성에 들이는 비용을 기존의 수 십 억 원 수준에서, 수 백, 수 십 만원 수준으로 낮출 수 있다고 예상된다
※ 파일 다운로드는 로그인 후 이용하여 주시기 바랍니다.





